Introduction
Le méthane (CH4) est un gaz à effet de serre majeur. Son émission à partir de milieux naturels tels que les zones humides, les océans et les sédiments représente plus de 70% du méthane atmosphérique au niveau mondial (GIEC, 2007). Une évaluation des données publiées a révélé des taux d’émission de méthane différents pour les zones humides, les lacs, les rivières, les estuaires et les océans (par ordre décroissant, figure supplémentaire S1). Les zones humides naturelles représentent à elles seules 62 % de la production de CH4 biogénique (Kirschke et al., 2013 ; Nazaries et al., 2013) et les émissions des zones humides dominent la variabilité interannuelle des sources de méthane (Bousquet et al., 2006). En revanche, la vaste superficie des écosystèmes marins ne contribue que pour environ 8 % aux sources naturelles de CH4 (Nazaries et al., 2013). Au sein de l’émission globale de méthane océanique, moins de 10% est contribué par les estuaires (Bange et al., 1994).
La méthanogénèse, la formation biologique du méthane, est réalisée par des archées méthanogènes qui produisent du méthane principalement à partir de H2/CO2, de groupes méthyles ou d’acétate dans des conditions anoxiques (Thauer et al., 2008). Cette réaction est catalysée par la méthyl-coenzyme M réductase (MCR). Le gène mcrA, qui code pour une sous-unité de cette enzyme, est un marqueur génétique couramment utilisé dans les études moléculaires (Conrad, 2007 ; Bridgham et al., 2013). L’avantage du marqueur génétique mcrA est de capturer à la fois les signatures phylogénétiques et fonctionnelles des méthanogènes, offrant une grande profondeur de séquençage pour cette fonction particulière (Luton et al., 2002 ; Borrel et al., 2013 ; Yang et al., 2014). Un grand nombre de séquences mcrA ont été récupérées dans une variété d’environnements naturels. L’ensemble public de données mcrA permet d’extraire des modèles écologiques généraux et d’étudier les gradients environnementaux à l’échelle mondiale et régionale. En outre, une base de données résumant les propriétés physiologiques de 152 isolats méthanogènes est disponible1 (Jabłoński et al., 2015). Récemment, le binning du génome a révélé des séquences mcrA inhabituelles dans la nouvelle classe de Methanofastidiosa (Nobu et al., 2016) et les nouveaux phyla de Bathyarchaeota (Evans et al., 2015) et Verstraetearchaeota (Vanwonterghem et al., 2016). Ces nouveaux résultats ont élargi nos connaissances sur la diversité des méthanogènes potentiels mais n’ont pas occulté l’applicabilité du gène mcrA comme marqueur moléculaire pour la grande majorité des communautés méthanogènes.
À ce jour, des communautés méthanogènes ont été détectées dans des zones humides, des sédiments, des zones de pergélisol, des rizières, des digesteurs, des sources géothermiques et des évents hydrothermaux (Conrad, 2007 ; Thauer et al., 2008 ; Wagner et Liebner, 2009). La structure de la communauté méthanogène s’est avérée être associée au pH, à la température, à la salinité, au niveau des eaux souterraines et à la dynamique de la végétation à différentes échelles spatiales et temporelles (Megonigal et al., 2005 ; Milferstedt et al., 2010 ; Frank-Fahle et al., 2014 ; McCalley et al., 2014 ; Cui et al., 2015 ; Liebner et al., 2015). Par exemple, la méthanogénèse acétoclastique est généralement entravée par un pH faible car il réduit la dissociation de l’acétate (Megonigal et al., 2005 ; Kotsyurbenko et al., 2007). La végétation peut fournir du carbone organique labile de haute qualité pour alimenter les méthanogènes sous forme d’exsudats de racines ou de détritus, de sorte que les exsudats de plantes favorisent généralement les méthanogènes acétoclastiques principalement dans les fens (Bridgham et al., 2013). Le sulfate de l’eau de mer inhibe la production de méthane dans les zones humides à marée, et la salinité a par conséquent été utilisée comme un prédicteur général des émissions de méthane (Holm et al., 2016). Une étude sur les sédiments de lacs tibétains a montré que l’augmentation de la salinité inhibe les méthanogènes hydrogénotrophes mais renforce la méthanogénèse acétoclastique (Liu et al., 2016). Ces études ont indiqué des facteurs environnementaux pour les communautés méthanogènes, mais se sont concentrées sur des habitats uniques ou des échelles spatiales limitées.
Comprendre l’adaptation des méthanogènes à différents changements environnementaux nécessite cependant une exploration systématique et globale des corrélations entre la composition des communautés microbiennes et les conditions environnementales (Lozupone et Knight, 2007). Actuellement, seules quelques études traitent de la dispersion et du filtrage de l’habitat des communautés méthanogènes (Auguet et al., 2010 ; Barreto et al., 2014). Nous émettons l’hypothèse que les assemblages méthanogènes sont principalement influencés par le filtrage de l’habitat et qu’il est piloté par les contrôles environnementaux globaux. Considérant que les taux d’émission de méthane diffèrent largement entre les écosystèmes naturels, l’intégration explicite de la composition, de la diversité et de la biogéographie des assemblages méthanogènes dans ces écosystèmes peut être fondamentale pour déterminer la réponse de la production de méthane aux changements climatiques actuels et futurs. Cette méta-étude est réalisée pour combler les lacunes associées à la biogéographie méthanogène, la diversité et ses contrôles environnementaux en utilisant les données de séquence mcrA disponibles publiquement et la littérature complétée par les données physiologiques des isolats méthanogènes.
Matériel et méthodes
Collecte de données
Nous avons récupéré les séquences mcrA disponibles dans GenBank (janvier 2016)2. Pour chaque résultat, l’article original a été vérifié et les séquences mcrA correspondantes ont été analysées par un script Perl personnalisé. Comme nous nous sommes concentrés sur les environnements naturels, les séquences mcrA méthanogènes ont été obtenues à partir d’habitats naturels et classées comme sol, sédiments lacustres, estuariens, marins et hydrothermaux, et volcans de boue. Cinq bibliothèques issues du séquençage de nouvelle génération (NGS) ont été incluses en plus des séquences des bibliothèques de clones. Les séquences ont été téléchargées sans tenir compte de l’abondance relative dans l’ensemble de données original. Étant donné que les séquences des bibliothèques de clones couvrent principalement les phylotypes abondants alors que le NGS peut capturer une diversité beaucoup plus profonde, nous avons fait un compromis afin d’utiliser les données NGS tout en atténuant une erreur potentielle due à la différence de résolution des méthodes de séquençage. Par conséquent, nous n’avons choisi que les séquences représentatives des OTUs abondantes avec une abondance relative supérieure à 1%. Nous avons également rejeté les séquences NGS qui ont échoué à la vérification de la traduction des nucléotides en séquences protéiques ou dont la qualité est faible (séquences < 250 bps). Enfin, nous avons construit un ensemble de données contenant 4466 séquences mcrA uniques provenant de 94 sites répartis dans le monde entier (Figure 1 et Tableaux supplémentaires S1, S2). En outre, nous n’avons pas soustrait de l’ensemble de données les séquences mcrA de méthanotrophes archéaux potentiels, qui ont été inévitablement détectés dans l’enquête génomique (Conrad, 2007). Cette partie dépasse le cadre de cette étude.
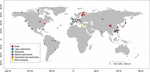
FIGURE 1. Localisation des sites utilisés pour cette étude. Les sites ont été regroupés en six catégories différentes en fonction de leurs habitats et ont été étiquetés avec des couleurs différentes. Un résumé des six habitats est donné dans les tableaux supplémentaires S1, S2.
Les coordonnées géographiques et les paramètres environnementaux, y compris le pH, la salinité, l’altitude, la température annuelle moyenne de l’air (MAAT) et les précipitations annuelles moyennes (MAP) ont été extraits pour chaque site de recherche considéré dans cette étude à partir des publications correspondantes étant donné que les données sont disponibles (voir le tableau supplémentaire S1). Pour tenir compte des paramètres environnementaux manquants dans plusieurs études, nous avons défini qualitativement certaines variables environnementales en fonction des descriptions des sites dans la littérature pertinente, puis nous avons converti ces données de catégorie en valeurs numériques semi-métriques, par exemple, nous avons défini les échantillons de sédiments marins, de sédiments hydrothermaux, de boue volcanique et de sédiments de lacs sodiques comme étant » salins « , les échantillons de sols et de sédiments de lacs d’eau douce comme étant » non salins « , et les échantillons de mangroves et d’estuaires comme étant des échantillons » mixtes « .
Traitement des séquences brutes
Le traitement des séquences a été mis en œuvre avec la plateforme logicielle Mothur (Schloss et al, 2009). Les séquences provenant de différentes bibliothèques ont été regroupées avant le traitement. Les séquences ayant une longueur inférieure à 350 pb ou plus de 8 bases ambiguës ont été écartées. Par la suite, ces séquences ont été alignées contre un sous-ensemble pré-aligné de séquences mcrA, qui ont été récupérées dans la base de données FunGene à http://fungene.cme.msu.edu/ (Fish et al., 2013). Les séquences chimériques ont été identifiées avec le logiciel Mothur en utilisant la méthode uchime (Edgar et al., 2011) avec le jeu de données lui-même comme référence. Ensuite, les séquences nucléotidiques valides du gène mcrA ont été utilisées pour calculer les distances par paires non corrigées entre les séquences d’ADN alignées et ensuite assignées à des unités taxonomiques opérationnelles (OTU) à un seuil de 84 % qui correspond à 97 % pour le gène de l’ARNr 16S (Yang et al., 2014). L’abondance de chaque OTU mcrA n’a été comptabilisée que comme présence ou absence. Nous avons augmenté la précision de la classification taxonomique des OTU en considérant les séquences de nucléotides et d’acides aminés. Au niveau de l’ADN, l’identité taxonomique a été attribuée par la plateforme Mothur selon une base de données de référence (Yang et al., 2014). Au niveau des protéines, les séquences protéiques alignées ont été utilisées pour construire un arbre dans ARB, puis l’affectation taxonomique a été basée sur la base de données correspondante. Si l’affectation d’une OTU était incohérente, nous avons procédé à un blast manuel de la séquence de nucléotides et de protéines dans NCBI et déterminé l’identité taxonomique finale en tenant compte de la couverture de la requête (>95%), de l’identité (>84%) et de la valeur e (<1E-5). Pour les séquences protéiques, le seuil de coupure au niveau du genre se référait au seuil de 83,5% (Hunger et al., 2011).
Analyse écologique et statistique
L’analyse statistique a été réalisée par différents packages R. Les ordinations de l’analyse des coordonnées principales (PCoA) ont été générées sur la base des matrices de distance Jaccard construites à l’aide du paquet vegan v2.2.0 (Oksanen et al., 2015). Une MANOVA (analyse multivariée de la variance) permutationnelle a été réalisée pour évaluer la source de variation de la matrice de Jaccard (McArdle et Anderson, 2001) dans la végane avec 104 permutations. Les mesures de distance de Jaccard sont basées sur la présence/absence de l’espèce, ce qui convient mieux à notre ensemble de données car la plupart des études n’ont fourni que les séquences représentatives alors que les informations sur les abondances sont manquantes. Les fréquences d’incidence taxonomique à travers les habitats ont été visualisées par des diagrammes à bulles avec le paquet ggplot2 (v1.0.0) (Wickham, 2009). Une analyse de regroupement hiérarchique des communautés de sols non salins et de sédiments lacustres a été réalisée par la fonction R » hclust » (R Core Team, 2014). Les clusters de communautés obtenus ont été décrits en fonction du régime de pH et de température des échantillons d’origine, car en utilisant la PCoA au préalable, nous avons identifié les deux paramètres pH et température pour influencer la composition des communautés méthanogènes dans les sols non salins et les sédiments lacustres. L’association de chaque lignée méthanogène avec chacun de ces clusters a été déterminée en utilisant une analyse des espèces indicatrices basée sur la corrélation (Dufrene et Legendre, 1997). Les espèces indicatrices sont définies ici comme étant celles qui sont à la fois abondantes dans un type d’habitat spécifique (spécificité) et qui se trouvent de manière prédominante dans ce type d’habitat (fidélité). Dans cette étude, les taxons indicateurs, similaires au concept d’espèce indicatrice, pour les sols non salins et les sédiments lacustres ont été choisis en fonction d’une valeur indicatrice (valeur IndVal) par le package R labdsv (Roberts, 2016) si la probabilité d’obtenir une valeur indicatrice aussi élevée que celle observée sur les itérations spécifiées est inférieure à 0,05. Les indices Chao2 ont été calculés pour chaque échantillon à l’aide du package vegan. Le test de la somme des rangs de Wilcoxon des indices Chao2 entre les habitats a été réalisé par la fonction R ‘wilcox.test’ (R Core Team, 2014). Pour consolider l’impact du filtrage de l’habitat sur les isolats d’archées méthanogènes, les caractéristiques physiologiques et biochimiques des cultures méthanogènes décrites ont été extraites de la3 ‘Methanogenic archaea database’ (Jabłoński et al., 2015). Parmi eux, les isolats présentant des informations sur les besoins optimaux en NaCl ont été filtrés, catégorisés et tracés en fonction de leur source d’isolement.
Pour examiner l’influence de la limitation de la dispersion sur la structure de la communauté méthanogène, une analyse de régression linéaire a été réalisée sur la base d’une matrice de distance géographique et d’une matrice de distance Jaccard de la communauté par la fonction R ‘lm’ (R Core Team, 2014). Nous avons effectué des tests de Mantel et de Mantel partiel pour évaluer les effets de la limitation de la dispersion selon les deux matrices, toujours à l’aide du paquet vegan de R (Oksanen et al., 2015). En outre, une analyse spatiale multivariée (ACP spatiale) a été appliquée à 16 échantillons européens de sols et de sédiments lacustres sur la base de l’indice I de Moran pour explorer la structure spatiale des méthanogènes par la fonction » multispati » du paquet R ade4 (Dray et Dufour, 2007). En outre, le clustering de Ward’s Minimum variance qui était basé sur la matrice de distance de Jaccard a été mis en œuvre sur ces 16 échantillons en utilisant la fonction R « hclust » (R Core Team, 2014) et nous avons ensuite projeté les résultats du clustering sur une carte géographique. Le fichier de forme européen pour la cartographie au niveau des États est disponible dans la base de données GSHHG (v2.3.6)4. La carte a été générée en utilisant QGIS v2.18.25.
Résultats
Biogéographie des archées méthanogènes dans les environnements naturels
Les séquences du gène mcrA de 94 environnements naturels répartis dans le monde entier ont été récupérées. L’emplacement et le type d’écosystème de chacun de ces 94 sites sont représentés sur la figure 1. Les fréquences d’incidence (présence/absence) des lignées méthanogènes ont été fusionnées en fonction du type d’écosystème et illustrées dans la figure 2. En bref, Methanoregula est le taxon le plus fréquemment observé dans les sols, avec Methanobacterium, Methanosaeta, Methanocella, Methanomassiliicoccus et Methanosarcina. Dans les sédiments d’estuaire, des séquences de Methanosaeta, Methanobacterium, Methanoregula et Methanoculleus ont été fréquemment détectées. De plus, dans les sédiments lacustres, Methanoregula et Methanosaeta sont principalement présents. Dans les sédiments marins, Methanoculleus et Methanosaeta sont les lignées les plus communes, suivies par Methanolinea.
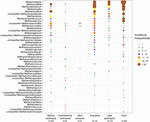
FIGURE 2. Graphique à bulles montrant les fréquences d’incidence des lignées méthanogènes dans différents environnements naturels. L’ordre de classement le long de l’axe vertical correspond aux fréquences d’incidence totales décroissantes des lignées. La taxonomie est indiquée pour le niveau du genre. Si une attribution au niveau du genre n’était pas possible, le niveau taxonomique supérieur suivant a été utilisé. Le nombre d’échantillons (n) est indiqué pour chaque habitat.
Même si de nombreux taxons ont été détectés dans différents environnements, certains montrent encore des préférences environnementales. Methanoregula, le taxon fréquemment présent dans les environnements non marins et de transition (sols, sédiments lacustres et estuaires), est absent des habitats marins (sédiments marins, sédiments hydrothermaux et volcans de boue). Methanobacterium et Methanocella, qui prédominent dans les environnements non marins et transitionnels, sont rarement trouvés dans les habitats marins. En revanche, Methanococcoides, en tant que lignée prédominante dans les sédiments marins, les sédiments hydrothermaux et les volcans de boue, est à peine observé dans les sols et les sédiments lacustres. En outre, Methanogenium et Methanolacinia ne sont observés que dans les sédiments estuariens et marins, tandis que Methanospirillum et Methanosphaerula ne sont présents que dans les environnements terrestres. En outre, certains taxons spécifiques sont exclusivement présents dans les sédiments hydrothermaux, notamment Methanocaldococcus, Methanothermococcus, Methanopyrus, Methanotorris et Methanococcus. Bien que certaines lignées telles que Methanosaeta soient présentes dans la plupart des environnements, aucune lignée ne peut être considérée comme omniprésente.
La plus grande richesse de lignées s’est produite dans les sédiments d’estuaire qui abritent également des fréquences d’incidence plus régulières de diverses lignées. En revanche, les volcans de boue et les écosystèmes hydrothermaux présentent une diversité méthanogène relativement faible. Les sols et les sédiments lacustres similaires aux estuaires sont caractérisés par des assemblages méthanogènes diversifiés.
Alpha-Diversité des communautés méthanogènes dans les environnements naturels
La richesse des archées méthanogènes selon l’indice Chao2 variait largement entre les types d’écosystèmes (figure 3). Dans le but de comparer directement les diversités alpha et d’obtenir un compromis raisonnable entre les échantillons, un sous-échantillonnage à 15 séquences pour chaque site a été effectué. L’indice Chao2 montre que les sédiments d’estuaire englobent la plus grande richesse en espèces de méthanogènes parmi les six types d’écosystèmes (tableau supplémentaire S3), ce qui souligne les résultats du graphique à bulles (figure 2). Les sols et les sédiments lacustres, montrant une richesse plus faible que les échantillons de l’estuaire, ont des indices de Chao2 significativement plus élevés que les sédiments marins et les sédiments hydrothermaux et sans différences significatives entre les sédiments marins et les sédiments hydrothermaux (tableau supplémentaire S3).
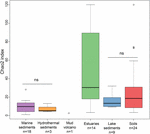
FIGURE 3. Box plot des indices de Chao2 des différents types d’écosystèmes. Le tracé est basé sur des ensembles de données sous-échantillonnées contenant 15 séquences pour chaque site afin de rendre la comparaison sur les mesures de diversité alpha plus robuste. Le nombre d’échantillons dans chaque habitat est indiqué par ‘n’ sous l’étiquette de l’habitat. Le ‘ns’ indique une absence de signification statistique dans le test de Wilcoxon. Le résultat statistique de la diversité alpha au niveau des OTU est donné dans le tableau supplémentaire S3.
Contrôles globaux des communautés méthanogènes dans les environnements naturels
Les 94 communautés méthanogènes distribuées globalement ont été regroupées dans un graphe d’ordination en appliquant la PCoA basée sur la matrice de distance de Jaccard. Selon l’analyse PCoA, le premier et le deuxième axe expliquent ensemble 16,3% de la variance totale. Les variations entre les échantillons peuvent donc être largement expliquées par la salinité (figure 4). Puisque les données initiales sur les concentrations de sel n’étaient pas disponibles dans certains cas, nous avons qualitativement assigné ces échantillons comme des échantillons salins, mixtes (intermédiaires) et non salins comme décrit ci-dessus. Les échantillons salins et non salins se séparent effectivement le long du premier axe. Les échantillons mixtes se situent globalement entre les échantillons salins et non salins. La MANOVA permutationnelle basée sur la matrice de distance de Jaccard suggère également que la salinité est le principal facteur abiotique contrôlant la distribution des communautés méthanogènes mondiales (R2 = 0,099, P < 0,001) (tableau 1).
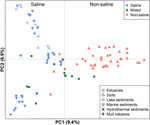
FIGURE 4. Ordination par analyse des coordonnées principales (PCoA) basée sur la matrice de distance de Jaccard des bibliothèques de séquences de gènes mcrA méthanogènes comparant 94 échantillons. La PCoA est colorée en fonction de la salinité : Les symboles rouges indiquent les environnements non salins, les bleus les environnements salins et les verts les environnements intermédiaires. Des symboles différents représentent des environnements différents. Le pourcentage de la variation expliquée par les coordonnées principales tracées est indiqué sur les axes.

TABLE 1. Analyse MANOVA permutationnelle sur la matrice de distance de Jaccard de tous les échantillons des six habitats pour tester l’association de la variance de la communauté avec différentes variables environnementales.
En outre, nous avons vérifié une relation potentielle entre la source d’isolement des cultures pures méthanogènes et la concentration optimale de NaCl pour la croissance. La concentration optimale de NaCl des cultures pures méthanogènes a démontré un déclin des écosystèmes marins aux estuaires, aux sédiments lacustres et aux sols (figure supplémentaire S2). Quelques isolats aberrants proviennent de sédiments de lacs sodiques ou de sols hypersalins.
Contrôles environnementaux et taxons indicateurs méthanogènes dans les sols non salins et les sédiments lacustres
À l’échelle mondiale, les communautés méthanogènes des sols non salins et des sédiments lacustres se regroupent étroitement (Figure 4), de sorte que nous avons analysé plus en détail les contrôles environnementaux des communautés méthanogènes de ces deux habitats qui représentent 33 sites d’étude au total. L’analyse de cluster basée sur la communauté pour ces deux types d’habitats a révélé quatre clusters sur la base de la matrice de distance de Jaccard (figure supplémentaire S3). La MANOVA permutationnelle suggère que le pH (R2 = 0,099, P < 0,001) et la température (R2 = 0,069, P < 0,001) influencent la β-diversité méthanogène dans les sols non salins et les sédiments lacustres (Tableau 2). En conséquence, nous avons attribué les quatre clusters au pH et à la MAAT du site d’échantillonnage initial et avons obtenu des sous-groupes largement cohérents au regroupement des communautés (Figures 5C,D). La combinaison des caractéristiques environnementales et de ces quatre clusters de communautés nous permet de définir ces quatre sous-groupes comme le groupe 1 neutre et froid, le groupe 2 acide et froid, le groupe 3 acide et modéré, et le groupe 4 neutre et chaud (Figure 5). Une ordination PCoA plus poussée basée sur la matrice de dissimilarité de Jaccard suggère que le long de l’axe PC1, la plupart des échantillons des groupes 2 et 3 proviennent de sols acides et de sédiments lacustres tandis que les groupes 1 et 4 proviennent principalement d’environnements neutres (Figure 5A). De plus, les échantillons des sites modérés (groupe 3) se sont séparés de ceux des sites chauds et froids le long de l’axe PC2, tandis que les échantillons des environnements chauds (groupe 4) se sont séparés des autres échantillons le long de l’axe PC3 (Figure 5B). Ainsi, les trois premiers axes de l’ordination PCoA expliquent 38,8% de la variation totale.

TABLE 2. MANOVA permutationnelle basée sur une matrice de distance de Jaccard des échantillons de sol non salin et de sédiments lacustres pour tester l’association de la variance de la communauté avec différentes variables environnementales.
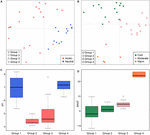
FIGURE 5. Tracé d’analyse des coordonnées principales basé sur une matrice de distance de Jaccard pour 33 sols non salins et sédiments lacustres. Le sous-plot (A) montre PC1 et PC2 et les symboles sont colorés par le pH, et le sous-plot (B) montre PC2 et PC3 et les symboles sont colorés par la température. Les trois premières composantes expliquent 17,1, 11,7 et 10 % de la variance. Les diagrammes en boîte montrent le pH (C) et la MAAT (température annuelle moyenne de l’air) (D) de quatre sous-groupes identifiés. La couleur de la boîte dans la figure C correspond à la catégorie de pH dans (A). Les couleurs des boxplots indiquent la signification statistique basée sur un test de Wilcoxon par paire (P < 0,05), où les échantillons avec la même couleur ne diffèrent pas significativement les uns des autres. De même, la couleur dans (D) suit le groupement de température dans (B). Les sous-groupes se réfèrent à l’analyse de cluster hiérarchique des similarités de la communauté.
Nous avons examiné l’occurrence des lignées méthanogènes dans chaque sous-groupe sur la base des fréquences d’incidence (Figure 6). Methanoregula prévaut dans tous les types d’habitats non salins. En plus de Methanoregula, le sous-groupe neutre et froid (groupe 1) montre une abondance élevée de Methanosaeta, Methanobacterium, et Methanosarcina. Le groupe 2 acide et froid est représenté par Methanobacterium, Methanocella, et Methanosarcina tandis que Methanosaeta est absent ici. Methanocella et Methanosaeta sont communs dans le groupe 3, acide et modéré. Dans le groupe 4, neutre et chaud, Methanolinea et Methanosaeta sont des membres importants. Ce groupe est le seul où Methanoculleus a été identifié.

FIGURE 6. Fréquences d’incidence des lignées méthanogènes dans les quatre groupes définis de communautés méthanogènes dans les sols et les sédiments lacustres. L’axe vertical est disposé par ordre alphabétique. La longueur des barres correspond aux fréquences d’incidence moyennes pour chaque lignée au sein du groupe correspondant. Les barres d’erreur représentent l’écart type d’un taxon donné sur différents échantillons de ce groupe. Les astérisques indiquent les taxons spécialisés avec une valeur P < 0,05. La taxonomie est indiquée pour le niveau du genre. Si une attribution au niveau du genre n’était pas possible, le niveau taxonomique supérieur suivant a été utilisé. La description des quatre groupes est donnée à la figure 5.
Pour les quatre groupes, les taxons ayant une incidence élevée sont Methanoregula, Methanobacterium, Methanosarcina, Methanosaeta, Methanomassiliicoccus et Methanocella. Les taxons spécialisés, qui sont significativement plus représentés dans la plupart des sites d’un groupe donné, ont été détectés selon l’analyse des espèces indicatrices comme décrit précédemment. Au total, six des 31 taxons présentaient une valeur indicatrice significative (P < 0,05) (marqués d’un astérisque dans la figure 6). Le groupe 1 (neutre et froid) a montré le plus grand nombre de spécialistes avec des lignées de Methanosaeta, Methanolobus, et Methanomethylovorans. Methanobacterium a servi de taxon spécialiste dans le groupe 2 (froid et acide) tandis que Methanolinea a été identifié comme spécialiste dans le groupe 4 (neutre et chaud) mais a été à peine observé dans les autres groupes. En outre, Methanoregula est largement représenté dans le groupe 3 acide et modéré.
Limitation de la dispersion
Une analyse de régression linéaire (R2= 0,05, P < 0,001) a indiqué une faible corrélation entre la distance géographique et la structure de la communauté méthanogène sur l’ensemble des données mondiales. En même temps, un test de Mantel a montré que les variables environnementales ont une corrélation plus élevée avec la structure de la communauté que les distances géographiques (voir tableau 3). Cette tendance est également confirmée par un test de Mantel partiel contrôlant les effets d’autocorrélation. Le tracé de la distance géographique par rapport à la similarité de la communauté de Jaccard ne montre pas de tendance linéaire claire mais des modèles qui résultent principalement de la distribution globale des points d’échantillonnage (voir la figure supplémentaire S4).

TABLE 3. Analyses des tests de Mantel et des tests de Mantel partiels pour la détermination de l’influence des variables environnementales et de la distance géographique sur la distribution microbienne pour l’ensemble des données mondiales et un sous-échantillon de 16 échantillons européens.
Afin d’analyser davantage l’influence de la dispersion, nous avons limité notre analyse à l’Europe, qui a été échantillonnée de la manière la plus dense et la plus régulière. Les tests de Mantel et les tests de Mantel partiels sur ce sous-ensemble ont reproduit la tendance selon laquelle les données de la communauté sont plus fortement corrélées aux variables environnementales qu’aux distances géographiques (voir tableau 3). Le test de Mantel partiel contrôlant les variables environnementales n’a pu détecter aucune corrélation statistiquement significative entre la communauté microbienne et la distance géographique. Une analyse PCA spatiale sur ces 16 sites européens implique une structure spatiale de la communauté méthanogène (23,7% de la variance totale a été expliquée par cette structure) qui correspond à une autocorrélation spatiale positive des sites comme indiqué par l’indice I de Moran (I de Moran = 0,4018). Seule la première valeur propre était stable et correspondait à une séparation des échantillons entre l’Europe centrale et les États baltes (figure supplémentaire S5). La petite taille de l’ensemble de données complique cependant l’attribution robuste de cette structure spatiale observée à des variables géographiques, environnementales ou aux deux. Nous avons donc effectué une analyse de regroupement sur les communautés méthanogènes comme décrit ci-dessus et avons révélé trois groupes (Figure 7A) que nous avons projetés sur une carte géographique (Figure 7B). Le clustering n’a pas reproduit la séparation de l’ACP spatiale le long de la mer Baltique. En conséquence, certains sites géographiquement très proches les uns des autres présentent des structures de communautés méthanogènes dissemblables et s’assemblent en différents groupes. D’autre part, certains sites géographiquement très éloignés présentent des compositions de communautés très similaires et se regroupent (Figure 7B). La dispersion régionale des groupes en Europe ne semble pas correspondre ou être limitée par une structure géographique.
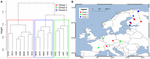
FIGURE 7. Analyse de cluster hiérarchique de la matrice de distance de Jaccard parmi 16 échantillons européens de sols et de sédiments lacustres. La méthode de clustering de Ward a été utilisée pour l’analyse. Les sites ont été divisés en trois groupes colorés par différents rectangles dans le dendrogramme de clustering (A) et ont été projetés sur une carte européenne (B).
Discussion
Identifier et appliquer les concepts de biogéographie sur les communautés microbiennes est d’un intérêt majeur en écologie microbienne. On pense que la biogéographie microbienne est régie par l’interaction évolutive et écologique de quatre processus majeurs : le filtrage de l’habitat, la dispersion, la dérive et la mutation (Hanson et al., 2012). Même si l’influence de la dérive et de la mutation dépasse le pouvoir d’interprétation de cette étude, nous montrons qu’il existe des modèles mondiaux de communautés méthanogènes dans les environnements naturels. Cette étude démontre un modèle biogéographique global des communautés méthanogènes qui est plus associé au filtrage de l’habitat qu’à la dispersion géographique. Les communautés méthanogènes des sédiments de lacs sodiques, par exemple, se regroupent étroitement avec des échantillons marins géographiquement éloignés (Figure 4) et des communautés méthanogènes très similaires sont présentes dans les sols et les sédiments lacustres européens bien qu’ils soient très éloignés les uns des autres (Figure 7). Dans l’ensemble, nos tentatives de révéler une limitation potentielle de la dispersion ont révélé une faible influence de la localisation géographique sur la structure de la communauté méthanogène, ce qui contraste avec une influence claire des conditions environnementales. Une exclusion concluante des effets spatiaux sur les communautés microbiennes n’est pas possible avec les points de données disponibles. Les points d’échantillonnage se concentrent sur certains habitats et/ou zones alors que le nombre total d’échantillons est faible. S’il existe un effet, nous supposons que les effets spatiaux se produisent uniquement à l’échelle régionale ou locale. Des limitations locales de la dispersion ont été signalées pour les archées hyperthermophiles en raison de barrières géographiques (Whitaker et al., 2003), pour les bactéries oxydant l’ammoniac dans les marais salants locaux, mais pas à l’échelle régionale ou continentale (Martiny et al., 2011), pour les micro-organismes dans les sédiments des eaux profondes en fonction des paramètres environnementaux (Schauer et al., 2011).2009), et pour les bactéries d’un large ensemble d’environnements de neige hétérogènes, principalement en raison de la disponibilité de carbone allochtone (Lutz et al., 2016). Une influence mineure de la limitation de la dispersion sur les méthanogènes dans les environnements naturels signifie que les méthanogènes peuvent se distribuer aléatoirement dans l’espace, sont sélectionnés avec succès par l’environnement local si leurs exigences physiologiques sont satisfaites et peuvent établir des communautés stables (Martiny et al., 2006 ; von Mering et al., 2007).
Le concept de filtrage de l’habitat implique que les espèces ayant des exigences écologiques similaires devraient cooccurber plus souvent que prévu par hasard (Weiher et Keddy, 2001 ; Cornwell et al., 2006 ; Ulrich et al., 2010). Nos résultats montrent que de grandes différences dans la composition des communautés méthanogènes se produisent entre les écosystèmes marins et ceux des zones humides et des lacs, tandis que les estuaires se situent entre les deux. Des modèles de biogéographie basés sur un ensemble d’études génétiques sur des échantillons environnementaux ont également été signalés pour les bactéries générales (Nemergut et al., 2011), le microbiome des suintements de méthane (Ruff et al., 2015), les archées oxydant l’ammoniac (Cao et al., 2013), les bactéries pélagiques et benthiques marines (Zinger et al., 2011) et les micro-organismes recyclant l’azote (Church et al., 2008). Le filtrage de l’habitat a été spécifiquement signalé pour des archées non cultivées (Auguet et al., 2010), des communautés bactériennes entières dans divers environnements (von Mering et al., 2007 ; Chaffron et al., 2010) ou dans les tourbières d’Amérique du Sud à l’échelle régionale (Oloo et al, 2016), ainsi que des groupes bactériens spécifiques tels que les bactéries oxydant le méthane (Knief, 2015) et les bactéries fixant l’azote (Nelson et al., 2016).
Nos résultats indiquent qu’à l’échelle globale, la salinité régule substantiellement la composition des communautés méthanogènes et détermine de grandes différences entre les assemblages méthanogènes marins et terrestres. De même, les méthanogènes des sédiments des lacs sodiques se regroupent avec ceux des sédiments marins (figure 4), mettant en évidence l’influence globale de la salinité. Ce résultat est en accord avec d’autres études basées sur le gène de l’ARNr 16S qui révèlent que la salinité est un facteur primaire qui façonne les modèles globaux des communautés bactériennes et archéales globales (Lozupone et Knight, 2007 ; Auguet et al., 2010 ; Caporaso et al., 2011 ; Cao et al., 2013). Une faible influence de la séparation géographique mais un fort impact de la salinité sur les communautés microbiennes générales ont également été observés dans des études précédentes (Logares et al., 2013 ; Yang et al., 2016). En conséquence, la salinité détermine en grande partie les lignées qui peuvent survivre. Dans divers habitats, l’activité de production de méthane était négativement corrélée à la salinité (Bartlett et al., 1987 ; Potter et al., 2009 ; Poffenbarger et al., 2011). Il est donc suggéré que l’inhibition de la production de méthane par la salinité coïncide avec une réduction de la taille de la population méthanogène (Pattnaik et al., 2000). L’effet de la salinité sur la méthanogénèse hydrogénotrophique, acétotrophique et méthylotrophique dépend ainsi du niveau de salinité et est différent pour les différentes voies de méthanogénèse (Liu et al., 2016). Actuellement, il n’y a pas de mécanisme clair pour expliquer l’impact de la salinité sur la structure des communautés, mais plusieurs hypothèses peuvent servir d’explication possible. Physiologiquement, la salinité influence l’osmolarité externe et interne des cellules. Les cellules méthanogènes non salines ont développé des adaptations physiologiques pour contrer la pression de turgescence interne, tandis que les cellules adaptées au sel ont perdu cette caractéristique (Zinder, 1993). En outre, l’augmentation de la salinité peut inciter les méthanogènes à synthétiser ou à absorber une proportion accrue de solutés compatibles, ce qui entraîne un coût énergétique et donc métabolique important (McGenity, 2010). Le trait de tolérance au sel se manifeste même dans la concentration optimale de NaCl pour la croissance des cultures pures méthanogènes puisque nous avons constaté que les isolats des sédiments marins et des sédiments hydrothermaux ont une concentration optimale de NaCl significativement plus élevée que ceux des sols (figure supplémentaire S2).
Dans les écosystèmes terrestres non salins, spécifiquement dans les sols et les sédiments lacustres, la composition de la communauté méthanogène est contrôlée par la combinaison de la température et du pH. En conséquence, les méthanogènes de ces environnements peuvent être classés en quatre groupes (figure 6). Contrairement aux écosystèmes marins, les écosystèmes terrestres non salins présentent une grande variabilité naturelle du pH et de la température. La température peut affecter non seulement la voie méthanogène mais aussi les populations méthanogènes elles-mêmes (Conrad, 2007 ; Rooney-Varga et al., 2007). La production de méthane peut être considérablement accrue si la température augmente, en raison des étapes sensibles à la température de la fermentation et de l’acétogenèse (Megonigal et al., 2005 ; Kotsyurbenko et al., 2007). En outre, un pH faible peut limiter considérablement la disponibilité de l’acétate en empêchant la dissociation de l’acétate et ainsi affecter négativement la méthanogénèse acétoclastique (Fukuzaki et al., 1990 ; Bridgham et al., 2013). Cela pourrait être une raison possible de l’absence de Methanosaeta dans le groupe2, alors que Methanosarcina peut passer d’une source à l’autre et n’a pas été influencé de manière substantielle. De plus, le pH peut réguler l’efficacité de la production de méthane et les voies méthanogènes des tourbières ombrotrophes à minérotrophes, par l’inhibition directe des deux voies de méthanogénèse et indirectement par ses effets sur la fermentation (Ye et al., 2012). Par conséquent, la température et le pH peuvent tous deux réguler directement ou indirectement les étapes métaboliques associées à la méthanogénèse et à la fermentation en amont, qui fournit un substrat aux méthanogènes.
Methanoregula est omniprésent et très abondant dans les quatre groupes d’habitats terrestres (Figure 6) mais pratiquement absent du système marin et peut donc s’avérer être un proxy de l’influence de l’eau douce dans le domaine marin. Sa pertinence mondiale a été récemment signalée ailleurs (Yang et al., 2017). Malgré sa distribution omniprésente dans les sols et les sédiments lacustres, Methanoregula apparaît comme une lignée indicatrice dans les habitats acides à température modérée. De plus, (1) Methanolinea semble s’être particulièrement adapté aux environnements terrestres chauds et neutres, (2) Methanobacterium aux environnements froids et acides, et (3) Methanosaeta aux environnements à pH neutre, ce qui est cohérent avec d’autres études (Rosenberg et al., 2014) et souligne la robustesse de notre approche. En général, les conditions géochimiques entourant les communautés méthanogènes conduiront à une différenciation de niche. Comme le tri des niches tend à laisser les spécialistes adaptatifs (Langenheder et Székely, 2011), la sélection environnementale progressive à long terme a généré une variété de niches qui ont été remplies par un ensemble de spécialistes de l’habitat endémique, qui peuvent être moins représentés ou absents dans d’autres conditions environnementales différentes. La communauté est également façonnée par des facteurs biotiques, tels que les interactions écologiques, la dynamique, la compétition et la symbiose. Malgré ces facteurs biologiques, von Mering et al. (2007) ont constaté que les préférences d’habitat sont souvent remarquablement stables dans le temps et que la composition taxonomique distinctive des communautés environnementales, à son tour, peut être un indicateur important de leur écologie et de leur fonction.
Consistant à la préférence d’habitat des archées méthanogènes, il apparaît que des souches méthanogènes étroitement apparentées ont souvent été isolées dans des environnements comparables. Par exemple, Methanoregulaceae semble être assez diversifié dans les environnements naturels (Yang et al., 2017) et les souches neutres peuvent résister à la culture jusqu’à présent. Les souches de Methanoregula actuellement décrites sont toutes deux obtenues dans des environnements légèrement acidophiles, tandis que les deux représentants de Methanolinea proviennent d’habitats relativement chauds, tels que les boues de digesteur et le sol des rizières, respectivement (Rosenberg et al., 2014). Bien que les souches de Methanobacterium aient été isolées de divers environnements, environ la moitié des isolats existants de ce genre présentent des optima de pH légèrement inférieurs à 7. Cela signifie que les lignées indicatrices, qui ont été identifiées sur la base des séquences environnementales, pourraient refléter les différenciations de physiologie et de sources des cultivars méthanogènes existants. Par exemple, la salinité de l’habitat, comme une propriété générale de l’habitat, peut progressivement exposer les organismes à une forte sélection environnementale et filtrer l’assemblage d’un nouvel ensemble d’espèces qui sont les mieux adaptées à la salinité ambiante (Logares et al., 2013).
La conservation et la gestion de la biodiversité est un défi primordial de notre société actuelle. Nous montrons ici que les archées méthanogènes des milieux naturels sont les plus diversifiées dans les sédiments des estuaires. Les estuaires sont des zones de transition entre les écosystèmes marins et terrestres. Cela permet deux processus majeurs qui peuvent contribuer à la richesse des espèces. Premièrement, les microbes provenant de la mer et de la terre se mélangent dans les estuaires et finissent par englober une grande diversité globale (McLusky et Elliott, 2004). Par exemple, on a observé une grande diversité de bactéries, d’archées et de champignons dans les estuaires, et même des bactéries spécifiques remplissant des fonctions uniques (Cunliffe et al., 2008 ; Mosier et Francis, 2008 ; Crump et al., 2012). Un autre aspect est le niveau élevé de nutriments dû aux apports terrestres et de marée dont se nourrissent les organismes de l’estuaire (McLusky et Elliott, 2004 ; Statham, 2012). Dans ce contexte, les environnements estuariens sont importants pour la récupération de la nouveauté générique pour les méthanogènes. Jusqu’à présent, les effets de la diversité des espèces sur les processus des écosystèmes ont suscité d’importants efforts de recherche. Le lien entre la biodiversité et la fonction de l’écosystème fait toujours l’objet de débats et reste insaisissable pour les communautés microbiennes (Loreau et al., 2001 ; Tilman et al., 2014). Même si les sols et les sédiments lacustres sont des sources primaires de méthane et également des habitats à forte diversité méthanogène, nous proposons que la richesse en espèces ne soit pas un indicateur approprié du potentiel de production de méthane et des émissions de méthane des écosystèmes ; elle semble plutôt refléter l’hétérogénéité et l’histoire de l’environnement. Le classement des environnements en fonction de leur richesse en espèces ne signifie pas nécessairement le potentiel des taux d’émission de méthane qui sont les plus élevés dans les sols et les lacs et comparativement mineurs dans les estuaires (Figure 3 et Figure supplémentaire S1).
Enfin, le manque d’informations environnementales dans les bases de données publiques peut avoir empêché une interprétation complète sur les facteurs environnementaux observés ici. Même s’il y a de plus en plus de données de séquençage sur les méthanogènes dans la littérature et les bases de données publiques, les variables abiotiques connexes fournies sont souvent incohérentes et éparses. La quantité limitée d’informations cohérentes pour les variables environnementales limite l’application des analyses statistiques multi-variées. Le fait que les facteurs abiotiques de cette étude ne puissent expliquer qu’une fraction limitée des variances des communautés suggère que d’autres variables explicatives manquent. Les concentrations et la disponibilité des substrats méthanogènes tels que l’acétate, l’hydrogène et les méthylamines pourraient être particulièrement importantes. Néanmoins, les paramètres abiotiques peuvent ne jamais suffire à expliquer complètement ce qui structure les assemblages méthanogènes, simplement parce que les habitats ont des histoires différentes et ne peuvent être étudiés que localement. De plus, parmi les données disponibles sur l’ARMC, les vastes zones du subarctique et de l’arctique russe et canadien sont mal représentées. Une meilleure couverture géographique et une distribution uniforme de l’ensemble des données sur les gènes mcrA amélioreraient une évaluation des communautés méthanogènes à l’échelle mondiale.
Contributions des auteurs
SL et XW ont conçu l’étude. XW et SY ont collecté et analysé les données. XW, SY et FH ont effectué l’analyse statistique. XW, MW et SY ont effectué la correction phylogénétique. XW, SY, FH, MW, DW et SL ont interprété les résultats et rédigé l’article. Tous les auteurs ont contribué aux discussions et ont révisé le manuscrit.
Déclaration de conflit d’intérêts
Les auteurs déclarent que la recherche a été menée en l’absence de toute relation commerciale ou financière qui pourrait être interprétée comme un conflit d’intérêts potentiel.
Remerciements
Le soutien financier à XW (subvention n° 201408620031 à XW) fourni par le China Scholarship Council (CSC) est remercié. Cette étude a été soutenue par la Helmholtz Gemeinschaft (HGF) en finançant le groupe de jeunes chercheurs Helmholtz de SL (VH-NG-919).
Matériel supplémentaire
Le matériel supplémentaire pour cet article peut être trouvé en ligne à : https://www.frontiersin.org/article/10.3389/fmicb.2017.01339/full#supplementary-material
Notes de bas de page
- ^ http://metanogen.biotech.uni.wroc.pl/
- ^ http://www.ncbi.nlm.nih.gov/
- ^ http://metanogen.biotech.uni.wroc.pl/
- ^ https://www.ngdc.noaa.gov/mgg/shorelines/gshhs.html
- ^ http://qgis.osgeo.org
Auguet, J. C., Barberan, A., et Casamayor, E. O. (2010). Modèles écologiques globaux dans les archées non cultivées. ISME J. 4, 182-190. doi : 10.1038/ismej.2009.109
PubMed Abstract | CrossRef Full Text | Google Scholar
Bange, H. W., Bartell, U. H., Rapsomanikis, S., and Andreae, M. O. (1994). Le méthane dans la mer Baltique et la mer du Nord et une réévaluation des émissions marines de méthane. Glob. Biogeochem. Cycles 8, 465-480. doi : 10.1029/94GB02181
CrossRef Full Text | Google Scholar
Barreto, D. P., Conrad, R., Klose, M., Claus, P., and Enrich-Prast, A. (2014). Décroissance de la distance et relations taxa-area pour les bactéries, les archées et les archées méthanogènes dans un sédiment lacustre tropical. PLoS ONE 9:e110128. doi : 10.1371/journal.pone.0110128
PubMed Abstract | CrossRef Full Text | Google Scholar
Bartlett, K. B., Bartlett, D. S., Harriss, R. C., and Sebacher, D. I.. (1987). Methane emissions along a salt marsh salinity gradient. Biogeochemistry 4, 183-202. doi : 10.1007/BF02187365
CrossRef Full Text | Google Scholar
Borrel, G., O’Toole, P. W., Harris, H. M. B., Peyret, P., Brugere, J. F., and Gribaldo, S. (2013). Les données phylogénomiques soutiennent un septième ordre de méthanogènes méthylotrophes et fournissent des informations sur l’évolution de la méthanogenèse. Genome Biol. Evol. 5, 1769-1780. doi : 10.1093/Gbe/Evt128
PubMed Abstract | CrossRef Full Text | Google Scholar
Bousquet, P., Ciais, P., Miller, J., Dlugokencky, E., Hauglustaine, D., Prigent, C., et al. (2006). Contribution des sources anthropiques et naturelles à la variabilité du méthane atmosphérique. Nature 443, 439-443. doi : 10.1038/nature05132
PubMed Abstract | CrossRef Full Text | Google Scholar
Bridgham, S. D., Cadillo-Quiroz, H., Keller, J. K., et Zhuang, Q. (2013). Émissions de méthane des zones humides : perspectives biogéochimiques, microbiennes et de modélisation de l’échelle locale à l’échelle mondiale. Glob. Chang. Biol. 19, 1325-1346. doi : 10.1111/gcb.12131
PubMed Abstract | CrossRef Full Text | Google Scholar
Cao, H., Auguet, J.-C., et Gu, J.-D.. (2013). Modèle écologique mondial des archées oxydant l’ammoniac. PLoS ONE 8:e52853. doi : 10.1371/journal.pone.0052853
PubMed Abstract | CrossRef Full Text | Google Scholar
Caporaso, J. G., Lauber, C. L., Walters, W. A., Berg-Lyons, D., Lozupone, C. A., Turnbaugh, P. J., et al. (2011). Global patterns of 16S rRNA diversity at a depth of millions of sequences per sample. Proc. Natl. Acad. Sci. U.S.A. 108, 4516-4522. doi : 10.1073/pnas.1000080107
PubMed Abstract | CrossRef Full Text | Google Scholar
Chaffron, S., Rehrauer, H., Pernthaler, J., et von Mering, C. (2010). Un réseau mondial de microbes coexistants à partir de données environnementales et de séquences du génome entier. Genome Res. 20, 947-959. doi : 10.1101/gr.104521.109
PubMed Abstract | CrossRef Full Text | Google Scholar
Church, M. J., Bjorkman, K. M., Karl, D. M., Saito, M. A., and Zehr, J. P. (2008). Regional distributions of nitrogen-fixing bacteria in the Pacific Ocean. Limnol. Oceanogr. 53, 63-77. doi : 10.4319/lo.2008.53.1.0063
CrossRef Full Text | Google Scholar
Conrad, R. (2007). » Écologie microbienne des méthanogènes et des méthanotrophes « , dans Advances in Agronomy, ed. L. S. Donald (Cambridge, MA : Academic Press), 1-63.
Google Scholar
Cornwell, W. K., Schwilk, D. W., and Ackerly, D. D. (2006). Un test basé sur les traits pour le filtrage de l’habitat : volume de la coque convexe. Ecology 87, 1465-1471. doi : 10.1890/0012-9658(2006)872.0.CO;2
PubMed Abstract | CrossRef Full Text | Google Scholar
Crump, B. C., Amaral-Zettler, L. A., et Kling, G. W. (2012). La diversité microbienne dans les eaux douces arctiques est structurée par l’inoculation de microbes provenant de sols. ISME J. 6, 1629-1639. doi : 10.1038/ismej.2012.9
PubMed Abstract | CrossRef Full Text | Google Scholar
Cui, M., Ma, A., Qi, H., Zhuang, X., Zhuang, G., et Zhao, G. (2015). Une température plus chaude accélère les émissions de méthane de la zone humide de Zoige sur le plateau tibétain sans changer la composition de la communauté méthanogène. Sci. Rep. 5:11616. doi : 10.1038/srep11616
PubMed Abstract | CrossRef Full Text | Google Scholar
Cunliffe, M., Schafer, H., Harrison, E., Cleave, S., Upstill-Goddard, R., et Murrell, J. C. (2008). Analyse phylogénétique et fonctionnelle des gènes des communautés bactériennes et archéales associées à la microcouche de surface d’un estuaire. ISME J. 2, 776-789. doi : 10.1038/ismej.2008.28
PubMed Abstract | CrossRef Full Text | Google Scholar
Dray, S., et Dufour, A. B. (2007). Le paquet ade4 : mise en œuvre du diagramme de dualité pour les écologistes. J. Stat. Softw. 22, 1-20. doi : 10.18637/jss.v022.i04
CrossRef Full Text | Google Scholar
Dufrene, M., and Legendre, P. (1997). Assemblages d’espèces et espèces indicatrices : la nécessité d’une approche asymétrique souple. Ecol. Monogr. 67, 345-366. doi : 10.2307/2963459
CrossRef Full Text | Google Scholar
Edgar, R. C., Haas, B. J., Clemente, J. C., Quince, C., and Knight, R. (2011). UCHIME améliore la sensibilité et la vitesse de détection des chimères. Bioinformatics 27, 2194-2200. doi : 10.1093/bioinformatics/btr381
PubMed Abstract | CrossRef Full Text | Google Scholar
Evans, P. N., Parks, D. H., Chadwick, G. L., Robbins, S. J., Orphan, V. J., Golding, S. D., et al. (2015). Le métabolisme du méthane dans le phylum archéen Bathyarchaeota révélé par la métagénomique centrée sur le génome. Science 350, 434-438. doi : 10.1126/science.aac7745
PubMed Abstract | CrossRef Full Text | Google Scholar
Fish, J. A., Chai, B., Wang, Q., Sun, Y., Brown, C. T., Tiedje, J. M., et al. (2013). FunGene : le pipeline et le référentiel de gènes fonctionnels. Front. Microbiol. 4:291. doi : 10.3389/fmicb.2013.00291
PubMed Abstract | CrossRef Full Text | Google Scholar
Frank-Fahle, B. A., Yergeau, E., Greer, C. W., Lantuit, H., et Wagner, D. (2014). Potentiel fonctionnel microbien et composition des communautés dans les sols affectés par le pergélisol dans le Nord-Ouest de l’Arctique canadien. PLoS ONE 9:e84761. doi : 10.1371/journal.pone.0084761.g001
PubMed Abstract | CrossRef Full Text | Google Scholar
Fukuzaki, S., Nishio, N., et Nagai, S. (1990). Cinétique de la fermentation méthanogène de l’acétate. Appl. Environ. Microbiol. 56, 3158-3163.
Google Scholar
Hanson, C. A., Fuhrman, J. A., Horner-Devine, M. C., et Martiny, J. B. (2012). Au-delà des modèles biogéographiques : les processus qui façonnent le paysage microbien. Nat. Rev. Microbiol. 10, 497-506. doi : 10.1038/Nrmicro2795
PubMed Abstract | CrossRef Full Text | Google Scholar
Holm, G. O., Perez, B. C., McWhorter, D. E., Krauss, K. W., Johnson, D. J., Raynie, R. C., et al. (2016). Flux de méthane au niveau de l’écosystème à partir des marais d’eau douce et saumâtre de marée du delta du fleuve Mississippi : implications pour les projets de carbone des zones humides côtières. Wetlands 36, 401-413. doi : 10.1007/s13157-016-0746-7
CrossRef Full Text | Google Scholar
Hunger, S., Schmidt, O., Hilgarth, M., Horn, M. A., Kolb, S., Conrad, R., et al. (2011). Competing formate- and carbon dioxide-utilizing procaryotes in anoxic methane-emitting fen soil. Appl. Environ. Microbiol. 77, 3773-3785. doi : 10.1128/AEM.00282-11
PubMed Abstract | CrossRef Full Text | Google Scholar
GIEC (2007). » Climate Change 2007 : the Physical Science Basis « , dans Proceedings of the Contribution of Working Group I to the Fourth Assessment Report of the Intergovernmental Panel on Climate Change, eds D. Qin, S. Solomon, M. Manning, Z. Chen, M. Marquis, K. B. Averyt, et al. (Cambridge : Cambridge University Press).
Google Scholar
Jabłoński, S., Rodowicz, P., et Łukaszewicz, M. (2015). Base de données des archées méthanogènes contenant des caractéristiques physiologiques et biochimiques. Int. J. Syst. Evol. Microbiol. 65, 1360-1368. doi : 10.1099/ijs.0.000065
PubMed Abstract | CrossRef Full Text | Google Scholar
Kirschke, S., Bousquet, P., Ciais, P., Saunois, M., Canadell, J. G., Dlugokencky, E. J., et al. (2013). Trois décennies de sources et de puits de méthane à l’échelle mondiale. Nat. Geosci. 6, 813-823. doi : 10.1038/Ngeo1955
CrossRef Full Text | Google Scholar
Knief, C. (2015). Diversité et préférences d’habitat des bactéries méthanotrophes aérobies cultivées et non cultivées évaluées sur la base de pmoA comme marqueur moléculaire. Front. Microbiol. 6:1346. doi : 10.3389/fmicb.2015.01346
PubMed Abstract | CrossRef Full Text | Google Scholar
Kotsyurbenko, O., Friedrich, M., Simankova, M., Nozhevnikova, A., Golyshin, P., Timmis, K., et al. (2007). Shift from acetoclastic to H2-dependent methanogenesis in a West Siberian peat bog at low pH values and isolation of an acidophilic Methanobacterium strain. Appl. Environ. Microbiol. 73, 2344-2348. doi : 10.1128/AEM.02413-06
PubMed Abstract | CrossRef Full Text | Google Scholar
Langenheder, S., et Székely, A. J. (2011). Le tri des espèces et les processus neutres sont tous deux importants lors de l’assemblage initial des communautés bactériennes. ISME J. 5, 1086-1094. doi : 10.1038/ismej.2010.207
PubMed Abstract | CrossRef Full Text | Google Scholar
Liebner, S., Ganzert, L., Kiss, A., Yang, S., Wagner, D., et Svenning, M. M. (2015). Shifts in methanogenic community composition and methane fluxes along the degradation of discontinuous permafrost. Front. Microbiol. 6:356. doi : 10.3389/fmicb.2015.00356
PubMed Abstract | CrossRef Full Text | Google Scholar
Liu, Y. Q., Priscu, J. C., Xiong, J. B., Conrad, R., Vick-Majors, T., Chu, H. Y., et al. (2016). La salinité conduit les modèles de distribution archéale dans les sédiments lacustres de haute altitude sur le plateau tibétain. FEMS Microbiol. Ecol. 92:fiw033. doi : 10.1093/femsec/fiw033
PubMed Abstract | CrossRef Full Text | Google Scholar
Logares, R., Lindstrom, E. S., Langenheder, S., Logue, J. B., Paterson, H., Laybourn-Parry, J., et al. (2013). Biogéographie des communautés bactériennes exposées à un changement environnemental progressif à long terme. ISME J. 7, 937-948. doi : 10.1038/ismej.2012.168
PubMed Abstract | CrossRef Full Text | Google Scholar
Loreau, M., Naeem, S., Inchausti, P., Bengtsson, J., Grime, J. P., Hector, A., et al. (2001). Biodiversité et fonctionnement des écosystèmes : connaissances actuelles et défis futurs. Science 294, 804-808. doi : 10.1126/science.1064088
PubMed Abstract | CrossRef Full Text | Google Scholar
Lozupone, C. A., et Knight, R. (2007). Modèles mondiaux de la diversité bactérienne. Proc. Natl. Acad. Sci. U.S.A. 104, 11436-11440. doi : 10.1073/pnas.0611525104
PubMed Abstract | CrossRef Full Text | Google Scholar
Luton, P. E., Wayne, J. M., Sharp, R. J., and Riley, P. W. (2002). Le gène mcrA comme une alternative à l’ARNr 16S dans l’analyse phylogénétique des populations de méthanogènes dans les décharges. Microbiology 148, 3521-3530. doi : 10.1099/00221287-148-11-3521
PubMed Abstract | CrossRef Full Text | Google Scholar
Lutz, S., Anesio, A. M., Raiswell, R., Edwards, A., Newton, R. J., Gill, F., et al. (2016). La biogéographie des microbiomes de la neige rouge et leur rôle dans la fonte des glaciers arctiques. Nat. Commun. 7:11968. doi : 10.1038/ncomms11968
PubMed Abstract | CrossRef Full Text | Google Scholar
Martiny, J. B. H., Bohannan, B. J. M., Brown, J. H., Colwell, R. K., Fuhrman, J. A., Green, J. L., et al. (2006). Microbial biogeography : putting microorganisms on the map. Nat. Rev. Microbiol. 4, 102-112. doi : 10.1038/Nrmicro1341
PubMed Abstract | CrossRef Full Text | Google Scholar
Martiny, J. B. H., Eisen, J. A., Penn, K., Allison, S. D., et Horner-Devine, M. C. (2011). Drivers of bacterial beta-diversity depend on spatial scale. Proc. Natl. Acad. Sci. U.S.A. 108, 7850-7854. doi : 10.1073/pnas.1016308108
PubMed Abstract | CrossRef Full Text | Google Scholar
McArdle, B. H., et Anderson, M. J. (2001). Fitting multivariate models to community data : a comment on distance-based redundancy analysis. Ecology 82, 290-297. doi : 10.1890/0012-9658(2001)0822.0.CO;2
CrossRef Full Text | Google Scholar
McCalley, C. K., Woodcroft, B. J., Hodgkins, S. B., Wehr, R. A., Kim, E.-H., Mondav, R., et al. (2014). La dynamique du méthane régulée par la réponse de la communauté microbienne au dégel du permafrost. Nature 514, 478-481. doi : 10.1038/nature13798
PubMed Abstract | CrossRef Full Text | Google Scholar
McGenity, T. J. (2010). » Methanogens and Methanogenesis in Hypersaline Environments « , dans Handbook of Hydrocarbon and Lipid Microbiology, ed. K. N. Timmis (Berlin : Springer), 665-680. doi : 10.1007/978-3-540-77587-4_53
CrossRef Full Text | Google Scholar
McLusky, D. S., et Elliott, M. (2004). L’écosystème estuarien : Ecology, Threats and Management. New York, NY : Oxford University Press. doi : 10.1093/acprof:oso/9780198525080.001.0001
CrossRef Full Text | Google Scholar
Megonigal, J. P., Mines, M. E., et Visscher, P. T. (2005). « Le métabolisme anaérobie : liens avec les gaz à l’état de traces et les processus aérobies », dans Biogeochemistry, ed. W. H. Schlesinger (Oxford : Elsevier), 350-362.
Google Scholar
Milferstedt, K., Youngblut, N. D., and Whitaker, R. J. (2010). Structure spatiale et persistance des populations de méthanogènes dans les lacs de tourbières humiques. ISME J. 4, 764-776. doi : 10.1038/Ismej.2010.7
PubMed Abstract | CrossRef Full Text | Google Scholar
Mosier, A. C., et Francis, C. A. (2008). Abondance relative et diversité des archées et bactéries oxydant l’ammoniac dans l’estuaire de la baie de San Francisco. Environ. Microbiol. 10, 3002-3016. doi : 10.1111/j.1462-2920.2008.01764.x
PubMed Abstract | CrossRef Full Text | Google Scholar
Nazaries, L., Murrell, J. C., Millard, P., Baggs, L., et Singh, B. K. (2013). Méthane, microbes et modèles : compréhension fondamentale du cycle du méthane du sol pour les prédictions futures. Environ. Microbiol. 15, 2395-2417. doi : 10.1111/1462-2920.12149
PubMed Abstract | CrossRef Full Text | Google Scholar
Nelson, M. B., Martiny, A. C., et Martiny, J. B. H. (2016). Biogéographie mondiale des traits microbiens de recyclage de l’azote dans le sol. Proc. Natl. Acad. Sci. U.S.A. 113, 8033-8040. doi : 10.1073/pnas.1601070113
PubMed Abstract | CrossRef Full Text | Google Scholar
Nemergut, D. R., Costello, E. K., Hamady, M., Lozupone, C., Jiang, L., Schmidt, S. K., et al. (2011). Global patterns in the biogeography of bacterial taxa. Environ. Microbiol. 13, 135-144. doi : 10.1111/j.1462-2920.2010.02315.x
PubMed Abstract | CrossRef Full Text | Google Scholar
Nobu, M. K., Narihiro, T., Kuroda, K., Mei, R., et Liu, W. T. (2016). Chasser la classe insaisissable d’Euryarchaeota WSA2 : les génomes révèlent un méthanogène méthyl-réducteur fastidieux unique. ISME J. 10, 2478-2487. doi : 10.1038/ismej.2016.33
PubMed Abstract | CrossRef Full Text | Google Scholar
Oksanen, J., Blanchet, F. G., Kindt, R., Legendre, P., Minchin, P. R., O’Hara, R. B., et al. (2015). vegan : Community Ecology Package. Paquet R version 2.2.0. Disponible à : https://CRAN.R-project.org/package=vegan
Google Scholar
Oloo, F., Valverde, A., Quiroga, M. V., Vikram, S., Cowan, D., et Mataloni, G. (2016). L’hétérogénéité et la connectivité de l’habitat façonnent les communautés microbiennes dans les tourbières sud-américaines. Sci. Rep. 6:25712. doi : 10.1038/srep25712
PubMed Abstract | CrossRef Full Text | Google Scholar
Pattnaik, P., Mishra, S. R., Bharati, K., Mohanty, S. R., Sethunathan, N., and Adhya, T. K. (2000). Influence de la salinité sur la méthanogénèse et la microflore associée dans les sols de riz tropicaux. Microbiol. Res. 155, 215-220. doi : 10.1016/S0944-5013(00)80035-X
PubMed Abstract | CrossRef Full Text | Google Scholar
Poffenbarger, H. J., Needelman, B. A., et Megonigal, J. P. (2011). Influence de la salinité sur les émissions de méthane des marais à marée. Wetlands 31, 831-842. doi : 10.1007/s13157-011-0197-0
PubMed Abstract | CrossRef Full Text | Google Scholar
Potter, E. G., Bebout, B. M., and Kelley, C. A. (2009). Composition isotopique du méthane et des substrats méthanogènes présumés le long d’un gradient de salinité dans un système de tapis microbien hypersalin. Astrobiologie 9, 383-390. doi : 10.1089/ast.2008.0260
PubMed Abstract | CrossRef Full Text | Google Scholar
R Core Team (2014). R : un langage et un environnement pour le calcul statistique (version 3.0.3). Vienne : R Foundation for Statistical Computing.
Google Scholar
Roberts, D. (2016). labdsv : Ordination et analyse multivariée pour l’écologie. Paquet R version 1.8-0. Disponible à l’adresse suivante : https://CRAN.R-project.org/package=labdsv
Google Scholar
Rooney-Varga, J. N., Giewat, M. W., Duddleston, K. N., Chanton, J. P., et Hines, M. E. (2007). Links between archaeal community structure, vegetation type and methanogenic pathway in Alaskan peatlands. FEMS Microbiol. Ecol. 60, 240-251. doi : 10.1111/j.1574-6941.2007.00278.x
PubMed Abstract | CrossRef Full Text | Google Scholar
Rosenberg, E., DeLong, E. F., Lory, S., Stackebrandt, E., et Thompson, F. (2014). The Prokaryotes : Les autres grandes lignées de bactéries et les archées. Berlin : Springer.
Google Scholar
Ruff, S. E., Biddle, J. F., Teske, A. P., Knittel, K., Boetius, A., et Ramette, A. (2015). Dispersion globale et diversification locale du microbiome des suintements de méthane. Proc. Natl. Acad. Sci. U.S.A. 112, 4015-4020. doi : 10.1073/pnas.1421865112
PubMed Abstract | CrossRef Full Text | Google Scholar
Schauer, R., Bienhold, C., Ramette, A., et Harder, J. (2009). Diversité et biogéographie bactériennes dans les sédiments de surface des eaux profondes de l’océan Atlantique Sud. ISME J. 4, 159-170. doi : 10.1038/ismej.2009.106
PubMed Abstract | CrossRef Full Text | Google Scholar
Schloss, P. D., Westcott, S. L., Ryabin, T., Hall, J. R., Hartmann, M., Hollister, E. B., et al. (2009). Introducing mothur : open-source, platform-independent, community-supported software for describing and comparing microbial communities. Appl. Environ. Microbiol. 75, 7537-7541. doi : 10.1128/Aem.01541-09
PubMed Abstract | CrossRef Full Text | Google Scholar
Statham, P. J. (2012). Les nutriments dans les estuaires – un aperçu et les impacts potentiels du changement climatique. Sci. Total Environ. 434, 213-227. doi : 10.1016/j.scitotenv.2011.09.088
PubMed Abstract | CrossRef Full Text | Google Scholar
Thauer, R. K., Kaster, A.-K., Seedorf, H., Buckel, W., et Hedderich, R. (2008). Archées méthanogènes : différences écologiquement pertinentes dans la conservation de l’énergie. Nat. Rev. Microbiol. 6, 579-591. doi : 10.1038/Nrmicro1931
PubMed Abstract | CrossRef Full Text | Google Scholar
Tilman, D., Isbell, F., et Cowles, J. M. (2014). La biodiversité et le fonctionnement des écosystèmes. Annu. Rev. Ecol. Evol. Syst. 45, 471-493. doi : 10.1146/annurev-ecolsys-120213-091917
CrossRef Full Text | Google Scholar
Ulrich, W., Hajdamowicz, I., Zalewski, M., Stañska, M., Ciurzycki, W., et Tykarski, P. (2010). Assortiment d’espèces ou filtrage de l’habitat : une étude de cas des communautés d’araignées sur les îles du lac. Ecol. Res. 25, 375-381. doi : 10.1007/s11284-009-0661-y
CrossRef Full Text | Google Scholar
Vanwonterghem, I., Evans, P. N., Parks, D. H., Jensen, P. D., Woodcroft, B. J., Hugenholtz, P., et al. (2016). La méthanogénèse méthylotrophique découverte dans le phylum archéen Verstraetearchaeota. Nat. Microbiol. 1:16170. doi : 10.1038/nmicrobiol.2016.170
PubMed Abstract | CrossRef Full Text | Google Scholar
von Mering, C., Hugenholtz, P., Raes, J., Tringe, S. G., Doerks, T., Jensen, L. J., et al. Évaluation phylogénétique quantitative des communautés microbiennes dans divers environnements. Science 315, 1126-1130. doi : 10.1126/science.1133420
PubMed Abstract | CrossRef Full Text | Google Scholar
Wagner, D., et Liebner, S. (2009). « Réchauffement climatique et dynamique du carbone dans les sols du pergélisol : production et oxydation du méthane », dans Permafrost Soils, ed. R. Margesin (Berlin : Springer), 219-236.
Google Scholar
Weiher, E., et Keddy, P. (2001). Règles d’assemblage écologique : Perspectives, avances, retraites. Cambridge : Cambridge University Press.
Google Scholar
Whitaker, R. J., Grogan, D. W., et Taylor, J. W. (2003). Les barrières géographiques isolent les populations endémiques d’archées hyperthermophiles. Science 301, 976-978. doi : 10.1126/science.1086909
PubMed Abstract | CrossRef Full Text | Google Scholar
Wickham, H. (2009). ggplot2 : Graphiques élégants pour l’analyse de données. New York, NY : Springer-Verlag. doi : 10.1007/978-0-387-98141-3
CrossRef Full Text | Google Scholar
Yang, J., Ma, L. A., Jiang, H., Wu, G., and Dong, H. (2016). La salinité façonne la diversité microbienne et la structure des communautés dans les sédiments de surface des lacs du Qinghai-Tibet. Sci. Rep. 6:25078. doi : 10.1038/srep25078
PubMed Abstract | CrossRef Full Text | Google Scholar
Yang, S., Liebner, S., Alawi, M., Ebenhöh, O., et Wagner, D. (2014). Base de données taxonomiques et valeur seuil pour le traitement des données de pyroséquençage 454 du gène mcrA par MOTHUR. J. Microbiol. Methods 103, 3-5. doi : 10.1016/j.mimet.2014.05.006
PubMed Abstract | CrossRef Full Text | Google Scholar
Yang, S., Liebner, S., Winkel, M., Alawi, M., Horn, F., Dörfer, C., et al. (2017). Analyse approfondie des communautés méthanogènes centrales des zones humides affectées par le pergélisol en haute altitude. Soil Biol. Biochem. 111, 66-77. doi : 10.1016/j.soilbio.2017.03.007
CrossRef Full Text | Google Scholar
Ye, R. Z., Jin, Q. S., Bohannan, B., Keller, J. K., McAllister, S. A., et Bridgham, S. D. (2012). pH controls over anaerobic carbon mineralization, the efficiency of methane production, and methanogenic pathways in peatlands across an ombrotrophic-minerotrophic gradient. Soil Biol. Biochem. 54, 36-47. doi : 10.1016/j.soilbio.2012.05.015
CrossRef Full Text | Google Scholar
Zinder, S. H. (1993). » Écologie physiologique des méthanogènes « , dans Methanogenesis : Ecology, Physiology, Biochemistry & Genetics, ed. J. G. Ferry (Boston, MA : Springer), 128-206.
Google Scholar
Zinger, L., Amaral-Zettler, L. A., Fuhrman, J. A., Horner-Devine, M. C., Huse, S. M., Welch, D. B. M., et al. (2011). Global patterns of bacterial beta-diversity in seafloor and seawater ecosystems. PLoS ONE 6:e24570. doi : 10.1371/journal.pone.0024570
PubMed Abstract | CrossRef Full Text | Google Scholar
.