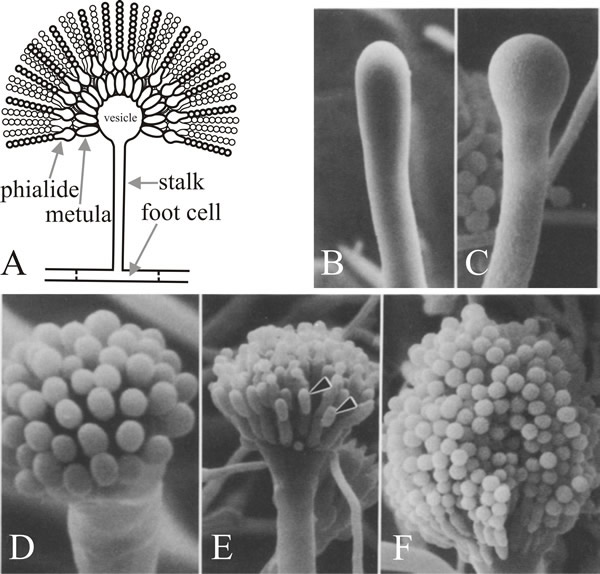
La célula de la que emerge la rama es la célula del pie del conidióforo, que se distingue de otras células vegetativas por tener un engrosamiento de la pared secundaria pigmentada de color marrón en el interior de su pared original. El tallo crece apicalmente hasta alcanzar una longitud de unos 100 µm cuando el ápice se hincha para formar la vesícula del conidióforo que tiene un diámetro de unos 10 µm. De la vesícula brota un único nivel de numerosos esterigmas primarios, denominados métulas, y de los ápices expuestos de las métulas brotan esterigmas secundarios, los fiálides. Las fiálides son las células madre que posteriormente sufren repetidas divisiones asimétricas para formar las largas cadenas de conidios enteroblásticos de aproximadamente 3 µm de diámetro (Fig. 7).
El análisis genético clásico, mediante el aislamiento y análisis de mutantes, estableció el esquema genético básico de la conidiación de Aspergillus. Se estimó que entre 300 y 1.000 loci de genes estaban relacionados con la conidiación, comparando las frecuencias de mutación en los loci que afectan a la conidiación con las de otras funciones (Martinelli & Clutterbuck, 1971). El análisis de las especies de ARNm indicó que aproximadamente 6.000 se expresaban en el micelio vegetativo y otros 1.200 se encontraban en cultivos que incluían conidióforos y conidios; 200 de estos ARNm adicionales se encontraban en los propios conidios. Sólo alrededor del 2% de los mutantes de A. nidulans que carecían de conidios presentaban defectos en las etapas relacionadas con el crecimiento y el desarrollo de los conidióforos, y el 85% de los mutantes de conidiación también eran defectuosos en el crecimiento vegetativo de los hifos y en la consecución de la competencia.
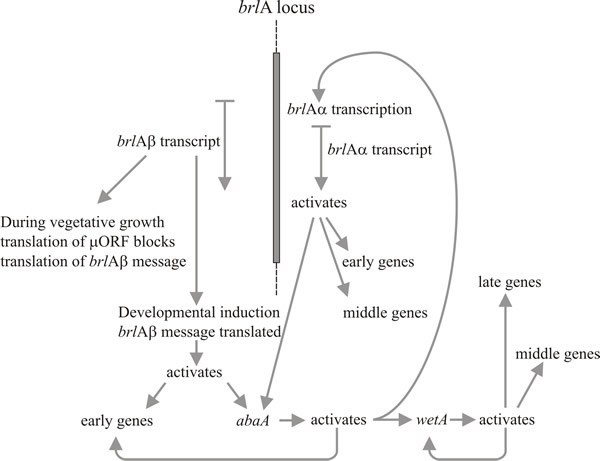
Dos genes, en particular, desempeñan un papel clave en la morfogénesis del conidióforo: se trata del gen «bristle» (brlA), que tiene defectos en la formación de vesículas y metsulas, y de «abacus» (abaA), en el que los conidios son sustituidos por longitudes de hifas en forma de cuentas, por lo que se presume que es defectuoso en la brotación de conidios desde la fiálida y la septación final. Un tercer gen, wetA, es defectuoso en una etapa temprana de la maduración de las esporas. Los conidios de los mutantes de wetA carecen de pigmento e hidrofobicidad; se autolizan después de unas horas y no expresan una serie de ARNm específicos de las esporas. El transcrito del gen wetA falta en los mutantes brlA y abaA (es decir, brlA y abaA son epistáticos con respecto a wetA), y los estudios de mutantes dobles muestran que estos tres genes actúan en el orden: brlA → abaA → wetA.
Una característica sorprendente del análisis mutacional del desarrollo del conidióforo en A. nidulans es que los mutantes de sólo estos tres genes causan defectos en la morfogénesis del conidióforo y de las esporas, mientras que hasta 1.000 otros loci génicos causan ausencia de conidiación cuando están mutados. Esto implica que brlA, abaA y wetA son reguladores que integran la expresión de otros genes que son necesarios para la conidiación pero que no se dedican ellos mismos a ella. Muchos de los mutantes de conidiación de Aspergillus también eran defectuosos en la reproducción sexual, por lo que otra conclusión a la que se puede llegar es que existe cierta economía de uso de los genes morfogenéticos en los diferentes procesos de desarrollo. Es de suponer que los diferentes modos de desarrollo emplean genes estructurales que no son exclusivamente de desarrollo, sino que funcionan en numerosas vías, teniendo su especificidad de desarrollo otorgada por los reguladores a los que responden. Esto se resume en la idea de que la clave del desarrollo de los eucariotas está en la capacidad de utilizar relativamente pocos genes reguladores para integrar las actividades de muchos otros.
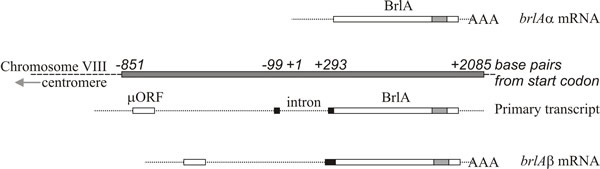
El análisis molecular apoya la interpretación de que brlA, abaA y wetA son reguladores; la secuencia brlA codifica una proteína de dedo de zinc, que es un activador de la transcripción de unión al ADN específico de los genes diana regulados por el desarrollo (Fig. 8). Sin embargo, eso no es todo, porque el producto brlA tiene diferentes afinidades por diferentes genes diana. De hecho, el locus brlA consta de unidades de transcripción superpuestas (Fig. 9), la unidad descendente se designa brlAα y la unidad ascendente brlAβ; entre ellas, sus productos resuelven dos problemas clásicos del desarrollo, cómo responder a una señal y cómo mantener esa respuesta cuando la señal se ha disipado.
Las dos unidades de transcripción de brlA comparten el mismo marco de lectura en la mayor parte de su longitud, pero brlAβ tiene 23 residuos de aminoácidos adicionales en el extremo aminoterminal de ese marco de lectura, y su transcripción también posee un marco de lectura separado de 41 residuos de aminoácidos (denominado µORF) cerca de su extremo 5′. El producto µORF reprime la traducción del marco de lectura posterior (BrlA) y aunque el transcrito brlAβ puede detectarse en las hifas vegetativas, no se produce el péptido BrlA. La represión causada por µORF sólo se supera cuando el micelio es competente, lo que probablemente se señala como una limitación de nitrógeno (una señal ambiental común para el inicio de la esporulación en Ascomycota), que reduce las reservas de aminoacil-ARNt y perturba la regulación traslacional por µORF. Cuando el efecto de µORF se estanca, el péptido BrlA puede traducirse a partir del transcrito existente.
El descubridor de este proceso describió la activación de la vía de conidiación como «activación traslacional» (Timberlake, 1993), sugiriendo que el activador traslacional es una forma de hacer la diferenciación sensible al estado nutricional de la hifa. Así, la hifa competente está preparada para emprender el desarrollo del conidióforo, pero la activación irreversible de la vía de la conidiación se evita mediante una represión traslacional que mantiene el crecimiento vegetativo hasta que las condiciones de esporulación son ideales.
La activación de brlA se considera, por tanto, el primer paso en el desarrollo del conidióforo, y su producto activa a su vez un panel de genes específicos de la conidiación entre los que se encuentra el siguiente regulador, abaA. El producto abaA es también una proteína reguladora transcripcional que se une al ADN y potencia la expresión de los genes estructurales inducidos por brlA. Los genes brlA y abaA son activadores recíprocos, porque abaA también activa a brlA. Por supuesto, la expresión de brlA debe producirse antes de que abaA pueda expresarse, pero la consiguiente activación de brlA por parte de abaA refuerza la expresión de este último y hace que el progreso de la vía sea independiente de los acontecimientos externos. El producto abaA también activa otros genes estructurales y el último gen regulador, wetA, que activa genes estructurales específicos de la espora. Dado que brlA y abaA no se expresan en las conidias en diferenciación, wetA está probablemente implicado en la inactivación de su expresión en las esporas (y quizás también en la fiálida; cuando el núcleo de la espora actual alcanza la fase de maduración que necesita los genes regulados por wetA, los núcleos de la fiálida deben «volver» al estado de iniciación del conidio para comenzar la formación de la siguiente espora). La expresión de wetA se activa inicialmente en la fiálida por acción secuencial de brlA y abaA, y luego se autorregula (el producto de wetA activa la transcripción de wetA). La autorregulación positiva de wetA mantiene posteriormente su expresión después de que el conidio se haya separado (física y citológicamente) de la fiálida.
Timberlake (1993) llamó a este mecanismo fijación por retroalimentación: la activación recíproca, la activación por retroalimentación y la autorregulación de las secuencias reguladoras centrales refuerzan la expresión de toda la vía, haciéndola independiente de las señales ambientales externas que la iniciaron y permitiendo que la espora continúe su maduración incluso después de la separación de la fiálida. El desarrollo del conidióforo, al igual que muchos otros procesos morfogenéticos, se divide naturalmente en pasos secuenciales. Esta red reguladora muestra cómo el desencadenamiento traslacional puede relacionar una vía morfogenética con el desarrollo de la competencia, por un lado, y con la iniciación en respuesta a la(s) señal(es) ambiental(es), por otro. Después de la iniciación, la fijación de la retroalimentación da lugar a la determinación del desarrollo en el sentido embriológico clásico de continuar la morfogénesis incluso cuando se retira del entorno de iniciación.
En esta sección hemos elegido deliberadamente basar nuestra descripción en la investigación realizada hace unos 50 años que comenzó con el aislamiento de un gran número de mutaciones con fenotipos de conidiación alterados y continuó con los estudios «clásicos» de segregación y complementación de genes (Martinelli & Clutterbuck, 1971). Además de mostrar cómo se ha logrado nuestra comprensión de la esporulación, demuestra el valor de utilizar estas técnicas «anticuadas» para investigar tales fenómenos.
Sin embargo, no podemos ignorar la extensa investigación que se ha completado sobre los mecanismos moleculares que subyacen al crecimiento y desarrollo de Aspergillus. Estos estudios han confirmado que el evento clave en la esporulación es la activación del factor de transcripción de dedo de zinc codificado por brlA, y que los genes abaA y wetA son reguladores necesarios de la conidiación. El factor de transcripción codificado por abaA es activado por brlA después de la diferenciación de las métulas y durante las etapas medias del desarrollo del conidióforo; y el genwetA, activado por abaA, funciona en la fase tardía de la conidiación dirigiendo la síntesis de componentes cruciales de la pared celular y transformando el metabolismo del conidio en maduración. En Aspergillus nidulans estos tres genes crean la vía reguladora central, que, con otros genes, controlan la expresión génica específica de la conidiación y la secuencia de activación génica implicada en la adquisición de la competencia de desarrollo, el desarrollo del conidióforo y la maduración de las esporas (Lee et al., 2016).
Muchos aspectos de estas vías de desarrollo se han conservado en otros aspergilos. Los miembros del género Aspergillus se encuentran entre los hongos más comunes, y todos se reproducen asexualmente formando largas cadenas de conidios. Varias especies, como Aspergillus oryzae y Aspergillus niger, se utilizan en la industria para la producción de enzimas y el procesamiento de alimentos, mientras que Aspergillus flavus es responsable del deterioro de los alimentos al producir los carcinógenos naturales más potentes conocidos, las aflatoxinas. Otra especie preocupante es el patógeno humano oportunista Aspergillus fumigatus, que produce un gran número de pequeñas conidias hidrofóbicas como principal medio de dispersión y se ha convertido en un patógeno fúngico de transmisión aérea muy extendido en los países desarrollados. En los pacientes inmunodeprimidos, A. fumigatus causa una aspergilosis invasiva que tiene una alta tasa de mortalidad. La vía de señalización del desarrollo BrlA-AbaA-WetA se ha conservado en la conidiación de estas especies de Aspergillus (Yu, 2010; Tao & Yu, 2011; Krijgsheld et al., 2013). Además, los estudios proteómicos, transcriptómicos y metabolómicos han proporcionado una imagen detallada de los cambios dinámicos que se producen en muchos miles de genes, transcritos, enzimas y reacciones metabólicas durante la formación, maduración, latencia y germinación de los conidios de Aspergillus (van Leeuwen et al., 2013; Novodvorska et al., 2016; Teertstra et al., 2017).
Actualizado en julio de 2019