La cellula da cui emerge il ramo è la cellula del piede del conidioforo, che si distingue dalle altre cellule vegetative per avere un ispessimento della parete secondaria pigmentata marrone all’interno della sua parete originale. Il peduncolo cresce apicalmente fino a raggiungere una lunghezza di circa 100 µm quando l’apice si gonfia per formare la vescicola conidioforica che ha un diametro di circa 10 µm. Un singolo livello di numerosi sterigmi primari, chiamati metule, germoglia poi dalla vescicola e gli sterigmi secondari, le fialidi, germogliano dagli apici esposti delle metule. Le fialidi sono le cellule staminali che successivamente subiscono ripetute divisioni asimmetriche per formare le lunghe catene di conidi enteroblastici che hanno un diametro di circa 3 µm (Fig. 7).
L’analisi genetica classica, attraverso l’isolamento e l’analisi di mutanti, ha stabilito lo schema genetico di base della conidiazione dell’Aspergillus. Tra 300 e 1.000 loci genici sono stati stimati essere interessati alla conidazione confrontando le frequenze di mutazione nei loci che influenzano la conidazione con quelle per altre funzioni (Martinelli & Clutterbuck, 1971). L’analisi delle specie di mRNA ha indicato che circa 6.000 sono stati espressi nel micelio vegetativo e altri 1.200 sono stati trovati in culture che includevano conidiofori e conidi; 200 di questi mRNA aggiuntivi sono stati trovati nei conidi stessi. Solo circa il 2% dei mutanti di A. nidulans che mancavano di conidi avevano difetti nelle fasi relative alla crescita e allo sviluppo dei conidiofori, e l’85% dei mutanti di conidiofilia erano anche difettosi nella crescita degli ifali vegetativi e nel raggiungimento della competenza.
Due geni, in particolare, giocano un ruolo chiave nella morfogenesi dei conidiofori: si tratta del gene ‘bristle’ (brlA) che presenta difetti nella formazione di vescicole e metule, e ‘abacus’ (abaA) in cui i conidi sono sostituiti da lunghezze di perline di ifa, quindi è presumibilmente difettoso nella gemmazione dei conidi dalla fialide e nella settatura finale. Un terzo gene, wetA, è difettoso in una fase iniziale della maturazione delle spore. I conidi dei mutanti wetA mancano di pigmento e idrofobicità; autolisi dopo poche ore e non riescono ad esprimere una serie di mRNA specifici delle spore. Il trascritto del gene wetA manca nei mutanti brlA e abaA (cioè brlA e abaA sono epistatici a wetA), e gli studi sui mutanti doppi mostrano che questi tre geni agiscono nell’ordine: brlA → abaA → wetA.
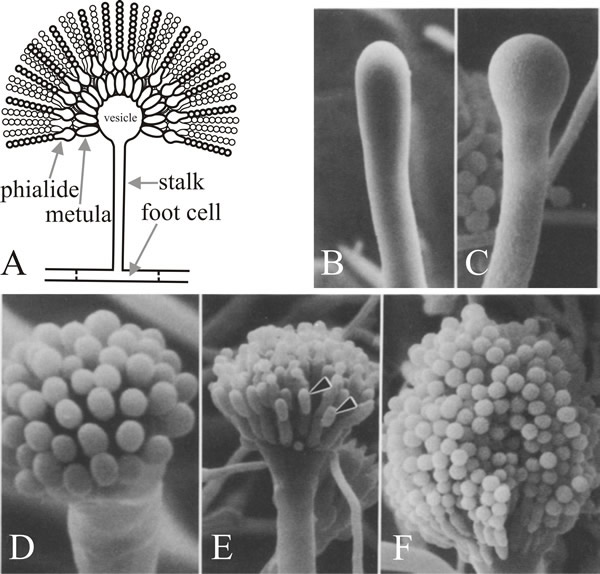
Una caratteristica sorprendente dell’analisi mutazionale dello sviluppo dei conidiofori in A. nidulans è che i mutanti di solo questi tre geni causano difetti nella morfogenesi dei conidiofori e delle spore, mentre fino a 1.000 altri loci genici causano assenza di conidiazione quando mutati. Questo implica che brlA, abaA e wetA sono regolatori che integrano l’espressione di altri geni che sono necessari per la conidazione ma non sono essi stessi dedicati ad essa. Molti dei mutanti di conidazione di Aspergillus erano anche difettosi nella riproduzione sessuale, quindi un’altra conclusione da trarre è che c’è una certa economia di utilizzo dei geni morfogenetici nei diversi processi di sviluppo. Presumibilmente, i diversi modi di sviluppo impiegano geni strutturali che non sono unicamente sviluppativi, ma funzionano in numerosi percorsi, avendo la loro specificità di sviluppo conferita loro dai regolatori a cui rispondono. Questo è incarnato dall’idea che la chiave dello sviluppo degli eucarioti è nella capacità di usare relativamente pochi geni regolatori per integrare le attività di molti altri.
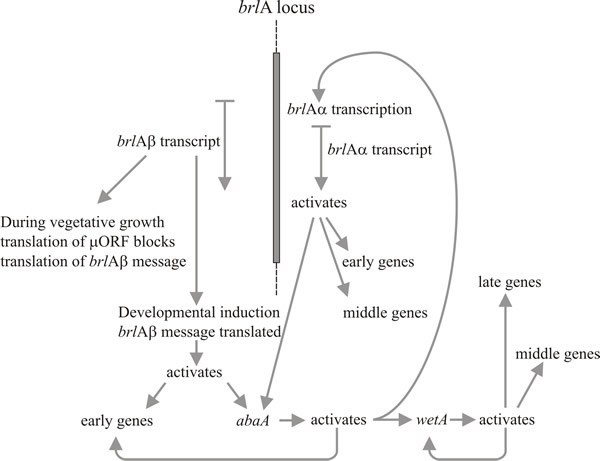
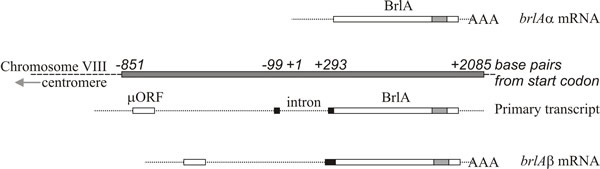
L’analisi molecolare supporta l’interpretazione che brlA, abaA e wetA sono regolatori; la sequenza brlA codifica una proteina zinc finger, che è un attivatore di trascrizione con legame al DNA specifico per la sequenza di geni target regolati dallo sviluppo (Fig. 8). Tuttavia, questa non è tutta la storia, perché il prodotto brlA ha diverse affinità per diversi geni bersaglio. Infatti, il locus brlA consiste di unità di trascrizione sovrapposte (Fig. 9), l’unità a valle è designata brlAα e l’unità a monte brlAβ; tra loro, i loro prodotti risolvono due problemi classici dello sviluppo, come rispondere a un segnale e come mantenere quella risposta quando il segnale si è dissipato.
Le due unità di trascrizione di brlA condividono la stessa struttura di lettura per la maggior parte della loro lunghezza, ma brlAβ ha 23 residui aminoacidici aggiuntivi all’estremità amino-terminale di quella struttura di lettura, e il suo trascritto possiede anche una struttura di lettura separata di 41 residui aminoacidici (chiamata µORF) vicino al suo termine 5′. Il prodotto µORF reprime la traduzione dal frame di lettura a valle (BrlA) e anche se il trascritto brlAβ può essere rilevato nelle ife vegetative, il peptide BrlA non viene prodotto. La repressione causata da µORF è superata solo quando il micelio è competente, il che è probabilmente segnalato da una limitazione di azoto (un segnale ambientale comune per l’inizio della sporulazione negli Ascomycota), che riduce i pool di aminoacil-tRNA e disturba la regolazione traslazionale da parte di µORF. Quando l’effetto di µORF è bloccato, il peptide BrlA può essere tradotto dal trascritto esistente.
Lo scopritore di questo processo ha descritto l’attivazione della via di conidazione come “innesco traslazionale” (Timberlake, 1993), suggerendo che l’innesco traslazionale è un modo di rendere sensibile il differenziamento allo stato nutrizionale dell’ifa. Così l’ifa competente è pronta a intraprendere lo sviluppo del conidioforo, ma l’attivazione irreversibile della via di conidiazione è impedita da una repressione traslazionale che mantiene la crescita vegetativa fino a quando le condizioni di sporulazione sono ideali.
L’attivazione di brlA è, quindi, vista come il primo passo nello sviluppo del conidioforo, e il suo prodotto a sua volta attiva un pannello di geni specifici per la conidiazione, tra cui il regolatore successivo, abaA. Il prodotto di abaA è anche una proteina regolatrice trascrizionale legata al DNA che aumenta l’espressione dei geni strutturali indotti da brlA. I geni brlA e abaA sono attivatori reciproci, perché abaA attiva anche brlA. Naturalmente l’espressione di brlA deve avvenire prima che abaA possa essere espressa, ma la conseguente attivazione di brlA da parte di abaA rafforza l’espressione di quest’ultimo e rende effettivamente il progresso della via indipendente da eventi esterni. Il prodotto di abaA attiva anche altri geni strutturali e il gene regolatore finale, wetA, che attiva i geni strutturali specifici della spora. Poiché brlA e abaA non sono espressi nei conidi in fase di differenziazione, wetA è probabilmente coinvolto nella disattivazione della loro espressione nelle spore (e forse anche nella fialide; quando il nucleo della spora corrente raggiunge lo stadio di maturazione che necessita dei geni regolati da wetA, i nuclei della fialide devono essere “girati indietro” allo stato di inizio del conidio per iniziare la formazione della spora successiva). L’espressione di wetA è inizialmente attivata nella fialide dall’azione sequenziale di brlA e abaA, ed è poi autoregolata (il prodotto wetA attiva la trascrizione di wetA). L’autoregolazione positiva di wetA mantiene successivamente la sua espressione dopo che il conidio è stato separato (fisicamente e citologicamente) dalla fialide.
Timberlake (1993) ha chiamato questo meccanismo fissazione a feedback: l’attivazione reciproca, l’attivazione a feedback e l’autoregolazione delle sequenze di regolazione centrale rafforzano l’espressione dell’intero percorso, rendendolo indipendente dagli spunti ambientali esterni che lo hanno avviato e permettendo alla spora di continuare la maturazione anche dopo la separazione dalla fialide. Lo sviluppo del conidioforo, come molti altri processi morfogenetici, è naturalmente diviso in fasi sequenziali. Questa rete di regolazione mostra come l’innesco traslazionale può mettere in relazione un percorso morfogenetico con lo sviluppo della competenza da un lato, e l’iniziazione in risposta a uno o più indizi ambientali dall’altro. Successivamente all’iniziazione, la fissazione del feedback ha come risultato la determinazione dello sviluppo nel classico senso embriologico di continuare la morfogenesi anche quando viene rimossa dall’ambiente di iniziazione.
In questa sezione abbiamo scelto deliberatamente di basare la nostra descrizione su ricerche fatte circa 50 anni fa che sono iniziate con l’isolamento di un gran numero di mutazioni con fenotipi di conidazione alterati e sono continuate con studi “classici” di segregazione e complementazione genica (Martinelli & Clutterbuck, 1971). Oltre a mostrare come la nostra comprensione della sporulazione è stata compiuta, dimostra il valore dell’uso di queste tecniche ‘antiquate’ per indagare tali fenomeni.
Tuttavia, non possiamo ignorare la vasta ricerca che è stata completata sui meccanismi molecolari alla base della crescita e dello sviluppo di Aspergillus. Questi studi hanno confermato che l’evento chiave nella sporulazione è l’attivazione del fattore di trascrizione zinc finger codificato da brlA, e che i geni abaA e wetA sono regolatori necessari della conidazione. Il fattore di trascrizione codificato da brlA è attivato da brlA dopo la differenziazione delle metule e durante le fasi centrali dello sviluppo del conidioforo; e il gene wetA, attivato da abaA, funziona nella fase tardiva della conidiazione dirigendo la sintesi di componenti cruciali della parete cellulare e trasformando il metabolismo del conidio in maturazione. In Aspergillus nidulans questi tre geni creano la via di regolazione centrale, che, con altri geni, controllano l’espressione genica specifica della conidazione e la sequenza di attivazione genica coinvolta nell’acquisizione della competenza di sviluppo, lo sviluppo del conidioforo e la maturazione della spora (Lee et al., 2016).
Molti aspetti di queste vie di sviluppo sono stati conservati in altri aspergilli. I membri del genere Aspergillus sono tra i funghi più comunemente incontrati, e tutti si riproducono asessualmente formando lunghe catene di conidi. Diverse specie, tra cui Aspergillus oryzae e Aspergillus niger, sono usate nell’industria per la produzione di enzimi e la lavorazione degli alimenti; mentre Aspergillus flavus è responsabile del deterioramento degli alimenti producendo le più potenti sostanze cancerogene conosciute in natura, le aflatossine. Un’altra specie che desta preoccupazione è il patogeno umano opportunista Aspergillus fumigatus, che produce un numero massiccio di piccoli conidi idrofobici come mezzo primario di dispersione ed è diventato un patogeno fungino diffuso nell’aria nei paesi sviluppati. Nei pazienti immunocompromessi, A. fumigatus causa un’aspergillosi invasiva che ha un alto tasso di mortalità. La via di segnalazione dello sviluppo BrlA-AbaA-WetA è stata conservata nella conidazione di queste specie di Aspergillus (Yu, 2010; Tao & Yu, 2011; Krijgsheld et al., 2013). Inoltre, gli studi di proteomica, trascrittomica e metabolomica hanno fornito un quadro dettagliato dei cambiamenti dinamici che avvengono in molte migliaia di geni, trascrizioni, enzimi e reazioni metaboliche durante la formazione, maturazione, dormienza e germinazione dei conidi di Aspergillus (van Leeuwen et al., 2013; Novodvorska et al., 2016; Teertstra et al., 2017).
Aggiornato a luglio 2019