Introduzione
Il metano (CH4) è un importante gas serra. La sua emissione da ambienti naturali come le zone umide, gli oceani e i sedimenti rappresenta oltre il 70% del metano atmosferico a livello globale (IPCC, 2007). Una valutazione dei dati pubblicati ha rivelato diversi tassi di emissione di metano per le zone umide, i laghi, i fiumi, gli estuari e gli oceani (in ordine decrescente, Figura supplementare S1). Le zone umide naturali rappresentano da sole il 62% della produzione biogenica di CH4 (Kirschke et al., 2013; Nazaries et al., 2013) e le emissioni delle zone umide dominano la variabilità interannuale delle fonti di metano (Bousquet et al., 2006). Al contrario, la vasta area degli ecosistemi marini contribuisce solo per l’8% circa alle fonti naturali di CH4 (Nazaries et al., 2013). All’interno dell’emissione globale di metano oceanico, meno del 10% è contribuito dagli estuari (Bange et al., 1994).
La metanogenesi, la formazione biologica di metano, è effettuata da archei metanogeni che producono metano principalmente da H2/CO2, gruppi metilici o acetato in condizioni anossiche (Thauer et al., 2008). Questa reazione è catalizzata dalla metil-coenzima M reduttasi (MCR). Il gene mcrA che codifica una subunità di questo enzima è un gene marcatore comunemente usato nelle indagini molecolari (Conrad, 2007; Bridgham et al., 2013). Il vantaggio del marcatore del gene mcrA è quello di catturare sia le firme filogenetiche che funzionali dei metanogeni, offrendo un’elevata profondità di sequenziamento per questa particolare funzione (Luton et al., 2002; Borrel et al., 2013; Yang et al., 2014). Un gran numero di sequenze mcrA sono state recuperate da una varietà di ambienti naturali. Il set di dati pubblici mcrA permette di estrarre modelli ecologici generali e di indagare i gradienti ambientali che formano a scala globale e regionale. Inoltre, è disponibile un database che riassume le proprietà fisiologiche di 152 isolati metanogenici1 (Jabłoński et al., 2015). Recentemente, il binning del genoma ha rivelato sequenze mcrA insolite nella nuova classe di Methanofastidiosa (Nobu et al., 2016) e i nuovi phyla di Bathyarchaeota (Evans et al., 2015) e Verstraetearchaeota (Vanwonterghem et al., 2016). Questi nuovi risultati hanno ampliato le nostre conoscenze sulla diversità dei potenziali metanogeni, ma non hanno oscurato l’applicabilità del gene mcrA come marcatore molecolare per la grande maggioranza delle comunità metanogeniche.
Ad oggi, le comunità metanogeniche sono state rilevate in zone umide, sedimenti, aree permafrost, risaie, digestori, sorgenti geotermiche e bocche idrotermali (Conrad, 2007; Thauer et al., 2008; Wagner e Liebner, 2009). La struttura della comunità metanigena è stata trovata associata al pH ambientale, alla temperatura, alla salinità, al livello delle acque sotterranee e alle dinamiche della vegetazione a diverse scale spaziali e temporali (Megonigal et al., 2005; Milferstedt et al., 2010; Frank-Fahle et al., 2014; McCalley et al., 2014; Cui et al., 2015; Liebner et al., 2015). Ad esempio, la metanogenesi acetoclastica è generalmente ostacolata da un basso pH, poiché riduce la dissociazione dell’acetato (Megonigal et al., 2005; Kotsyurbenko et al., 2007). La vegetazione può fornire carbonio organico labile e di alta qualità per alimentare i metanogeni sotto forma di essudati radicali o detriti, così che gli essudati vegetali generalmente favoriscono i metanogeni acetoclastici principalmente nelle torbiere (Bridgham et al., 2013). Il solfato dall’acqua di mare inibisce la produzione di metano nelle zone umide di marea, e la salinità è stata di conseguenza utilizzata come un predittore generale per le emissioni di metano (Holm et al., 2016). Uno studio sui sedimenti dei laghi tibetani ha mostrato che l’aumento della salinità inibisce i metanogeni idrogenotrofi ma aumenta la metanogenesi acetoclastica (Liu et al., 2016). Questi studi hanno indicato i driver ambientali per le comunità metanogeniche, ma si sono concentrati su singoli habitat o su scale spaziali limitate.
Comprendere l’adattamento dei metanogeni ai diversi cambiamenti ambientali, tuttavia, richiede un’esplorazione sistematica e globale delle correlazioni tra la composizione della comunità microbica e le condizioni ambientali (Lozupone e Knight, 2007). Al momento, solo pochi studi affrontano la dispersione e il filtraggio dell’habitat delle comunità metanigene (Auguet et al., 2010; Barreto et al., 2014). Noi ipotizziamo che gli assemblaggi metanogenici siano principalmente influenzati dal filtraggio dell’habitat e che questo sia guidato da controlli ambientali globali. Considerando che i tassi di emissione di metano differiscono ampiamente tra gli ecosistemi naturali, l’integrazione esplicita della composizione, diversità e biogeografia degli assemblaggi metanogenici in questi ecosistemi può essere fondamentale per determinare la risposta della produzione di metano ai cambiamenti climatici attuali e futuri. Questo metastudio viene eseguito per colmare le lacune associate alla biogeografia metanogenica, alla diversità e ai suoi controlli ambientali utilizzando i dati di sequenza mcrA pubblicamente disponibili e la letteratura integrata da dati fisiologici di isolati metanogenici.
Materiali e metodi
Raccolta dati
Abbiamo recuperato le sequenze mcrA disponibili in GenBank (gennaio 2016)2. Per ogni hit, la carta originale è stato controllato e le sequenze mcrA secondo sono stati analizzati da uno script Perl personalizzato. Poiché ci siamo concentrati sugli ambienti naturali, le sequenze mcrA metanogeniche sono state ottenute da habitat naturali e classificate come suolo, lago, estuario, sedimenti marini e idrotermali e vulcani di fango. Cinque librerie di sequenziamento di nuova generazione (NGS) sono state incluse oltre alle sequenze delle librerie di cloni. Le sequenze sono state scaricate senza tener conto dell’abbondanza relativa nel dataset originale. Poiché le sequenze delle biblioteche di cloni coprono principalmente i fenotipi abbondanti mentre NGS può catturare una diversità molto più profonda, abbiamo fatto un compromesso per utilizzare i dati NGS ma mitigato un potenziale errore dovuto alla diversa risoluzione dei metodi di sequenziamento. Pertanto, abbiamo scelto solo le sequenze rappresentative di OTU abbondanti con un’abbondanza relativa superiore all’1%. Abbiamo inoltre scartato quelle sequenze NGS che non hanno superato il controllo di traduzione da nucleotide a sequenze proteiche o con una bassa qualità (sequenze < 250 bps). Infine, abbiamo costruito un set di dati contenente 4466 sequenze uniche di mcrA da 94 siti distribuiti globalmente (Figura 1 e tabelle supplementari S1, S2). Inoltre, non abbiamo sottratto le sequenze mcrA di potenziali metanotrofi arcaici dal dataset, che è stato inevitabilmente rilevato nell’indagine genomica (Conrad, 2007). Questa parte esula dal focus di questo studio.
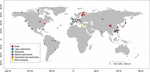
FIGURA 1. Ubicazione dei siti utilizzati per questo studio. I siti sono stati raggruppati in sei diverse categorie secondo i loro habitat e sono stati etichettati con colori diversi. Un riassunto dei sei habitat è riportato nelle tabelle supplementari S1, S2.
Le coordinate geografiche e le impostazioni ambientali tra cui pH, salinità, elevazione, temperatura media annuale dell’aria (MAAT) e precipitazione media annuale (MAP) sono stati estratti per ogni sito di ricerca considerato in questo studio dalle pubblicazioni corrispondenti, dato che i dati sono disponibili (vedi tabella supplementare S1). Per tenere conto dei parametri ambientali mancanti in più studi, abbiamo definito qualitativamente alcune variabili ambientali in base alle descrizioni del sito nella letteratura pertinente, e poi abbiamo convertito questi dati di categoria in valori numerici semi-metrici, per esempio, abbiamo definito i sedimenti marini, i sedimenti idrotermali, il fango vulcanico e i campioni di sedimenti di lago di soda come “salini”, il suolo e i campioni di sedimenti di lago di acqua dolce come “non salini”, e i campioni di mangrovie ed estuario come campioni “misti”.
L’elaborazione della sequenza grezza
L’elaborazione della sequenza è stata implementata con la piattaforma software Mothur (Schloss et al, 2009). Le sequenze provenienti da diverse biblioteche sono state raggruppate prima dell’elaborazione. Le sequenze con una lunghezza inferiore a 350 bp o più di 8 basi ambigue sono state scartate. Successivamente, queste sequenze sono state allineate contro un sottoinsieme pre-allineato di sequenze mcrA, che sono stati recuperati dal database FunGene a http://fungene.cme.msu.edu/ (Fish et al., 2013). Le sequenze chimeriche sono state identificate con il software Mothur utilizzando il metodo uchime (Edgar et al., 2011) con il dataset stesso come riferimento. Poi, le sequenze nucleotidiche valide del gene mcrA sono state utilizzate per calcolare le distanze a coppie non corrette tra le sequenze di DNA allineate e ulteriormente assegnate in unità tassonomiche operative (OTU) con un cutoff dell’84% che corrisponde al 97% per il gene 16S rRNA (Yang et al., 2014). L’abbondanza di ogni OTU mcrA è stata contabilizzata solo come presenza o assenza. Abbiamo aumentato l’accuratezza della classificazione tassonomica delle OTU considerando sia le sequenze nucleotidiche che quelle di aminoacidi. A livello di DNA, l’identità tassonomica è stata assegnata dalla piattaforma Mothur secondo un database di riferimento (Yang et al., 2014). A livello di proteine, le sequenze proteiche allineate sono state utilizzate per costruire un albero in ARB, e poi l’assegnazione tassonomica è stata basata sul database corrispondente. Se l’assegnazione di un OTU era incoerente, abbiamo fatto manualmente un blast sia del nucleotide che della sequenza proteica in NCBI e determinato l’identità tassonomica finale prendendo in considerazione la copertura della query (>95%), l’identità (>84%), e il valore e- (<1E-5). Per le sequenze proteiche, il cutoff a livello di genere ha fatto riferimento alla soglia dell’83,5% (Hunger et al., 2011).
Analisi ecologica e statistica
L’analisi statistica è stata fatta da vari pacchetti R. Le ordinazioni dell’analisi delle coordinate principali (PCoA) sono state generate sulla base delle matrici di distanza Jaccard costruite utilizzando il pacchetto vegan v2.2.0 (Oksanen et al., 2015). Permutazionale MANOVA (analisi multivariata della varianza) è stata condotta per valutare la fonte di variazione nella matrice Jaccard (McArdle e Anderson, 2001) in vegan con 104 permutazioni. Le misure di distanza di Jaccard si basano sulla presenza/assenza delle specie, che è più adatto per il nostro set di dati in quanto la maggior parte degli studi ha fornito solo le sequenze rappresentative mentre le informazioni sulle abbondanze sono mancanti. Le frequenze di incidenza tassonomica attraverso gli habitat sono state visualizzate attraverso grafici a bolle con il pacchetto ggplot2 (v1.0.0) (Wickham, 2009). L’analisi di clustering gerarchico delle comunità del suolo non salino e dei sedimenti lacustri è stata eseguita con la funzione R ‘hclust’ (R Core Team, 2014). I cluster di comunità ottenuti sono stati descritti in base al regime di pH e temperatura dei campioni originali perché utilizzando PCoA in precedenza abbiamo identificato entrambi i parametri pH e temperatura per influenzare la composizione delle comunità metanogeniche nei suoli non salini e nei sedimenti lacustri. L’associazione di ogni stirpe metanigena con ciascuno di questi cluster è stata determinata utilizzando l’analisi delle specie indicatrici basata sulla correlazione (Dufrene e Legendre, 1997). Le specie indicatrici sono qui definite come quelle che sono sia abbondanti in un tipo specifico di habitat (specificità) sia che si trovano prevalentemente in questo tipo di habitat (fedeltà). In questo studio, i taxa indicatori, simili al concetto di specie indicatrici, per i suoli non salini e i sedimenti lacustri sono stati scelti in base a un valore indicatore (valore IndVal) dal pacchetto R labdsv (Roberts, 2016) se la probabilità di ottenere un valore indicatore alto come quello osservato nelle iterazioni specificate è inferiore a 0,05. Gli indici Chao2 sono stati calcolati per ogni campione utilizzando il pacchetto vegan. Il test Wilcoxon rank sum degli indici Chao2 tra gli habitat è stato eseguito dalla funzione R ‘wilcox.test’ (R Core Team, 2014). Per consolidare l’impatto del filtraggio dell’habitat sugli isolati di archaea metanogenici, le caratteristiche fisiologiche e biochimiche delle culture metanogeniche descritte sono state recuperate dal3 ‘Methanogenic archaea database’ (Jabłoński et al., 2015). Tra questi, gli isolati con informazioni sul requisito ottimale di NaCl sono stati filtrati, categorizzati e tracciati secondo la loro fonte di isolamento.
Per esaminare l’influenza della limitazione della dispersione sulla struttura della comunità metanigena, è stata eseguita un’analisi di regressione lineare basata su una matrice di distanza geografica e sulla matrice di distanza Jaccard della comunità mediante la funzione R ‘lm’ (R Core Team, 2014). Abbiamo eseguito i test di Mantel e Mantel parziale per valutare gli effetti della limitazione della dispersione in base alle due matrici utilizzando nuovamente il pacchetto vegan in R (Oksanen et al., 2015). Inoltre, l’analisi spaziale multivariata (PCA spaziale) è stata applicata a 16 campioni europei di suolo e sedimenti lacustri sulla base dell’indice I di Moran per esplorare la struttura spaziale dei metanogeni tramite la funzione “multispati” nel pacchetto R ade4 (Dray e Dufour, 2007). Inoltre, il clustering Ward’s Minimum variance basato sulla matrice di distanza Jaccard è stato implementato su questi 16 campioni utilizzando la funzione R “hclust” (R Core Team, 2014) e abbiamo ulteriormente proiettato i risultati del clustering su una mappa geografica. Lo shapefile europeo per la mappatura a livello statale è disponibile presso il database GSHHG (v2.3.6)4. La mappa è stata generata utilizzando QGIS v2.18.25.
Risultati
Biogeografia degli archei metanogeni in ambienti naturali
Sono state recuperate le sequenze del gene mcrA da 94 ambienti naturali distribuiti globalmente. L’ubicazione e il tipo di ecosistema di ognuno di questi 94 siti sono rappresentati nella Figura 1. Le frequenze di incidenza (presenza/assenza) dei lignaggi metanogeni sono state unite secondo il tipo di ecosistema e illustrate nella Figura 2. In breve, Methanoregula è il taxon più frequentemente osservato nei suoli, insieme a Methanobacterium, Methanosaeta, Methanocella, Methanomassiliicoccus e Methanosarcina. Nei sedimenti dell’estuario, sono state comunemente rilevate sequenze di Methanosaeta, Methanobacterium, Methanoregula e Methanoculleus. Inoltre, nei sedimenti lacustri, si sono verificati principalmente Methanoregula e Methanosaeta. Nei sedimenti marini, Methanoculleus e Methanosaeta sono i lignaggi più comuni, seguiti da Methanolinea.
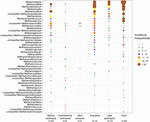
FIGURA 2. Grafico a bolle che mostra le frequenze di incidenza dei lignaggi metanogeni in diversi ambienti naturali. L’ordine di rango lungo l’asse verticale corrisponde alle frequenze di incidenza totale decrescenti dei lignaggi. La tassonomia è mostrata per il livello di genere. Se un’assegnazione al livello di genere non è stata possibile, è stato utilizzato il livello tassonomico superiore assegnabile. Il numero di campioni (n) è dato per ogni habitat.
Anche se molti taxa sono stati rilevati in diversi ambienti, alcuni mostrano ancora preferenze ambientali. Methanoregula, il taxon frequentemente presente in ambienti non marini e di transizione (suoli, sedimenti lacustri ed estuari), è assente dagli habitat marini (sedimenti marini, sedimenti idrotermali e vulcani di fango). Methanobacterium e Methanocella, che prevalgono negli ambienti non marini e di transizione, si trovano raramente negli habitat marini. Al contrario, Methanococcoides, come stirpe predominante nei sedimenti marini, nei sedimenti idrotermali e nei vulcani di fango, è appena osservato nei suoli e nei sedimenti lacustri. Inoltre, Methanogenium e Methanolacinia sono osservati solo nei sedimenti di estuario e marini, mentre Methanospirillum e Methanosphaerula si trovano solo in ambienti terrestri. Inoltre, alcuni taxa specifici si trovano esclusivamente nei sedimenti idrotermali, tra cui Methanocaldococcus, Methanothermococcus, Methanopyrus, Methanotorris, e Methanococcus. Anche se alcuni lignaggi come Methanosaeta sono presenti nella maggior parte degli ambienti, nessun lignaggio può essere considerato onnipresente.
La più alta ricchezza di lignaggi si è verificata nei sedimenti di estuario che ospitano anche frequenze di incidenza più uniformi di vari lignaggi. Al contrario, i vulcani di fango e gli ecosistemi idrotermali mostrano una diversità metanogenica relativamente bassa. I suoli e i sedimenti lacustri simili agli estuari sono caratterizzati da diversi assemblaggi metanogenici.
Alpha-Diversity of Methanogenic Communities in Natural Environments
La ricchezza degli archei metanogenici secondo l’indice Chao2 varia ampiamente tra i tipi di ecosistemi (Figura 3). Allo scopo di confrontare direttamente le diversità alfa e per ottenere un ragionevole compromesso tra i campioni, è stato effettuato un sottocampionamento a 15 sequenze per ogni sito. L’indice Chao2 mostra che i sedimenti dell’estuario comprendono la più alta ricchezza di specie di metanogeni lungo i sei tipi di ecosistema (Tabella supplementare S3), che sottolinea i risultati del bubble plot (Figura 2). I suoli e i sedimenti lacustri, che mostrano una ricchezza inferiore rispetto ai campioni di estuario, hanno indici Chao2 significativamente più alti rispetto ai sedimenti marini e idrotermali e senza differenze significative tra i sedimenti marini e quelli idrotermali (Tabella supplementare S3).
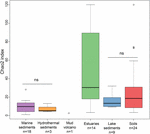
FIGURA 3. Box plot degli indici Chao2 dei diversi tipi di ecosistema. Il grafico si basa su set di dati sottocampionati contenenti 15 sequenze per ogni sito per rendere più robusto il confronto sulle misure di diversità alfa. Il numero di campioni in ogni habitat è dato come ‘n’ sotto l’etichetta dell’habitat. Il “ns” non dona significatività statistica nel test di Wilcoxon. Il risultato statistico della diversità alfa a livello di OTU è riportato nella Tabella supplementare S3.
Controlli globali sulle comunità metanogeniche in ambienti naturali
Le 94 comunità metanogeniche distribuite globalmente sono state raggruppate in un grafico di ordinamento applicando PCoA basato sulla matrice di distanza Jaccard. Secondo l’analisi PCoA, il primo e il secondo asse spiegano insieme il 16,3% della varianza totale. Le variazioni tra i campioni possono quindi essere ampiamente spiegate dalla salinità (Figura 4). Poiché i dati iniziali sulle concentrazioni di sale non erano disponibili in alcuni casi, abbiamo assegnato qualitativamente questi campioni come campioni salini, misti (intermedi) e non salini come descritto sopra. I campioni salini e non salini si separano effettivamente lungo il primo asse. I campioni misti si collocano complessivamente tra i campioni salini e non salini. La MANOVA permutazionale basata sulla matrice di distanza Jaccard suggerisce anche che la salinità è il fattore abiotico primario che controlla la distribuzione delle comunità metanogeniche globali (R2 = 0,099, P < 0,001) (Tabella 1).
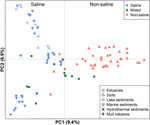
FIGURA 4. Ordinazione dell’analisi delle coordinate principali (PCoA) basata sulla matrice di distanza Jaccard di librerie di sequenze di geni mcrA metanogenici, confrontando 94 campioni. La PCoA è colorata in base alla salinità: I simboli rossi indicano ambienti non salini, quelli blu indicano ambienti salini, e quelli verdi indicano ambienti intermedi. Simboli diversi rappresentano ambienti diversi. La percentuale della variazione spiegata dalle coordinate principali tracciate è indicata sugli assi.

TABELLA 1. Analisi MANOVA permutazionale sulla matrice di distanza Jaccard di tutti i campioni dai sei habitat per testare l’associazione della varianza della comunità con diverse variabili ambientali.
Inoltre, abbiamo controllato una potenziale relazione tra la fonte di isolamento delle colture pure metanogeniche e la concentrazione ottimale di NaCl per la crescita. La concentrazione ottimale di NaCl delle colture pure metanogeniche ha dimostrato un declino dagli ecosistemi marini agli estuari, ai sedimenti lacustri e al suolo (Figura supplementare S2). Alcuni isolati anomali provengono da sedimenti di laghi sodi o suoli ipersalini.
Controlli ambientali e taxa indicatori metanogenici in suoli non salini e sedimenti di lago
Su scala globale, le comunità metanogeniche da suoli non salini e sedimenti di lago si raggruppano strettamente (Figura 4), così che abbiamo ulteriormente analizzato i controlli ambientali delle comunità metanogeniche di questi due habitat che rappresentano 33 siti di studio in totale. L’analisi dei cluster basata sulla comunità per questi due tipi di habitat ha rivelato quattro cluster basati sulla matrice di distanza Jaccard (Figura supplementare S3). La MANOVA permutazionale suggerisce che sia il pH (R2 = 0,099, P < 0,001) che la temperatura (R2 = 0,069, P < 0,001) influenzano la β-diversità metanogenica nei suoli non salini e nei sedimenti del lago (Tabella 2). Di conseguenza, abbiamo assegnato i quattro cluster al pH e al MAAT del sito di campionamento iniziale e abbiamo ottenuto sottogruppi ampiamente coerenti al clustering della comunità (Figure 5C,D). La combinazione delle caratteristiche ambientali e di questi quattro cluster di comunità ci permette di definire questi quattro sottogruppi come neutro e freddo gruppo 1, acido e freddo gruppo 2, acido e moderato gruppo 3, e neutro e caldo gruppo 4 (Figura 5). Un’ulteriore ordinamento PCoA basato sulla matrice di dissimilarità Jaccard suggerisce che lungo l’asse PC1, la maggior parte dei campioni del gruppo 2 e del gruppo 3 provengono da suoli acidi e sedimenti lacustri mentre il gruppo 1 e il gruppo 4 provengono principalmente da ambienti neutri (Figura 5A). Inoltre, i campioni dei siti moderati (gruppo 3) si sono separati da quelli dei siti caldi e freddi lungo PC2, mentre i campioni degli ambienti caldi (gruppo 4) si sono separati dagli altri campioni lungo PC3 (Figura 5B). In questo modo i primi tre assi dell’ordinamento PCoA spiegano il 38,8% della variazione totale.

TABELLA 2. MANOVA permutazionale basata su una matrice di distanza Jaccard di campioni di suolo non salino e sedimenti lacustri per testare l’associazione della varianza di comunità con diverse variabili ambientali.
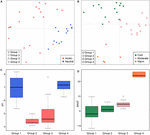
FIGURA 5. Trama dell’analisi delle coordinate principali basata su una matrice di distanza Jaccard per 33 suoli non salini e sedimenti lacustri. La sottotrama (A) mostra PC1 e PC2 e i simboli sono colorati per il pH, e la sottotrama (B) mostra PC2 e PC3 e i simboli sono colorati per la temperatura. Le prime tre componenti spiegano 17,1, 11,7 e 10% della varianza. I box plot mostrano il pH (C) e la MAAT (temperatura media annuale dell’aria) (D) di quattro sottogruppi identificati. Il colore del riquadro nella figura C corrisponde alla categoria di pH in (A). I colori dei boxplot mostrano la significatività statistica basata su un test Wilcoxon a coppie (P < 0,05), dove i campioni con lo stesso colore non differiscono significativamente l’uno dall’altro. Allo stesso modo, il colore in (D) segue il raggruppamento della temperatura in (B). I sottogruppi si riferiscono all’analisi gerarchica dei cluster delle somiglianze di comunità.
Abbiamo esaminato la presenza di lignaggi metanogeni in ogni sottogruppo in base alle frequenze di incidenza (Figura 6). Methanoregula prevale in tutti i tipi di habitat non salini. Oltre a Methanoregula, il sottogruppo neutro e freddo (gruppo 1) mostra un’alta abbondanza di Methanosaeta, Methanobacterium e Methanosarcina. Il gruppo 2, acido e freddo, è rappresentato da Methanobacterium, Methanocella e Methanosarcina, mentre Methanosaeta è assente. Methanocella e Methanosaeta sono comuni nel gruppo acido e moderato 3. Nel gruppo neutro e caldo 4, Methanolinea e Methanosaeta sono membri importanti. Questo gruppo è l’unico in cui è stato identificato Methanoculleus.

FIGURA 6. Frequenze di incidenza dei lignaggi metanogenici all’interno dei quattro gruppi definiti di comunità metanogeniche in suoli e sedimenti lacustri. L’asse verticale è disposto in ordine alfabetico. La lunghezza della barra corrisponde alla media delle frequenze di incidenza per ogni lignaggio all’interno del gruppo corrispondente. Le barre di errore rappresentano la deviazione standard di un dato taxon su diversi campioni di quel gruppo. Gli asterischi mostrano i taxa specializzati con P-value < 0,05. La tassonomia è mostrata per il livello di genere. Se un’assegnazione al livello di genere non era possibile, è stato utilizzato il livello tassonomico superiore assegnabile. La descrizione per i quattro gruppi è riportata nella Figura 5.
Per tutti e quattro i gruppi, i taxa con un’alta incidenza sono Methanoregula, Methanobacterium, Methanosarcina, Methanosaeta, Methanomassiliicoccus e Methanocella. I taxa specializzati, che sono significativamente più rappresentati nella maggior parte dei siti all’interno di un dato gruppo, sono stati individuati secondo l’analisi delle specie indicatrici come descritto prima. In totale, sei dei 31 taxa hanno mostrato un valore significativo di indicatore (P < 0,05) (etichettato con un asterisco nella Figura 6). Il gruppo 1 (neutro e freddo) ha mostrato il maggior numero di specialisti con lignaggi di Methanosaeta, Methanolobus e Methanomethylovorans. Methanobacterium è servito come taxon specialista nel gruppo 2, freddo e acido, mentre Methanolinea è stato identificato come specialista nel gruppo 4 (neutro e caldo), ma non è stato praticamente osservato negli altri gruppi. Inoltre, Methanoregula è ampiamente rappresentato nel gruppo 3 (acido e moderato).
Limitazione di dispersione
Un’analisi di regressione lineare (R2= 0,05, P < 0,001) ha indicato una debole correlazione tra la distanza geografica e la struttura della comunità metanigena sul dataset globale. Allo stesso tempo, un test di Mantel ha mostrato che le variabili ambientali hanno una maggiore correlazione con la struttura della comunità rispetto alle distanze geografiche (vedi Tabella 3). Questa tendenza è confermata anche da un test di Mantel parziale che controlla gli effetti di autocorrelazione. Tracciare la distanza geografica contro la similarità di comunità Jaccard non mostra una chiara tendenza lineare, ma modelli che derivano principalmente dalla distribuzione globale dei punti di campionamento (vedi Figura supplementare S4).

TABELLA 3. Analisi di Mantel e test di Mantel parziali per la determinazione dell’influenza delle variabili ambientali e della distanza geografica sulla distribuzione microbica per il dataset globale e un sottocampione di 16 campioni europei.
Per analizzare ulteriormente l’influenza della dispersione, abbiamo limitato la nostra analisi all’Europa, che è stata campionata più densamente e più uniformemente. I test di Mantel e i test di Mantel parziali su questo sottoinsieme hanno riprodotto la tendenza secondo cui i dati della comunità sono più correlati alle variabili ambientali che alle distanze geografiche (vedi tabella 3). Il test di Mantel parziale che controlla le variabili ambientali non ha potuto rilevare alcuna correlazione statisticamente significativa tra la comunità microbica e la distanza geografica. Un’analisi PCA spaziale su questi 16 siti europei implica una struttura spaziale della comunità metanigena (il 23,7% della varianza totale è stata spiegata da questa struttura) che corrisponde a un’autocorrelazione spaziale positiva dei siti come indicato dall’indice I di Moran (I di Moran = 0,4018). Solo il primo autovalore era stabile e corrispondeva a una separazione dei campioni tra l’Europa centrale e gli Stati baltici (Figura supplementare S5). Il piccolo set di dati, tuttavia, complica una robusta assegnazione di questa struttura spaziale osservata a variabili geografiche, ambientali o a entrambe. Abbiamo quindi effettuato una cluster analysis sulle comunità metanigene come descritto sopra e abbiamo rivelato tre gruppi (Figura 7A) che abbiamo proiettato su una mappa geografica (Figura 7B). Il clustering non ha riprodotto la separazione della PCA spaziale lungo il Mar Baltico. Di conseguenza, alcuni siti che sono geograficamente molto vicini l’uno all’altro mostrano strutture di comunità metanogeniche dissimili e si assemblano con cluster diversi. D’altra parte, alcuni siti geograficamente molto distanti mostrano composizioni di comunità molto simili e si raggruppano insieme (Figura 7B). La dispersione regionale dei gruppi in Europa non sembra corrispondere o essere limitata da una struttura geografica.
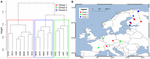
FIGURA 7. Analisi gerarchica dei cluster della matrice di distanza Jaccard tra 16 campioni europei di suolo e sedimenti lacustri. Il metodo di clustering di Ward è stato utilizzato per l’analisi. I siti sono stati divisi in tre gruppi colorati da diversi rettangoli nel dendrogramma di clustering (A) e sono stati proiettati su una mappa europea (B).
Discussione
Identificare e applicare i concetti di biogeografia sulle comunità microbiche è di grande interesse in ecologia microbica. Si ritiene che la biogeografia microbica sia governata dall’interazione evolutiva ed ecologica di quattro processi principali: filtraggio dell’habitat, dispersione, deriva e mutazione (Hanson et al., 2012). Anche se l’influenza della deriva e della mutazione è al di là del potere interpretativo di questo studio, dimostriamo che esistono modelli globali di comunità metanigene in ambienti naturali. Questo studio dimostra un modello biogeografico globale delle comunità metanogeniche che è più associato al filtraggio dell’habitat che alla dispersione geografica. Le comunità metanogeniche dei sedimenti dei laghi di soda, per esempio, si raggruppano strettamente con campioni marini geograficamente distanti (Figura 4) e comunità metanogeniche molto simili si verificano nel suolo europeo e nei sedimenti lacustri nonostante si trovino a grande distanza tra loro (Figura 7). Nel complesso, i nostri tentativi di rivelare una potenziale limitazione della dispersione hanno rivelato una debole influenza della posizione geografica sulla struttura della comunità metanogenica che contrasta con una chiara influenza delle condizioni ambientali. Un’esclusione definitiva degli effetti spaziali sulle comunità microbiche non è possibile con i punti di dati disponibili. I punti di campionamento si concentrano su determinati habitat e/o aree mentre il numero complessivo di campioni è basso. Se c’è un effetto, presumiamo che gli effetti spaziali si verifichino solo su scala regionale o locale. Sono state riportate limitazioni di dispersione locale per le arche ipertermofile causate da barriere geografiche (Whitaker et al., 2003), batteri ossidanti l’ammoniaca in paludi saline locali, ma non su scala regionale e continentale (Martiny et al., 2011), microrganismi in sedimenti di acque profonde insieme alle impostazioni ambientali (Schauer et al., 2009), e per i batteri di un ampio insieme di ambienti di neve eterogenei causati principalmente dalla disponibilità di carbonio alloctono (Lutz et al., 2016). Una minore influenza della limitazione della dispersione sui metanogeni in ambienti naturali significa che i metanogeni possono distribuirsi casualmente nello spazio, sono selezionati con successo dall’ambiente locale se i loro requisiti fisiologici sono soddisfatti e possono stabilire comunità stabili (Martiny et al., 2006; von Mering et al., 2007).
Il concetto di filtraggio degli habitat implica che specie con requisiti ecologici simili dovrebbero co-occorrere più spesso di quanto previsto dal caso (Weiher e Keddy, 2001; Cornwell et al., 2006; Ulrich et al., 2010). Il nostro risultato mostra che grandi differenze nella composizione delle comunità metanogeniche si verificano tra gli ecosistemi marini e quelli delle zone umide e dei laghi, mentre gli estuari si collocano nel mezzo. Modelli di biogeografia basati su una serie di indagini geniche su campioni ambientali sono stati segnalati anche per i batteri generali (Nemergut et al., 2011), il microbioma delle sorgenti di metano (Ruff et al., 2015), gli archei ossidanti dell’ammoniaca (Cao et al., 2013), i batteri marini pelagici e bentonici (Zinger et al., 2011), e i microrganismi che riciclano l’azoto (Church et al., 2008). Il filtraggio dell’habitat è stato specificamente segnalato per archei non coltivati (Auguet et al., 2010), intere comunità batteriche in ambienti diversi (von Mering et al., 2007; Chaffron et al., 2010) o in torbiere sudamericane a scala regionale (Oloo et al, 2016), così come specifici gruppi batterici come i batteri metanogeni (Knief, 2015) e azotofissatori (Nelson et al., 2016).
I nostri risultati indicano che su scala globale, la salinità regola sostanzialmente la composizione della comunità metanogena e determina grandi differenze tra i gruppi metanogeni marini e terrestri. Anche i metanogeni dei sedimenti dei laghi di soda si raggruppano con quelli dei sedimenti marini (Figura 4) evidenziando l’influenza globale della salinità. Questo risultato è in accordo con altri studi basati sul gene 16S rRNA che rivelano che la salinità è un fattore primario che modella i modelli globali delle comunità batteriche e archeologiche complessive (Lozupone e Knight, 2007; Auguet et al., 2010; Caporaso et al., 2011; Cao et al., 2013). Una bassa influenza della separazione geografica ma un forte impatto della salinità sulle comunità microbiche generali è stata osservata anche in studi precedenti (Logares et al., 2013; Yang et al., 2016). Di conseguenza, la salinità determina in gran parte quali lignaggi possono sopravvivere. In vari habitat l’attività di produzione di metano era negativamente correlata alla salinità (Bartlett et al., 1987; Potter et al., 2009; Poffenbarger et al., 2011). L’inibizione della produzione di metano attraverso la salinità è quindi suggerito che coincida con una ridotta dimensione della popolazione metanigena (Pattnaik et al., 2000). L’effetto della salinità sulla metanogenesi idrogenotrofica, acetotrofica e metilotrofica dipende quindi dal livello di salinità ed è diverso per le diverse vie di metanogenesi (Liu et al., 2016). Attualmente, non esiste un meccanismo chiaro per spiegare l’impatto della salinità sulla struttura della comunità, ma diverse ipotesi possono servire come possibile spiegazione. Fisiologicamente, la salinità influenza l’osmolarità esterna e interna delle cellule. Le cellule metanogeniche non saline hanno sviluppato adattamenti fisiologici per contrastare la pressione di turgore interna, mentre le cellule adattate al sale hanno perso tale caratteristica (Zinder, 1993). Inoltre, l’aumento della salinità può indurre i metanogeni a sintetizzare o assumere una maggiore proporzione di soluti compatibili con un costo energetico e quindi metabolico significativo (McGenity, 2010). Il tratto della tolleranza al sale si manifesta anche nella concentrazione ottimale di NaCl per la crescita di colture pure di metanogeni, poiché abbiamo trovato che gli isolati provenienti da sedimenti marini e sedimenti idrotermali hanno una concentrazione ottimale di NaCl significativamente più alta di quelli provenienti dal suolo (Figura supplementare S2).
Negli ecosistemi terrestri non salini, in particolare nei suoli e nei sedimenti lacustri, la composizione della comunità metanogena è controllata dalla combinazione di temperatura e pH. Di conseguenza, i metanogeni di questi ambienti potrebbero essere classificati in quattro gruppi (Figura 6). A differenza degli ecosistemi marini, gli ecosistemi terrestri non salini mostrano una grande variabilità naturale sia del pH che della temperatura. La temperatura può influenzare non solo il percorso metanogenico ma anche le popolazioni metanogeniche stesse (Conrad, 2007; Rooney-Varga et al., 2007). La produzione di metano può essere notevolmente aumentata se le temperature aumentano come conseguenza delle fasi sensibili alla temperatura durante la fermentazione e l’acetogenesi (Megonigal et al., 2005; Kotsyurbenko et al., 2007). Inoltre, un pH basso può limitare sostanzialmente la disponibilità di acetato impedendo all’acetato di dissociarsi e quindi influenzare negativamente la metanogenesi acetoclastica (Fukuzaki et al., 1990; Bridgham et al., 2013). Questo potrebbe essere un possibile motivo per cui Methanosaeta era assente nel gruppo2, mentre Methanosarcina può passare da una fonte all’altra e non è stata sostanzialmente influenzata. Inoltre, il pH può regolare l’efficienza della produzione di metano e le vie metanogeniche dalle torbiere ombrotrofe a quelle minerotrofiche, attraverso l’inibizione diretta di entrambe le vie di metanogenesi e indirettamente attraverso i suoi effetti sulla fermentazione (Ye et al., 2012). Pertanto, sia la temperatura che il pH possono regolare direttamente o indirettamente le fasi metaboliche associate alla metanogenesi e la fermentazione a monte, che fornisce il substrato per i metanogeni.
Methanoregula è onnipresente e molto abbondante in tutti e quattro i gruppi di habitat terrestri (Figura 6) ma virtualmente assente dal sistema marino e può quindi rivelarsi un proxy per l’influenza dell’acqua dolce nel regno marino. La sua rilevanza globale è stata recentemente riportata altrove (Yang et al., 2017). Nonostante la sua distribuzione onnipresente nei suoli e nei sedimenti lacustri, Methanoregula si presenta come una stirpe indicatrice in habitat acidi con temperature moderate. Inoltre, (1) Methanolinea sembra essersi particolarmente adattato agli ambienti terrestri caldi e neutri, (2) Methanobacterium agli ambienti freddi e acidi, e (3) Methanosaeta agli ambienti a pH neutro, che è coerente con altri studi (Rosenberg et al., 2014) e sottolinea la robustezza del nostro approccio. In generale, le condizioni geochimiche che circondano le comunità metanogeniche porteranno alla differenziazione di nicchia. Poiché l’ordinamento di nicchia tende a lasciare gli specialisti adattativi (Langenheder e Székely, 2011), la progressiva selezione ambientale a lungo termine ha generato una varietà di nicchie che sono state riempite da una serie di specialisti di habitat endemici, che possono essere meno rappresentati o assenti in altre condizioni ambientali diverse. La comunità è anche modellata da fattori biotici, come le interazioni ecologiche, le dinamiche, la competizione e la simbiosi. Nonostante questi fattori biologici, von Mering et al. (2007) hanno trovato che le preferenze di habitat sono spesso notevolmente stabili nel tempo e la composizione tassonomica distintiva delle comunità ambientali, a sua volta, può essere un indicatore importante della loro ecologia e funzione.
Consistente alla preferenza di habitat degli archei metanogeni, sembra che ceppi metanogeni strettamente correlati siano stati spesso isolati da ambienti comparabili. Per esempio, Methanoregulaceae sembra essere abbastanza diversificata in ambienti naturali (Yang et al., 2017) e i ceppi neutri possono resistere alla coltura finora. I ceppi attualmente descritti di Methanoregula sono entrambi ottenuti da ambienti leggermente acidofili, mentre i due rappresentanti di Methanolinea provengono da habitat relativamente caldi come i fanghi dei digestori e il suolo delle risaie, rispettivamente (Rosenberg et al., 2014). Anche se i ceppi di Methanobacterium sono stati isolati da vari ambienti, circa la metà degli isolati esistenti di questo genere esibiscono pH optima leggermente inferiori a 7. Ciò significa che i lignaggi indicatori, che sono stati identificati sulla base delle sequenze ambientali, potrebbero riflettere le differenziazioni di fisiologia e fonti delle cultivar metanogeniche esistenti. Per esempio, la salinità dell’habitat, come proprietà generale dell’habitat, può esporre progressivamente gli organismi a una forte selezione ambientale e filtrare l’assemblaggio di un nuovo insieme di specie che sono più adatte alla salinità ambientale (Logares et al., 2013).
La conservazione e gestione della biodiversità è una sfida primaria della nostra società attuale. Qui dimostriamo che gli archei metanogenici degli ambienti naturali sono più diversi nei sedimenti di estuario. Gli estuari sono zone di transizione tra gli ecosistemi marini e terrestri. Questo permette due processi principali che possono contribuire alla ricchezza delle specie. In primo luogo, i microbi del mare e della terra si mescolano negli estuari e alla fine comprendono un’alta diversità generale (McLusky e Elliott, 2004). Per esempio, è stata osservata l’alta diversità di batteri di estuario, archei, funghi e persino batteri specifici che svolgono funzioni uniche (Cunliffe et al., 2008; Mosier e Francis, 2008; Crump et al., 2012). Un altro aspetto è l’alto livello di nutrienti dovuto agli apporti terrestri e di marea di cui si nutrono gli organismi dell’estuario (McLusky e Elliott, 2004; Statham, 2012). In questo contesto, gli ambienti di estuario sono importanti per recuperare la novità generica per i metanogeni. Finora, gli effetti della diversità delle specie sui processi ecosistemici hanno attirato notevoli sforzi di ricerca. Il legame tra biodiversità e funzione dell’ecosistema è ancora in discussione e rimane elusivo per le comunità microbiche (Loreau et al., 2001; Tilman et al., 2014). Anche se i suoli e i sedimenti lacustri sono fonti primarie di metano e anche habitat con elevata diversità metanogena, proponiamo che la ricchezza di specie non sia un proxy appropriato del potenziale di produzione di metano e delle emissioni di metano dell’ecosistema; sembra piuttosto riflettere l’eterogeneità e la storia dell’ambiente. Classificare gli ambienti secondo la loro ricchezza di specie non significa necessariamente il potenziale dei tassi di emissione di metano che sono più alti dai suoli e dai laghi e comparabilmente minori dagli estuari (Figura 3 e Figura supplementare S1).
Infine, la mancanza di informazioni ambientali nei database pubblici può aver ostacolato una piena interpretazione sui driver ambientali osservati qui. Anche se ci sono sempre più dati di sequenziamento sui metanogeni nella letteratura e nei database pubblici, le relative variabili abiotiche fornite sono spesso inconsistenti e scarse. La quantità limitata di informazioni coerenti per le variabili ambientali limita l’applicazione di analisi statistiche multivariate. Ricordando che i fattori abiotici in questo studio possono spiegare solo una frazione limitata delle varianze di comunità suggerisce che mancano altre variabili esplicative. Di particolare importanza potrebbero essere le concentrazioni e la disponibilità di substrati metanogenici come acetato, idrogeno e metilammine. Tuttavia, i parametri abiotici potrebbero non essere mai sufficienti a spiegare completamente ciò che struttura gli assemblaggi metanogenici semplicemente perché gli habitat hanno storie diverse e possono essere studiati solo localmente. Inoltre, tra i dati mcrA disponibili le vaste aree del Subartico e dell’Artico russo e canadese sono scarsamente rappresentate. Una migliore copertura geografica e una distribuzione uniforme dei dati del gene mcrA migliorerebbero la valutazione delle comunità metanogeniche su scala globale.
Contributi degli autori
SL e XW hanno progettato lo studio. XW e SY hanno raccolto e analizzato i dati. XW, SY e FH hanno eseguito l’analisi statistica. XW, MW e SY hanno effettuato la correzione filogenetica. XW, SY, FH, MW, DW e SL hanno interpretato i risultati e scritto l’articolo. Tutti gli autori hanno contribuito alle discussioni e hanno rivisto il manoscritto.
Conflict of Interest Statement
Gli autori dichiarano che la ricerca è stata condotta in assenza di relazioni commerciali o finanziarie che potrebbero essere interpretate come un potenziale conflitto di interessi.
Acknowledgments
Il supporto finanziario a XW (Grant No. 201408620031 a XW) fornito dal China Scholarship Council (CSC) è grato. Questo studio è stato sostenuto dalla Helmholtz Gemeinschaft (HGF) finanziando il Helmholtz Young Investigators Group di SL (VH-NG-919).
Materiale supplementare
Il materiale supplementare per questo articolo può essere trovato online a: https://www.frontiersin.org/article/10.3389/fmicb.2017.01339/full#supplementary-material
Footnotes
- ^ http://metanogen.biotech.uni.wroc.pl/
- ^ http://www.ncbi.nlm.nih.gov/
- ^ http://metanogen.biotech.uni.wroc.pl/
- ^ https://www.ngdc.noaa.gov/mgg/shorelines/gshhs.html
- ^ http://qgis.osgeo.org
Auguet, J. C., Barberan, A., e Casamayor, E. O. (2010). Modelli ecologici globali in Archaea non coltivati. ISME J. 4, 182-190. doi: 10.1038/ismej.2009.109
PubMed Abstract | CrossRef Full Text | Google Scholar
Bange, H. W., Bartell, U. H., Rapsomanikis, S., e Andreae, M. O. (1994). Metano nel Mar Baltico e nel Mare del Nord e una rivalutazione delle emissioni marine di metano. Glob. Biogeochem. Cycles 8, 465-480. doi: 10.1029/94GB02181
CrossRef Full Text | Google Scholar
Barreto, D. P., Conrad, R., Klose, M., Claus, P., e Enrich-Prast, A. (2014). Distanza-decadimento e taxa-area relazioni per batteri, archei e archei metanogeni in un sedimento di lago tropicale. PLoS ONE 9:e110128. doi: 10.1371/journal.pone.0110128
PubMed Abstract | CrossRef Full Text | Google Scholar
Bartlett, K. B., Bartlett, D. S., Harriss, R. C., e Sebacher, D. I. (1987). Emissioni di metano lungo un gradiente di salinità delle paludi salmastre. Biogeochemistry 4, 183-202. doi: 10.1007/BF02187365
CrossRef Full Text | Google Scholar
Borrel, G., O’Toole, P. W., Harris, H. M. B., Peyret, P., Brugere, J. F., e Gribaldo, S. (2013). Dati filogenomici supportano un settimo ordine di metanogeni metilotrofi e fornire intuizioni sull’evoluzione della metanogenesi. Genoma Biol. Evol. 5, 1769-1780. doi: 10.1093/Gbe/Evt128
PubMed Abstract | CrossRef Full Text | Google Scholar
Bousquet, P., Ciais, P., Miller, J., Dlugokencky, E., Hauglustaine, D., Prigent, C., et al. Contributo delle fonti antropogeniche e naturali alla variabilità del metano atmosferico. Natura 443, 439-443. doi: 10.1038/nature05132
PubMed Abstract | CrossRef Full Text | Google Scholar
Bridgham, S. D., Cadillo-Quiroz, H., Keller, J. K., e Zhuang, Q. (2013). Emissioni di metano dalle zone umide: prospettive biogeochimiche, microbiche e di modellazione dalla scala locale a quella globale. Glob. Chang. Biol. 19, 1325-1346. doi: 10.1111/gcb.12131
PubMed Abstract | CrossRef Full Text | Google Scholar
Cao, H., Auguet, J.-C., e Gu, J.-D. (2013). Modello ecologico globale di ammoniaca-ossidante archaea. PLoS ONE 8:e52853. doi: 10.1371/journal.pone.0052853
PubMed Abstract | CrossRef Full Text | Google Scholar
Caporaso, J. G., Lauber, C. L., Walters, W. A., Berg-Lyons, D., Lozupone, C. A., Turnbaugh, P. J., et al. (2011). Modelli globali di 16S rRNA diversità ad una profondità di milioni di sequenze per campione. Proc. Natl. Acad. Sci. U.S.A. 108, 4516-4522. doi: 10.1073/pnas.1000080107
PubMed Abstract | CrossRef Full Text | Google Scholar
Chaffron, S., Rehrauer, H., Pernthaler, J., e von Mering, C. (2010). Una rete globale di microbi coesistenti da dati ambientali e di sequenza del genoma intero. Genome Res. 20, 947-959. doi: 10.1101/gr.104521.109
PubMed Abstract | CrossRef Full Text | Google Scholar
Church, M. J., Bjorkman, K. M., Karl, D. M., Saito, M. A. e Zehr, J. P. (2008). Distribuzioni regionali di batteri che fissano l’azoto nell’Oceano Pacifico. Limnol. Oceanogr. 53, 63-77. doi: 10.4319/lo.2008.53.1.0063
CrossRef Full Text | Google Scholar
Conrad, R. (2007). “Ecologia microbica dei metanogeni e dei metanotrofi”, in Advances in Agronomy, ed. L. S. Donald (Cambridge, MA: Academic Press), 1-63.
Google Scholar
Cornwell, W. K., Schwilk, D. W., and Ackerly, D. D. (2006). Un test basato sul tratto per il filtraggio dell’habitat: volume convesso dello scafo. Ecology 87, 1465-1471. doi: 10.1890/0012-9658(2006)872.0.CO;2
PubMed Abstract | CrossRef Full Text | Google Scholar
Crump, B. C., Amaral-Zettler, L. A., and Kling, G. W. (2012). La diversità microbica nelle acque dolci artiche è strutturata dall’inoculazione di microbi dal suolo. ISME J. 6, 1629-1639. doi: 10.1038/ismej.2012.9
PubMed Abstract | CrossRef Full Text | Google Scholar
Cui, M., Ma, A., Qi, H., Zhuang, X., Zhuang, G., e Zhao, G. (2015). La temperatura più calda accelera le emissioni di metano dalla zona umida Zoige sull’altopiano tibetano senza cambiare la composizione della comunità metanogena. Sci. Rep. 5:11616. doi: 10.1038/srep11616
PubMed Abstract | CrossRef Full Text | Google Scholar
Cunliffe, M., Schafer, H., Harrison, E., Cleave, S., Upstill-Goddard, R., e Murrell, J. C. (2008). Analisi filogenetica e funzionale dei geni delle comunità batteriche e arcaiche associate al microstrato superficiale di un estuario. ISME J. 2, 776-789. doi: 10.1038/ismej.2008.28
PubMed Abstract | CrossRef Full Text | Google Scholar
Dray, S., and Dufour, A. B. (2007). Il pacchetto ade4: implementazione del diagramma di dualità per gli ecologi. J. Stat. Softw. 22, 1-20. doi: 10.18637/jss.v022.i04
CrossRef Full Text | Google Scholar
Dufrene, M., and Legendre, P. (1997). Assemblaggi di specie e specie indicatrici: la necessità di un approccio asimmetrico flessibile. Ecol. Monogr. 67, 345-366. doi: 10.2307/2963459
CrossRef Full Text | Google Scholar
Edgar, R. C., Haas, B. J., Clemente, J. C., Quince, C., and Knight, R. (2011). UCHIME migliora la sensibilità e la velocità di rilevamento chimera. Bioinformatica 27, 2194-2200. doi: 10.1093/bioinformatica/btr381
PubMed Abstract | CrossRef Full Text | Google Scholar
Evans, P. N., Parks, D. H., Chadwick, G. L., Robbins, S. J., Orphan, V. J., Golding, S. D., et al. (2015). Metanismo del metano nel phylum Bathyarchaeota arcaico rivelato dalla metagenomica genoma-centrica. Scienza 350, 434-438. doi: 10.1126/science.aac7745
PubMed Abstract | CrossRef Full Text | Google Scholar
Pesce, J. A., Chai, B., Wang, Q., Sun, Y., Brown, C. T., Tiedje, J. M., et al. (2013). FunGene: il gene funzionale pipeline e repository. Fronte. Microbiol. 4:291. doi: 10.3389/fmicb.2013.00291
PubMed Abstract | CrossRef Full Text | Google Scholar
Frank-Fahle, B. A., Yergeau, E., Greer, C. W., Lantuit, H., e Wagner, D. (2014). Potenziale funzionale microbico e composizione della comunità nei suoli affetti da permafrost dell’Artico canadese nord-occidentale. PLoS ONE 9:e84761. doi: 10.1371/journal.pone.0084761.g001
PubMed Abstract | CrossRef Full Text | Google Scholar
Fukuzaki, S., Nishio, N., e Nagai, S. (1990). Cinetica della fermentazione metanogenica di acetato. Appl. Ambiente. Microbiol. 56, 3158-3163.
Google Scholar
Hanson, C. A., Fuhrman, J. A., Horner-Devine, M. C., and Martiny, J. B. (2012). Oltre i modelli biogeografici: i processi che modellano il paesaggio microbico. Nat. Rev. Microbiol. 10, 497-506. doi: 10.1038/Nrmicro2795
PubMed Abstract | CrossRef Full Text | Google Scholar
Holm, G. O., Perez, B. C., McWhorter, D. E., Krauss, K. W., Johnson, D. J., Raynie, R. C., et al. (2016). Flussi di metano a livello di ecosistema dalle paludi d’acqua dolce e salmastre di marea del delta del fiume Mississippi: implicazioni per i progetti di carbonio delle zone umide costiere. Wetlands 36, 401-413. doi: 10.1007/s13157-016-0746-7
CrossRef Full Text | Google Scholar
Hunger, S., Schmidt, O., Hilgarth, M., Horn, M. A., Kolb, S., Conrad, R., et al. (2011). Competere formate- e anidride carbonica-utilizzando procarioti in un anossico metano-emettendo suolo fen. Appl. Environ. Microbiol. 77, 3773-3785. doi: 10.1128/AEM.00282-11
PubMed Abstract | CrossRef Full Text | Google Scholar
IPCC (2007). “Climate Change 2007: the Physical Science Basis,” in Proceedings of the Contribution of Working Group I to the Fourth Assessment Report of the Intergovernmental Panel on Climate Change, eds D. Qin, S. Solomon, M. Manning, Z. Chen, M. Marquis, K. B. Averyt, et al. (Cambridge: Cambridge University Press).
Google Scholar
Jabłoński, S., Rodowicz, P., e Łukaszewicz, M. (2015). Database di archei metanogeni contenente caratteristiche fisiologiche e biochimiche. Int. J. Syst. Evol. Microbiol. 65, 1360-1368. doi: 10.1099/ijs.0.000065
PubMed Abstract | CrossRef Full Text | Google Scholar
Kirschke, S., Bousquet, P., Ciais, P., Saunois, M., Canadell, J. G., Dlugokencky, E. J., et al. (2013). Tre decenni di fonti e pozzi di metano globale. Nat. Geosci. 6, 813-823. doi: 10.1038/Ngeo1955
CrossRef Full Text | Google Scholar
Knief, C. (2015). Diversità e preferenze di habitat di batteri metanotrofi aerobi coltivati e incolti valutati sulla base di pmoA come marcatore molecolare. Fronte. Microbiol. 6:1346. doi: 10.3389/fmicb.2015.01346
PubMed Abstract | CrossRef Full Text | Google Scholar
Kotsyurbenko, O., Friedrich, M., Simankova, M., Nozhevnikova, A., Golyshin, P., Timmis, K., et al. Passaggio da acetoclastico a H2-dipendente metanogenesi in una torbiera siberiana occidentale a bassi valori di pH e isolamento di un ceppo di Methanobacterium acidofilo. Appl. Environ. Microbiol. 73, 2344-2348. doi: 10.1128/AEM.02413-06
PubMed Abstract | CrossRef Full Text | Google Scholar
Langenheder, S., e Székely, A. J. (2011). Specie ordinamento e processi neutri sono entrambi importanti durante l’assemblaggio iniziale delle comunità batteriche. ISME J. 5, 1086-1094. doi: 10.1038/ismej.2010.207
PubMed Abstract | CrossRef Full Text | Google Scholar
Liebner, S., Ganzert, L., Kiss, A., Yang, S., Wagner, D., e Svenning, M. M. (2015). Shifts in composizione della comunità metanogenica e flussi di metano lungo la degradazione del permafrost discontinuo. Front. Microbiol. 6:356. doi: 10.3389/fmicb.2015.00356
PubMed Abstract | CrossRef Full Text | Google Scholar
Liu, Y. Q., Priscu, J. C., Xiong, J. B., Conrad, R., Vick-Majors, T., Chu, H. Y., et al. (2016). La salinità guida i modelli di distribuzione degli archei nei sedimenti lacustri d’alta quota sull’altopiano tibetano. FEMS Microbiol. Ecol. 92:fiw033. doi: 10.1093/femsec/fiw033
PubMed Abstract | CrossRef Full Text | Google Scholar
Logares, R., Lindstrom, E. S., Langenheder, S., Logue, J. B., Paterson, H., Laybourn-Parry, J., et al. (2013). Biogeografia delle comunità batteriche esposte al progressivo cambiamento ambientale a lungo termine. ISME J. 7, 937-948. doi: 10.1038/ismej.2012.168
PubMed Abstract | CrossRef Full Text | Google Scholar
Loreau, M., Naeem, S., Inchausti, P., Bengtsson, J., Grime, J. P., Hector, A., et al. Biodiversità e funzionamento degli ecosistemi: conoscenze attuali e sfide future. Scienza 294, 804-808. doi: 10.1126/science.1064088
PubMed Abstract | CrossRef Full Text | Google Scholar
Lozupone, C. A., e Knight, R. (2007). Modelli globali nella diversità batterica. Proc. Natl. Acad. Sci. U.S.A. 104, 11436-11440. doi: 10.1073/pnas.0611525104
PubMed Abstract | CrossRef Full Text | Google Scholar
Luton, P. E., Wayne, J. M., Sharp, R. J., and Riley, P. W. (2002). Il gene mcrA come alternativa al 16S rRNA nell’analisi filogenetica delle popolazioni di metanogeni in discarica. Microbiologia 148, 3521-3530. doi: 10.1099/00221287-148-11-3521
PubMed Abstract | CrossRef Full Text | Google Scholar
Lutz, S., Anesio, A. M., Raiswell, R., Edwards, A., Newton, R. J., Gill, F., et al. (2016). La biogeografia dei microbiomi di neve rossa e il loro ruolo nello scioglimento dei ghiacciai artici. Nat. Commun. 7:11968. doi: 10.1038/ncomms11968
PubMed Abstract | CrossRef Full Text | Google Scholar
Martiny, J. B. H., Bohannan, B. J. M., Brown, J. H., Colwell, R. K., Fuhrman, J. A., Green, J. L., et al. (2006). Biogeografia microbica: mettere i microrganismi sulla mappa. Nat. Rev. Microbiol. 4, 102-112. doi: 10.1038/Nrmicro1341
PubMed Abstract | CrossRef Full Text | Google Scholar
Martiny, J. B. H., Eisen, J. A., Penn, K., Allison, S. D., and Horner-Devine, M. C. (2011). Driver di beta-diversità batterica dipendono dalla scala spaziale. Proc. Natl. Acad. Sci. U.S.A. 108, 7850-7854. doi: 10.1073/pnas.1016308108
PubMed Abstract | CrossRef Full Text | Google Scholar
McArdle, B. H., e Anderson, M. J. (2001). Adattamento di modelli multivariati ai dati di comunità: un commento sull’analisi di ridondanza basata sulla distanza. Ecology 82, 290-297. doi: 10.1890/0012-9658(2001)0822.0.CO;2
CrossRef Full Text | Google Scholar
McCalley, C. K., Woodcroft, B. J., Hodgkins, S. B., Wehr, R. A., Kim, E.-H., Mondav, R., et al. (2014). Dinamica del metano regolata dalla risposta della comunità microbica al disgelo del permafrost. Nature 514, 478-481. doi: 10.1038/nature13798
PubMed Abstract | CrossRef Full Text | Google Scholar
McGenity, T. J. (2010). “Metanogeni e metanogenesi in ambienti ipersalini”, in Handbook of Hydrocarbon and Lipid Microbiology, ed. K. N. Timmis (Berlin: Springer), 665-680. doi: 10.1007/978-3-540-77587-4_53
CrossRef Full Text | Google Scholar
McLusky, D. S., and Elliott, M. (2004). L’ecosistema estuarino: Ecologia, minacce e gestione. New York, NY: Oxford University Press. doi: 10.1093/acprof:oso/9780198525080.001.0001
CrossRef Full Text | Google Scholar
Megonigal, J. P., Mines, M. E., and Visscher, P. T. (2005). “Metabolismo anaerobico: legami con i gas traccia e i processi aerobici”, in Biogeochemistry, ed. W. H. Schlesinger (Oxford: Elsevier), 350-362.
Google Scholar
Milferstedt, K., Youngblut, N. D., and Whitaker, R. J. (2010). Struttura spaziale e persistenza delle popolazioni di metanogeni in laghi di torbiera umica. ISME J. 4, 764-776. doi: 10.1038/Ismej.2010.7
PubMed Abstract | CrossRef Full Text | Google Scholar
Mosier, A. C., e Francis, C. A. (2008). Abbondanza relativa e la diversità di ammoniaca-ossidante archei e batteri nell’estuario della baia di San Francisco. Environ. Microbiol. 10, 3002-3016. doi: 10.1111/j.1462-2920.2008.01764.x
PubMed Abstract | CrossRef Full Text | Google Scholar
Nazaries, L., Murrell, J. C., Millard, P., Baggs, L., e Singh, B. K. (2013). Metano, microbi e modelli: comprensione fondamentale del ciclo del metano del suolo per le previsioni future. Environ. Microbiol. 15, 2395-2417. doi: 10.1111/1462-2920.12149
PubMed Abstract | CrossRef Full Text | Google Scholar
Nelson, M. B., Martiny, A. C., and Martiny, J. B. H. (2016). Biogeografia globale dei tratti microbici di riciclaggio dell’azoto nel suolo. Proc. Natl. Acad. Sci. U.S.A. 113, 8033-8040. doi: 10.1073/pnas.1601070113
PubMed Abstract | CrossRef Full Text | Google Scholar
Nemergut, D. R., Costello, E. K., Hamady, M., Lozupone, C., Jiang, L., Schmidt, S. K., et al. (2011). Modelli globali nella biogeografia dei taxa batterici. Environ. Microbiol. 13, 135-144. doi: 10.1111/j.1462-2920.2010.02315.x
PubMed Abstract | CrossRef Full Text | Google Scholar
Nobu, M. K., Narihiro, T., Kuroda, K., Mei, R., e Liu, W. T. (2016). Inseguendo l’inafferrabile Euryarchaeota classe WSA2: i genomi rivelano un metanogeno metil-riduttore unicamente fastidioso. ISME J. 10, 2478-2487. doi: 10.1038/ismej.2016.33
PubMed Abstract | CrossRef Full Text | Google Scholar
Oksanen, J., Blanchet, F. G., Kindt, R., Legendre, P., Minchin, P. R., O’Hara, R. B., et al. (2015). vegan: Community Ecology Package. Pacchetto R versione 2.2.0. Disponibile presso: https://CRAN.R-project.org/package=vegan
Google Scholar
Oloo, F., Valverde, A., Quiroga, M. V., Vikram, S., Cowan, D., and Mataloni, G. (2016). L’eterogeneità dell’habitat e la connettività modellano le comunità microbiche nelle torbiere sudamericane. Sci. Rep. 6:25712. doi: 10.1038/srep25712
PubMed Abstract | CrossRef Full Text | Google Scholar
Pattnaik, P., Mishra, S. R., Bharati, K., Mohanty, S. R., Sethunathan, N., e Adhya, T. K. (2000). Influenza della salinità sulla metanogenesi e sulla microflora associata in terreni tropicali di riso. Microbiol. Res. 155, 215-220. doi: 10.1016/S0944-5013(00)80035-X
PubMed Abstract | CrossRef Full Text | Google Scholar
Poffenbarger, H. J., Needelman, B. A., e Megonigal, J. P. (2011). Influenza della salinità sulle emissioni di metano dalle paludi di marea. Wetlands 31, 831-842. doi: 10.1007/s13157-011-0197-0
PubMed Abstract | CrossRef Full Text | Google Scholar
Potter, E. G., Bebout, B. M., and Kelley, C. A. (2009). Composizione isotopica del metano e dei substrati metanogenici dedotti lungo un gradiente di salinità in un sistema microbico ipersalino. Astrobiologia 9, 383-390. doi: 10.1089/ast.2008.0260
PubMed Abstract | CrossRef Full Text | Google Scholar
R Core Team (2014). R: Un linguaggio e un ambiente per il calcolo statistico (versione 3.0.3). Vienna: R Foundation for Statistical Computing.
Google Scholar
Roberts, D. (2016). labdsv: Ordinazione e analisi multivariata per l’ecologia. Pacchetto R versione 1.8-0. Disponibile presso: https://CRAN.R-project.org/package=labdsv
Google Scholar
Rooney-Varga, J. N., Giewat, M. W., Duddleston, K. N., Chanton, J. P., and Hines, M. E. (2007). Legami tra la struttura della comunità arcaica, il tipo di vegetazione e il percorso metanogenico nelle torbiere dell’Alaska. FEMS Microbiol. Ecol. 60, 240-251. doi: 10.1111/j.1574-6941.2007.00278.x
PubMed Abstract | CrossRef Full Text | Google Scholar
Rosenberg, E., DeLong, E. F., Lory, S., Stackebrandt, E., and Thompson, F. (2014). I procarioti: Altri principali lignaggi di batteri e gli archei. Berlino: Springer.
Google Scholar
Ruff, S. E., Biddle, J. F., Teske, A. P., Knittel, K., Boetius, A., e Ramette, A. (2015). Dispersione globale e diversificazione locale del microbioma metano seep. Proc. Natl. Acad. Sci. U.S.A. 112, 4015-4020. doi: 10.1073/pnas.1421865112
PubMed Abstract | CrossRef Full Text | Google Scholar
Schauer, R., Bienhold, C., Ramette, A., e Harder, J. (2009). Diversità batterica e biogeografia nei sedimenti superficiali di acque profonde dell’Oceano Atlantico meridionale. ISME J. 4, 159-170. doi: 10.1038/ismej.2009.106
PubMed Abstract | CrossRef Full Text | Google Scholar
Schloss, P. D., Westcott, S. L., Ryabin, T., Hall, J. R., Hartmann, M., Hollister, E. B., et al. (2009). Introducendo mothur: open-source, indipendente dalla piattaforma, software supportato dalla comunità per descrivere e confrontare le comunità microbiche. Appl. Environ. Microbiol. 75, 7537-7541. doi: 10.1128/Aem.01541-09
PubMed Abstract | CrossRef Full Text | Google Scholar
Statham, P. J. (2012). Nutrienti negli estuari – una panoramica e gli impatti potenziali del cambiamento climatico. Sci. Total Environ. 434, 213-227. doi: 10.1016/j.scitotenv.2011.09.088
PubMed Abstract | CrossRef Full Text | Google Scholar
Thauer, R. K., Kaster, A.-K., Seedorf, H., Buckel, W., e Hedderich, R. (2008). Archaea metanogeni: differenze ecologicamente rilevanti nella conservazione dell’energia. Nat. Rev. Microbiol. 6, 579-591. doi: 10.1038/Nrmicro1931
PubMed Abstract | CrossRef Full Text | Google Scholar
Tilman, D., Isbell, F., and Cowles, J. M. (2014). Biodiversità e funzionamento degli ecosistemi. Annu. Rev. Ecol. Evol. Syst. 45, 471-493. doi: 10.1146/annurev-ecolsys-120213-091917
CrossRef Full Text | Google Scholar
Ulrich, W., Hajdamowicz, I., Zalewski, M., Stañska, M., Ciurzycki, W., and Tykarski, P. (2010). Assortimento di specie o filtraggio dell’habitat: un caso di studio delle comunità di ragni sulle isole del lago. Ecol. Res. 25, 375-381. doi: 10.1007/s11284-009-0661-y
CrossRef Full Text | Google Scholar
Vanwonterghem, I., Evans, P. N., Parks, D. H., Jensen, P. D., Woodcroft, B. J., Hugenholtz, P., et al. (2016). Metanogenesi metilotrofica scoperta nel phylum arcaico Verstraetearchaeota. Nat. Microbiol. 1:16170. doi: 10.1038/nmicrobiol.2016.170
PubMed Abstract | CrossRef Full Text | Google Scholar
von Mering, C., Hugenholtz, P., Raes, J., Tringe, S. G., Doerks, T., Jensen, L. J., et al. Valutazione filogenetica quantitativa delle comunità microbiche in ambienti diversi. Scienza 315, 1126-1130. doi: 10.1126/science.1133420
PubMed Abstract | CrossRef Full Text | Google Scholar
Wagner, D., and Liebner, S. (2009). “Riscaldamento globale e dinamica del carbonio nei suoli di permafrost: produzione e ossidazione del metano”, in Permafrost Soils, ed. R. Margesin (Berlin: Springer), 219-236.
Google Scholar
Weiher, E., and Keddy, P. (2001). Regole dell’Assemblea Ecologica: Perspectives, Advances, Retreats. Cambridge: Cambridge University Press.
Google Scholar
Whitaker, R. J., Grogan, D. W., and Taylor, J. W. (2003). Barriere geografiche isolano popolazioni endemiche di archei ipertermofili. Scienza 301, 976-978. doi: 10.1126/science.1086909
PubMed Abstract | CrossRef Full Text | Google Scholar
Wickham, H. (2009). ggplot2: Elegant Graphics for Data Analysis. New York, NY: Springer-Verlag. doi: 10.1007/978-0-387-98141-3
CrossRef Full Text | Google Scholar
Yang, J., Ma, L. A., Jiang, H., Wu, G., and Dong, H. (2016). Salinità forme diversità microbica e la struttura della comunità nei sedimenti superficiali del Qinghai-Tibetan Lakes. Sci. Rep. 6:25078. doi: 10.1038/srep25078
PubMed Abstract | CrossRef Full Text | Google Scholar
Yang, S., Liebner, S., Alawi, M., Ebenhöh, O., e Wagner, D. (2014). Database tassonomico e valore di cut-off per l’elaborazione del gene mcrA 454 dati pirosequencing da MOTHUR. J. Microbiol. Methods 103, 3-5. doi: 10.1016/j.mimet.2014.05.006
PubMed Abstract | CrossRef Full Text | Google Scholar
Yang, S., Liebner, S., Winkel, M., Alawi, M., Horn, F., Dörfer, C., et al. (2017). Analisi approfondita delle comunità metanogeniche del nucleo da zone umide di alta quota affette da permafrost. Soil Biol. Biochem. 111, 66-77. doi: 10.1016/j.soilbio.2017.03.007
CrossRef Full Text | Google Scholar
Ye, R. Z., Jin, Q. S., Bohannan, B., Keller, J. K., McAllister, S. A, e Bridgham, S. D. (2012). Il pH controlla la mineralizzazione anaerobica del carbonio, l’efficienza della produzione di metano e i percorsi metanogenici nelle torbiere attraverso un gradiente ombrotrofico-minerotrofico. Soil Biol. Biochem. 54, 36-47. doi: 10.1016/j.soilbio.2012.05.015
CrossRef Full Text | Google Scholar
Zinder, S. H. (1993). “Ecologia fisiologica dei metanogeni”, in Metanogenesi: Ecology, Physiology, Biochemistry & Genetics, ed. J. G. Ferry (Boston, MA: Springer), 128-206.
Google Scholar
Zinger, L., Amaral-Zettler, L. A., Fuhrman, J. A., Horner-Devine, M. C., Huse, S. M., Welch, D. B. M., et al. (2011). Modelli globali di beta-diversità batterica in ecosistemi del fondo marino e acqua di mare. PLoS ONE 6:e24570. doi: 10.1371/journal.pone.0024570
PubMed Abstract | CrossRef Full Text | Google Scholar