Figura 1. Via di segnalazione del recettore Toll-like
Che cos’è il recettore Toll-like?
I recettori Toll-like (TLR) sono una classe di proteine che svolgono un ruolo chiave nell’immunità innata. Sono recettori trans-membrana a dominio singolo e appartengono ai recettori di riconoscimento dei modelli (PRR) che di solito sono espressi nelle cellule sentinella come i macrofagi e le cellule dendritiche e in molte altre cellule non immunitarie come i fibroblasti e le cellule epiteliali. Riconoscono molecole strutturalmente conservate derivate da microbi che sono chiamate pattern molecolari associati ai patogeni (PAMPs) o molecole derivate da cellule danneggiate, indicate come pattern molecolari associati ai danni (DAMPs). I PAMP includono vari componenti della parete cellulare batterica come il lipopolisaccaride (LPS), il peptidoglicano (PGN) e i lipopeptidi, così come la flagellina, il DNA batterico e l’RNA virale a doppio filamento. I DAMP includono proteine intracellulari come le proteine da shock termico e frammenti di proteine della matrice extracellulare. I PRR attivano vie di segnalazione a valle che portano all’induzione di risposte immunitarie innate producendo citochine infiammatorie, interferone di tipo I (IFN) e altri mediatori. Questi processi non solo innescano risposte difensive immediate dell’ospite come l’infiammazione, ma anche innescano e orchestrano risposte immunitarie adattive specifiche all’antigene. Queste risposte sono essenziali per l’eliminazione dei microbi infettanti e cruciali per la conseguente istruzione delle risposte immunitarie adattative antigene-specifiche.
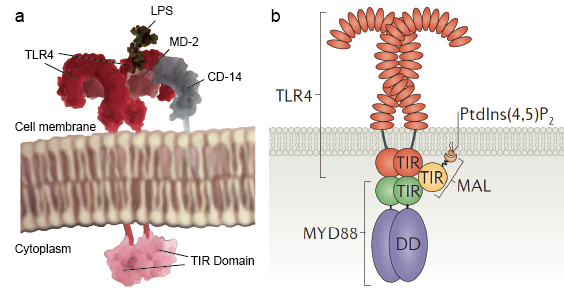
Figura 2. Il diagramma schematico della struttura molecolare dei TLR.
Famiglia di recettori TLR
La famiglia TLR comprende 10 membri (TLR1-TLR10) nell’uomo e 12 (TLR1-TLR9, TLR11-TLR13) nel topo. I TLR localizzano sulla superficie cellulare o in compartimenti intracellulari come l’ER, l’endosoma e il lisosoma. I TLR di superficie cellulare includono TLR1, TLR2, TLR4, TLR5, TLR6 e TLR10, mentre i TLR intracellulari sono localizzati nell’endosoma e includono TLR3, TLR7, TLR8, TLR9, TLR11, TLR12 e TLR13 (Figura 1). I TLR della superficie cellulare riconoscono principalmente i componenti della membrana microbica come lipidi, lipoproteine e proteine. I TLR intracellulari riconoscono gli acidi nucleici derivati da batteri e virus, e riconoscono anche gli acidi auto-nucleici in condizioni di malattia come l’autoimmunità.
La funzione del recettore Toll-like si basa solitamente su un processo di dimerizzazione di due molecole TLR, ma non sempre. Per esempio, TLR-1 e TLR-2 si legheranno l’un l’altro per formare una dimerizzazione quando riconoscono le molecole PAMPs principalmente tra cui lipoproteine, peptidoglicani, acidi lipotechoici (LTA, Gram-), zymosan, mannan, e tGPI-mucina. TLR-2 può anche formare un dimmer con TLR-6 quando riconoscono gli stessi PAMP elencati sopra. TLR-4 può riconoscere il lipopolisaccaride (LPS, Gram+) e formare un omodimero con un’altra molecola TLR-4. TLR-5 può riconoscere la flagellina batterica, ma non forma un omodimero. TLR-11 è funzionale nei topi e riconosce principalmente i batteri uropatogeni. TLR-3, 7, 8, 9, 13 sono espressi sulla superficie dell’endosoma nel citoplasma. TLR3 riconosce l’RNA virale a doppio filamento (dsRNA), i piccoli RNA interferenti e gli auto-RNA derivati da cellule danneggiate. Il TLR-7 è espresso prevalentemente nelle DC plasmacitoidi (pDC) e riconosce l’RNA a singolo filamento (ss) dei virus. Riconosce anche l’RNA dei batteri dello streptococco B nelle DC convenzionali (cDC). TLR8 risponde all’RNA virale e batterico. TLR-9 riconosce il DNA batterico e virale che è ricco di motivi CpG-DNA non metilati. TLR13 riconosce il 23S rRNA batterico e componenti sconosciuti del virus della stomatite vescicolare.
Anche se ci sono tanti tipi di molecole TLR che riconoscono un’ampia gamma di ligandi, tutti questi TLR condividono un quadro strutturale comune nei loro domini extracellulari di legame al ligando. Questi domini adottano tutti strutture a forma di ferro di cavallo costruite da motivi di ripetizione ricchi di leucina. Tipicamente, sul legame con il ligando, due domini extracellulari formano un dimero a forma di ”m” che incastra la molecola del ligando portando i domini transmembrana e citoplasmatici in stretta vicinanza e innescando una cascata di segnalazione a valle (Figura 2).
Percorso di segnalazione del recettore Toll-like
1. Cascata di segnalazione dei recettori Toll-like
I recettori Toll-like permettono alle cellule sentinella come i macrofagi di rilevare i microbi attraverso PAMPs come LPS. LPS è un componente della parete cellulare batterica. Il meccanismo di riconoscimento del lipopolisaccaride da parte dei recettori Toll-like è complesso e richiede diverse proteine accessorie. Una proteina del siero, la LPS-binding protein, lega i monomeri di LPS e li trasferisce a una proteina chiamata CD14. CD14 può essere solubile o legarsi alla superficie cellulare attraverso un’ancora glicosilfosfatidilinositolo. CD 14 consegna e carica LPS al dominio extracellulare dei recettori Toll-like. I TLR sono in grado di rilevare l’LPS con l’aiuto di una proteina accessoria chiamata MD-2. Quindi l’omodimerizzazione dei TLR è indotta quando LPS si lega al complesso TLR-CD14-MD2. Il cambiamento conformazionale dei domini extracellulari avvia la dimerizzazione del dominio citoplasmatico del recettore Toll IL-1 (TIR). Il cambiamento conformazionale TIR fornisce una nuova impalcatura che permette il reclutamento di proteine adattatrici per formare un complesso di segnalazione post-recettore. Il TIR contiene una proteina adattatore myeloid differentiation primary-response protein 88 (MyD88).
MyD88 funziona come un adattatore che collega TLRs/IL-1Rs con molecole di segnalazione a valle che hanno DDs. Riconosce il cambiamento conformazionale nel dominio TIR dei TLR, si lega al nuovo complesso recettoriale e trasferisce la segnalazione attraverso l’interazione amino (N)-terminale del dominio della morte (DD) con le chinasi associate a IL-1R (IRAKs). Questi risultati una cascata complessa con la segnalazione inventa che avverte la cellula di invasione del patogeno. Ci sono 4 IRAKs (IRAK 1, 2, 4, M). Contengono un DD N-terminale e un dominio centrale di serina/treonina-chinasi. IRAK1 e IRAK4 hanno attività chinasica intrinseca, mentre IRAK2 e IRAK-M non hanno attività chinasica rilevabile. IRAK4 attivato da MyD88 e continua ad attivare IRAK1. IRAK1 poi attiva il TRAF6 a valle. TRAF6 è un membro della famiglia TRAF (tumor necrosis factor receptor – TNFR) che media le vie di segnalazione delle citochine. Dopo la stimolazione, TRAF6 viene reclutato nel complesso del recettore e attivato da IRAK-1 che si lega al dominio TRAF di TRAF6. Poi, il complesso IRAK-1/TRAF6 si dissocia dal recettore e si associa alla chinasi 1 attivata dal TGF-beta (TAK1) e alle proteine leganti TAK1, TAB1 e TAB2. Il complesso di TRAF6, TAK1, TAB1 e TAB2 si sposta nel citoplasma, dove forma un grande complesso con altre proteine, come le ligasi E2 Ubc13 e Uev1A. È stato dimostrato che il complesso Ubc13 e Uev1A catalizza la sintesi di una catena di poliubiquitina legata a Lys 63 di TRAF6 e quindi induce l’attivazione mediata da TRAF6 di TAK1 e infine di NF-kB. Questa via di segnale descritta sopra è chiamata via MyD88-dipendente, poiché il segnale parte dalla molecola MyD88. C’è anche un altro percorso chiamato MyD88-independedt pathway, che la segnalazione non parte da MyD88. Invece, il segnale parte dalla proteina TRIF. TRIF interagisce con TRAF6 e TRAF3.TRAF6 recluta la chinasi RIP-1, che a sua volta interagisce con e attiva il complesso TAK1, portando all’attivazione di NF-kB e MAPKs e all’induzione di citochine infiammatorie. Al contrario, TRAF3 recluta le chinasi legate a IKKTBK1 e IKKi insieme a NEMO per la fosforilazione e l’attivazione di IRF3. IRF3 forma un dimero e trasloca nel nucleo dal citoplasma, induce l’espressione di IFN di tipo I.
2. Segnalazione a valle
Il segnale di TLRs in realtà principalmente attraverso il reclutamento di specifiche molecole adattatrici, portando all’attivazione dei fattori di trascrizione NF-kB e IRFs, che dettano il risultato delle risposte immunitarie innate. Quindi questo percorso di segnalazione a valle è quello di attivare il fattore di trascrizione IRFs, il percorso di segnalazione NF-kB e il percorso MAKP. Puoi trovare informazioni più dettagliate sul percorso di NF-kB e MAKP da:
Percorso di segnalazione NF-kB, percorso di segnalazione P38 e percorso di segnalazione MAKP.
3. Regolazione del percorso
Perché c’è una regolazione negativa da una serie di molecole attraverso vari meccanismi per prevenire o terminare le risposte immunitarie eccessive che portano a conseguenze dannose associate all’autoimmunità e alle malattie infiammatorie. L’attivazione della via MyD88-dipendente è soppressa da ST2825, SOCS1 e Cbl-b, e l’attivazione della via TRIF-dipendente è soppressa da SARM e TAG. Queste molecole si associano con MyD88 o TRIF per impedire loro di legarsi ai TLR o alle molecole a valle. L’attivazione di TRAF3 è regolata negativamente da SOCS3 e DUBA. TRAF6 è bersaglio di una serie di molecole inibitorie come A20, USP4, CYLD, TANK, TRIM38 e SHP. L’attivazione di TAK1 è inibita da TRIM30a e A20.
4. Relazione con le malattie
Come TLR è coinvolto nel rilevamento di LPS e potrebbe avere un ruolo nella sepsi, il targeting di TLR è importante per il trattamento di diverse malattie. Oltre a interferire con le risposte dei TLR per trattare le infezioni da agenti patogeni, un’ovvia applicazione clinica delle conoscenze acquisite dagli studi sui TLR è stata quella di utilizzare i ligandi dei TLR come adiuvanti dei vaccini. Inoltre, l’inibizione dei TLR è stata anche tentata in clinica, il cui obiettivo è quello di limitare l’infiammazione eccessiva che è presumibilmente guidata dalla sovra attivazione di un particolare TLR.
| Takeda, Kiyoshi, e Shizuo Akira. “Percorsi di segnalazione TLR”. Seminari in immunologia. Vol. 16. No. 1. Academic Press, 2004. | |
| Akira, Shizuo, e Kiyoshi Takeda. “Segnalazione dei recettori Toll-like”. Nature reviews immunology 4.7 (2004): 499-511. | |
| Lim, Kian-Huat, and Louis M. Staudt. “Segnalazione del recettore Toll-like”. Cold Spring Harbor prospettive in biologia 5.1 (2013): a011247. | |
| eTaro Kawai, Takumi Kawasaki. “Percorsi di segnalazione dei recettori Toll-like”. Pattern Recognition Receptors and Cancer 8916 (2015): 7. | |
| Gay, Nicholas J., et al. “Assembly and localization of Toll-like receptor signalling complexes.” Nature reviews Immunology 14.8 (2014): 546-558. | |
| O’Neill, Luke AJ, Douglas Golenbock, e Andrew G. Bowie. “La storia dei recettori Toll-like che ridefiniscono l’immunità innata”. Nature Reviews Immunology 13.6 (2013): 453-460. | |
| Botos, Istvan, David M. Segal, e David R. Davies. “La biologia strutturale dei recettori Toll-like”. Structure 19.4 (2011): 447-459. |