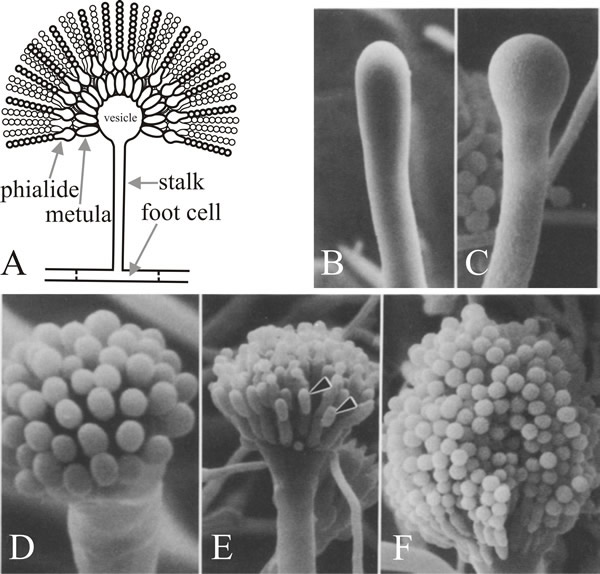
A célula de onde emerge o ramo é a célula do pé conidióforo, que se distingue de outras células vegetativas por ter uma parede secundária pigmentada castanha espessada no interior da sua parede original. O talo cresce apicalmente até atingir um comprimento de cerca de 100 µm quando o ápice incha para formar a vesícula do conidióforo que tem um diâmetro de cerca de 10 µm. Um único nível de numerosos esterigmatos primários, chamados metules, então gema da vesícula e esterigmatos secundários, os piálidos, gema dos ápices expostos dos metules. Os piálidos são as células estaminais que posteriormente sofrem divisões assimétricas repetidas para formar as longas cadeias de conídios enteroblásticos que têm aproximadamente 3 µm de diâmetro (Fig. 7).
Análise genética clássica, por isolamento e análise de mutantes, estabeleceu o esboço genético básico da conidiação de Aspergillus. Entre 300 e 1.000 loci gênicos foram estimados como preocupados com a conidiação através da comparação das frequências de mutação nos loci que afetam a conidiação com as de outras funções (Martinelli & Clutterbuck, 1971). A análise das espécies de mRNA indicou que aproximadamente 6.000 foram expressas em micélio vegetativo e 1.200 adicionais foram encontrados em culturas que incluíam conidióforos e conidia; 200 desses mRNAs adicionais foram encontrados nas próprias conidias. Apenas cerca de 2% dos mutantes de A. nidulans que não tinham conidia tinham defeitos em estágios preocupados com o crescimento e desenvolvimento dos conidióforos, e 85% dos mutantes de conidia também eram defeituosos no crescimento vegetativo hifálico e na obtenção de competência.
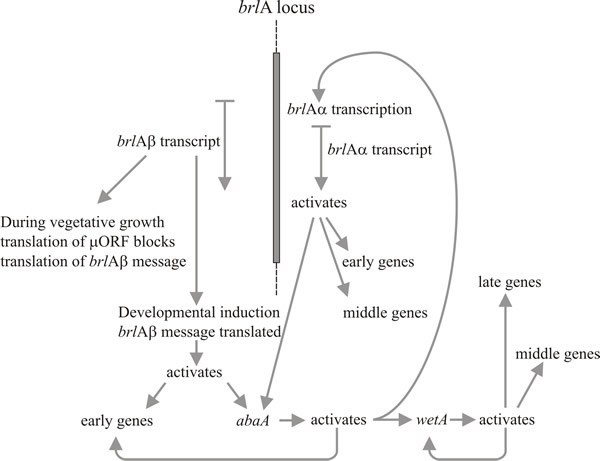
Dois genes, em particular, desempenham um papel chave na morfogênese dos conidios: estes são o gene ‘bristle’ (brlA) que tem defeitos na formação da vesícula e metula, e ‘abacus’ (abaA) no qual as conidias são substituídas por comprimentos de hifas com contas, então presumivelmente é defeituoso no florescimento conidial a partir da pialide e na septação final. Um terceiro gene, wetA, é defeituoso numa fase inicial de maturação dos esporos. Conidia de mutantes do wetA carecem de pigmento e hidrofobicidade; eles se autolisam após algumas horas e não conseguem expressar uma gama de mRNAs específicos do esporte. A transcrição do gene do wetA está ausente nos mutantes brlA e abaA (ou seja, brlA e abaA são epistaticos ao wetA), e estudos de mutantes duplos mostram que estes três genes atuam na ordem: brlA → abaA → wetA.
Uma característica marcante da análise mutacional do desenvolvimento dos conidóforos em A. nidulans é que mutantes destes três genes causam defeitos no conidióforo e na morfogênese dos esporos, enquanto que até 1000 outros loci gênicos causam ausência de conidiação quando mutantes. Isto implica que brlA, abaA e wetA são reguladores que integram a expressão de outros genes que são necessários para a conidiação, mas não são eles próprios dedicados a ela. Muitos dos mutantes da conidiação Aspergillus também eram defeituosos na reprodução sexual, portanto outra conclusão a ser tirada é que existe alguma economia no uso de genes morfogenéticos em diferentes processos de desenvolvimento. Presumivelmente, diferentes modos de desenvolvimento empregam genes estruturais que não são exclusivamente de desenvolvimento, mas funcionam em numerosos caminhos, tendo sua especificidade de desenvolvimento conferida pelos reguladores aos quais eles respondem. Isto é resumido na idéia de que a chave para o desenvolvimento de eucariotas está na capacidade de usar relativamente poucos genes reguladores para integrar as atividades de muitos outros.
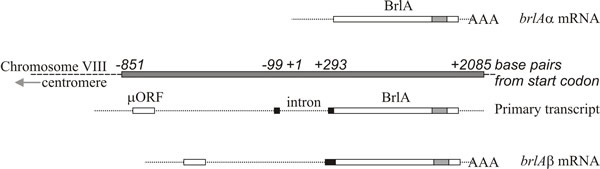
Análise molecular suporta a interpretação de que brlA, abaA e wetA são reguladores; a seqüência brlA codifica uma proteína do dedo de zinco, que é um ativador de transcrição de DNA específico de genes alvo regulados para o desenvolvimento (Fig. 8). Entretanto, essa não é a história toda, porque o produto brlA tem afinidades diferentes para diferentes genes-alvo. De fato, o locus brlA consiste de unidades de transcrição sobrepostas (Fig. 9), sendo a unidade a jusante designada brlAα e a unidade a montante brlAβ; entre elas, seus produtos resolvem dois problemas clássicos de desenvolvimento, como responder a um sinal e como manter essa resposta quando o sinal se dissipou.
As duas unidades de transcrição brlA partilham o mesmo quadro de leitura para a maior parte do seu comprimento, mas brlAβ tem um adicional de 23 resíduos de aminoácidos na extremidade amino-terminal desse quadro de leitura, e a sua transcrição também possui um quadro de leitura separado de 41 resíduos de aminoácidos (chamado µORF) perto do seu terminal 5′. O produto µORF reprime a tradução do quadro de leitura a jusante (BrlA) e, embora a transcrição do brlAβ possa ser detectada em hifas vegetativas, o peptídeo BrlA não é produzido. A repressão causada por µORF só é superada quando o micélio é competente, o que provavelmente é sinalizado como uma limitação de nitrogênio (um sinal ambiental comum para início de esporulação em Ascomycota), o que reduz as piscinas de aminoáctil-tRNA e perturba a regulação translacional por µORF. Quando o efeito do µORF é interrompido, o peptídeo BrlA pode ser traduzido da transcrição existente.
O descobridor deste processo descreveu a ativação do caminho de conidiação como ‘gatilho translacional’ (Timberlake, 1993), sugerindo que o gatilho translacional é uma forma de tornar a diferenciação sensível ao estado nutricional da hifa. Assim, a hifa competente está preparada para empreender o desenvolvimento do conidióforo, mas a ativação irreversível da via de conidiação é impedida por uma repressão translacional que mantém o crescimento vegetativo até que as condições de esporulação sejam ideais.
Ativação do brlA é, portanto, visto como o primeiro passo no desenvolvimento do conidióforo, e seu produto, por sua vez, ativa um painel de genes específicos da conidiação entre os quais está o próximo regulador, abaA. O produto abaA é também uma proteína reguladora transcrítica de ligação ao DNA que melhora a expressão dos genes estruturais induzidos pelo brlA. Os genes brlA e abaA são ativadores recíprocos, porque o abaA também ativa o brlA. Claro que a expressão do brlA deve ocorrer antes que o abaA possa ser expresso, mas a conseqüente ativação do abaA do brlA reforça a expressão deste último e efetivamente faz o progresso do caminho independente de eventos externos. O produto abaA também ativa genes estruturais adicionais e o gene regulador final, o wetA, que ativa os genes estruturais específicos do esporte. Como brlA e abaA não são expressos em conidia diferenciadora, o wetA está provavelmente envolvido na inativação de sua expressão nos esporos (e talvez no phialide também; como o núcleo do esporo atual atinge o estágio de maturação que precisa de genes regulados pelo wetA, os núcleos do phialide devem ser “voltados” para o estado de iniciação do conidium para iniciar a formação do próximo esporo). A expressão de wetA é inicialmente ativada no pialide pela ação sequencial de brlA e abaA, e depois é auto-regulada (o produto wetA ativa a transcrição do wetA). A auto-regulação positiva do wetA mantém sua expressão depois que o conídio foi separado (fisicamente e citologicamente) do pialide.
Timberlake (1993) chamou este mecanismo de fixação de feedback: ativação recíproca, ativação de feedback e auto-regulação das seqüências reguladoras do núcleo reforçam a expressão de todo o caminho, tornando-o independente dos sinais ambientais externos que o iniciaram e permitindo que o esporo continue a maturação mesmo depois da separação do pialide. O desenvolvimento do conidióforo, como muitos outros processos morfogenéticos, está naturalmente dividido em etapas sequenciais. Esta rede reguladora mostra como o desencadeamento translacional pode relacionar um caminho morfogenético ao desenvolvimento da competência, por um lado, e à iniciação em resposta ao(s) taco(s) ambiental(ais), por outro. Após a iniciação, a fixação de feedback resulta na determinação do desenvolvimento no sentido clássico embriológico da morfogênese contínua mesmo quando removida do ambiente iniciador.
Escolhemos deliberadamente nesta seção para basear nossa descrição em pesquisas feitas há cerca de 50 anos, que começaram com o isolamento de um grande número de mutações com fenótipos de conidiação alterados e continuaram com estudos ‘clássicos’ de segregação de genes e complementação de genes (Martinelli & Clutterbuck, 1971). Além de mostrar como nosso entendimento da esporulação tem sido realizado, ele demonstra o valor do uso dessas técnicas ‘antiquadas’ para investigar tais fenômenos.
No entanto, não podemos ignorar a extensa pesquisa que foi concluída sobre os mecanismos moleculares subjacentes ao crescimento e desenvolvimento do Aspergillus. Estes estudos confirmaram que o evento chave na esporulação é a ativação do fator de transcrição do dedo de zinco codificado pelo brlA, e que os genes abaA e wetA são reguladores necessários da conidiação. O fator de transcrição codificado pelo abaA é ativado pelo brlA após a diferenciação dos metulos e durante os estágios médios de desenvolvimento do conidio; e o genewetA, ativado pelo abaA, funciona na fase tardia da conidiação direcionando a síntese de componentes cruciais da parede celular e transformando o metabolismo do conídio maduro. Em Aspergillus nidulans esses três genes criam a via regulatória central, que, com outros genes, controla a expressão gênica específica da conidiação e a seqüência de ativação dos genes envolvidos na aquisição de competência de desenvolvimento, desenvolvimento dos conidióforos e maturação dos esporos (Lee et al., 2016).
Muitos aspectos dessas vias de desenvolvimento têm sido conservados em outros aspergilli. Os membros do gênero Aspergillus estão entre os fungos mais comumente encontrados, e todos se reproduzem assexuadamente formando longas cadeias de conidias. Várias espécies, incluindo Aspergillus oryzae e Aspergillus niger, são utilizadas na indústria para a produção de enzimas e processamento de alimentos; enquanto Aspergillus flavus é responsável pela deterioração dos alimentos ao produzir os mais potentes carcinógenos naturais conhecidos, as aflatoxinas. Outra espécie de preocupação é o patógeno humano oportunista Aspergillus fumigatus, que produz um grande número de pequenas conidias hidrofóbicas como seu principal meio de dispersão e se tornou um patógeno fúngico disseminado pelo ar em países desenvolvidos. Em pacientes imunocomprometidos, A. fumigatus causa uma aspergilose invasiva que tem uma alta taxa de mortalidade. A via de sinalização de desenvolvimento BrlA-AbaA-WetA tem sido conservada na conidiação destas espécies de Aspergillus (Yu, 2010; Tao & Yu, 2011; Krijgsheld et al., 2013). Além disso, estudos proteômicos, transcriptômicos e metabólicos têm fornecido um quadro detalhado das mudanças dinâmicas que ocorrem em muitos milhares de genes, transcrições, enzimas e reações metabólicas durante a formação, maturação, dormência e germinação de Aspergillus conidia (van Leeuwen et al., 2013; Novodvorska et al., 2016; Teertstra et al., 2017).
Atualizado em Julho de 2019