Introdução
Metano (CH4) é um dos principais gases com efeito de estufa. A sua emissão de ambientes naturais como zonas húmidas, oceanos e sedimentos é responsável por mais de 70% do metano atmosférico a nível global (IPCC, 2007). Uma avaliação dos dados publicados revelou diferentes taxas de emissão de metano para zonas húmidas, lagos, rios, estuários e oceanos (em ordem decrescente, Figura Suplementar S1). As zonas húmidas naturais são responsáveis por 62% da produção biogénica de CH4 (Kirschke et al., 2013; Nazaries et al., 2013) e as emissões das zonas húmidas dominam a variabilidade inter-anual das fontes de metano (Bousquet et al., 2006). Em contrapartida, a vasta área de ecossistemas marinhos contribui apenas com cerca de 8% para as fontes naturais de CH4 (Nazaries et al., 2013). Dentro da emissão global de metano oceânico, menos de 10% é contribuído por estuários (Bange et al., 1994).
Methanogenesis, a formação biológica do metano, é realizada por arcaea metanogénica que produz metano principalmente a partir de H2/CO2, grupos metilo ou acetato em condições anóxicas (Thauer et al., 2008). Esta reacção é catalisada pela metil-coenzima M redutase (MCR). O gene mcrA que codifica uma subunidade dessa enzima é um marcador genético comumente usado em pesquisas moleculares (Conrad, 2007; Bridgham et al., 2013). A vantagem do marcador genético mcrA é capturar tanto as assinaturas filogenéticas quanto funcionais dos metanogênicos, oferecendo uma alta profundidade de seqüenciamento para esta função em particular (Luton et al., 2002; Borrel et al., 2013; Yang et al., 2014). Um grande número de seqüências de mcrA foram recuperadas de uma variedade de ambientes naturais. O conjunto de dados mcrA público permite extrair padrões ecológicos gerais e investigar os gradientes ambientais em escala global e regional. Adicionalmente, está disponível uma base de dados que resume as propriedades fisiológicas de 152 isolados metanogénicos1 (Jabłoński et al., 2015). Recentemente, o genoma de fabricação revelou seqüências incomuns de mcrA na nova classe de Methanofastidiosa (Nobu et al., 2016) e a nova phyla de Bathyarchaeota (Evans et al., 2015) e Verstraetearchaeota (Vanwonterghem et al., 2016). Estas novas descobertas expandiram nosso conhecimento sobre a diversidade de potenciais metanogênicos, mas não ocultaram a aplicabilidade do gene mcrA como marcador molecular para a grande maioria das comunidades metanogênicas.
Até agora, comunidades metanogênicas foram detectadas em áreas úmidas, sedimentos, áreas de permafrost, arrozais, digestores, fontes geotérmicas e respiradouros hidrotermais (Conrad, 2007; Thauer et al., 2008; Wagner e Liebner, 2009). A estrutura metanogênica da comunidade foi associada ao pH ambiental, temperatura, salinidade, nível de água subterrânea e dinâmica da vegetação em diferentes escalas espaciais e temporais (Megonigal et al., 2005; Milferstedt et al., 2010; Frank-Fahle et al., 2014; McCalley et al., 2014; Cui et al., 2015; Liebner et al., 2015). Por exemplo, a metanogênese acetoclástica é geralmente dificultada pelo baixo pH, pois reduz a dissociação do acetato (Megonigal et al., 2005; Kotsyurbenko et al., 2007). A vegetação pode fornecer carbono orgânico lábil de alta qualidade para alimentar metanogênicos na forma de exsudados radiculares ou detritos, de modo que os exsudados vegetais geralmente favorecem os metanogênicos acetoclásticos principalmente nos pântanos (Bridgham et al., 2013). O sulfato da água do mar inibe a produção de metano nas zonas húmidas das marés, pelo que a salinidade tem sido usada como um preditor geral das emissões de metano (Holm et al., 2016). Um estudo sobre sedimentos de lagos tibetanos mostrou que o aumento da salinidade inibe os metanogênicos hidrogeno-tróficos, mas melhora a metanogênese acetoclástica (Liu et al., 2016). Estes estudos indicaram factores ambientais para as comunidades metanogénicas, mas focaram-se em habitats únicos ou escalas espaciais limitadas.
A compreensão da adaptação dos metanogénicos a diferentes mudanças ambientais, contudo, requer uma exploração sistemática e global das correlações entre a composição da comunidade microbiana e as condições ambientais (Lozupone e Knight, 2007). Atualmente, apenas alguns estudos abordam a dispersão e filtragem de habitat de comunidades metanogênicas (Auguet et al., 2010; Barreto et al., 2014). Hipótese é que as assembléias metanogênicas são principalmente influenciadas pela filtragem de habitats e que são impulsionadas por controles ambientais globais. Considerando que as taxas de emissão de metano diferem muito entre ecossistemas naturais, a integração explícita da composição, diversidade e biogeografia das assembléias metanogênicas nesses ecossistemas pode ser fundamental para determinar a resposta da produção de metano às mudanças climáticas atuais e futuras. Este meta-estudo é realizado para preencher as lacunas associadas à biogeografia metanogênica, diversidade e seus controles ambientais, usando dados de seqüência mcrA disponíveis publicamente e literatura complementada através de dados fisiológicos de isolados metanogênicos.
Materiais e Métodos
Recolha de dados
Recuperamos seqüências mcrA disponíveis no GenBank (janeiro 2016)2. Para cada acerto, o papel original foi verificado e as seqüências mcrA foram analisadas por um script Perl personalizado. Como nos concentramos em ambientes naturais, as sequências metanogénicas de mcrA foram obtidas de habitats naturais e classificadas como solos, lagos, estuários, sedimentos marinhos e hidrotermais, e vulcões de lama. Foram incluídas cinco bibliotecas de sequenciamento de próxima geração (NGS), além de sequências de bibliotecas de clones. As seqüências foram baixadas sem levar em conta a abundância relativa no conjunto de dados original. Como as seqüências de bibliotecas de clones cobrem principalmente os filótipos abundantes, enquanto o NGS pode capturar uma diversidade muito mais profunda, fizemos um compromisso para usar os dados do NGS, mas mitigamos um erro potencial devido à resolução diferente dos métodos de seqüenciamento. Portanto, escolhemos apenas as sequências representativas das OTUs abundantes com uma abundância relativa superior a 1%. Rejeitamos ainda as sequências NGS que falharam na verificação da tradução de nucleotídeos para sequências proteicas ou com uma qualidade baixa (sequências < 250 bps). Finalmente, construímos um conjunto de dados contendo 4466 sequências únicas de mcrA de 94 locais globalmente distribuídos (Figura 1 e Tabelas Suplementares S1, S2). Além disso, não subtraímos as sequências mcrA de potenciais metanótrofos arqueológicos do conjunto de dados, que foi inevitavelmente detectado no levantamento genómico (Conrad, 2007). Esta parte está além do foco deste estudo.
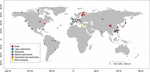
FIGURA 1. Localização dos locais utilizados para este estudo. Os sítios foram agrupados em seis categorias diferentes de acordo com seus habitats e foram etiquetados com cores diferentes. Um resumo dos seis habitats é apresentado nas Tabelas Suplementares S1, S2.
As coordenadas geográficas e as configurações ambientais, incluindo pH, salinidade, elevação, temperatura média anual do ar (MAAT) e precipitação média anual (MAPA) foram extraídas para cada sítio de pesquisa considerado neste estudo das publicações correspondentes, uma vez que os dados estão disponíveis (ver Tabela Suplementar S1). Para contabilizar a falta de parâmetros ambientais em múltiplos estudos, definimos qualitativamente algumas variáveis ambientais de acordo com as descrições do local na literatura relevante, e depois convertemos estes dados da categoria em valores numéricos semi-métricos, por exemplo, definimos sedimentos marinhos, sedimentos hidrotermais, lama vulcânica e amostras de sedimentos de lagos de soda como “salinos”, amostras de sedimentos de solos e lagos de água doce como “não salinos”, e amostras de mangais e estuários como amostras “mistas”.
Processamento da sequência bruta
O processamento da sequência foi implementado com a plataforma do software Mothur (Schloss et al.., 2009). Sequências de diferentes bibliotecas foram agrupadas antes do processamento. Sequências com um comprimento inferior a 350 bp ou mais de 8 bases ambíguas foram descartadas. Subsequentemente, estas sequências foram alinhadas contra um subconjunto pré-alinhado de sequências mcrA, que foram recuperadas da base de dados do FunGene em http://fungene.cme.msu.edu/ (Fish et al., 2013). As sequências quiméricas foram identificadas com o software Mothur usando o método uchime (Edgar et al., 2011), tendo como referência o próprio conjunto de dados. Em seguida, as sequências válidas de nucleotídeos do gene mcrA foram usadas para calcular as distâncias não corrigidas entre sequências de DNA alinhadas em pares e posteriormente atribuídas a unidades taxonômicas operacionais (OTU) a um corte de 84% que corresponde a 97% para o gene 16S rRNA (Yang et al., 2014). A abundância de cada UTR mcrA só foi contabilizada como presença ou ausência. Aumentamos a precisão da classificação taxonômica das OTUs considerando tanto as seqüências de nucleotídeos como de aminoácidos. Ao nível do ADN, a identidade taxonómica foi atribuída pela plataforma Mothur de acordo com uma base de dados de referência (Yang et al., 2014). A nível proteico, as sequências proteicas alinhadas foram utilizadas para construir uma árvore em ARB, e depois a atribuição taxonómica foi baseada na base de dados correspondente. Se a atribuição de uma OTU foi inconsistente, nós jateamos manualmente tanto o nucleotídeo quanto a seqüência proteica na NCBI e determinamos a identidade taxonômica final levando em conta a cobertura da consulta (>95%), a identidade (>84%), e o valor e (<1E-5). Para sequências proteicas, o corte ao nível do género referia-se ao limiar de 83,5% (Hunger et al., 2011).
Análise Ecológica e Estatística
A análise estatística foi feita por vários pacotes R. Ordenações de análise de coordenadas principais (PCoA) foram geradas com base em matrizes de distância Jaccard construídas usando o pacote vegan v2.2.0 (Oksanen et al., 2015). A MANOVA permutacional (análise multivariada de variância) foi realizada para avaliar a fonte de variação da matriz Jaccard (McArdle e Anderson, 2001) em vegan com 104 permutações. As medidas de distância Jaccard são baseadas na presença/ausência da espécie, o que é mais adequado para o nosso conjunto de dados, uma vez que a maioria dos estudos apenas forneceu as seqüências representativas enquanto faltam as informações sobre abundâncias. As frequências de incidência taxonómica em habitats foram visualizadas através de parcelas de bolhas com o pacote ggplot2 (v1.0.0) (Wickham, 2009). A análise hierárquica de agrupamento das comunidades de solos não salinos e sedimentos lacustres foi realizada pela função R ‘hclust’ (R Core Team, 2014). Os aglomerados comunitários obtidos foram descritos de acordo com o regime de pH e temperatura das amostras originais porque usando PCoA previamente identificamos ambos os parâmetros pH e temperatura para influenciar a composição metanogênica da comunidade em solos não-salinos e sedimentos lacustres. A associação de cada linhagem metanogênica com cada um desses aglomerados foi determinada usando análise de espécies indicadoras baseadas em correlação (Dufrene e Legendre, 1997). As espécies indicadoras são definidas aqui como aquelas que são abundantes num tipo específico de habitat (especificidade) e predominantemente encontradas neste tipo de habitat (fidelidade). Neste estudo, os taxa indicadores, semelhantes ao conceito de espécie indicadora, para solos não salinos e sedimentos lacustres foram escolhidos de acordo com um valor indicador (valor IndVal) pelo pacote R labdsv (Roberts, 2016) se a probabilidade de obter um valor indicador tão alto quanto o observado ao longo das iterações especificadas for inferior a 0,05. Os índices Chao2 foram calculados para cada amostra usando a embalagem vegan. O teste Wilcoxon da soma dos índices de Chao2 entre habitats foi realizado pela função R ‘wilcox.test’ (R Core Team, 2014). Para consolidar o impacto da filtragem de habitats nos isolados metanogênicos de arcaea, as características fisiológicas e bioquímicas das culturas metanogênicas descritas foram recuperadas da3 ‘Methanogenic archaea database’ (Jabłoński et al., 2015). Entre eles, os isolados com informação de exigência de NaCl ótima foram filtrados, categorizados e plotados de acordo com sua fonte de isolamento.
Para examinar a influência da limitação da dispersão na estrutura da comunidade metanogênica, uma análise de regressão linear foi realizada com base em uma matriz de distância geográfica e matriz de distância Jaccard da comunidade pela função R ‘lm’ (R Core Team, 2014). Realizamos testes de Mantel e Mantel parcial para avaliar os efeitos da limitação da dispersão de acordo com as duas matrizes novamente usando o pacote vegan em R (Oksanen et al., 2015). Além disso, a análise espacial multivariada (PCA espacial) foi aplicada a 16 amostras de solo e sedimentos lacustres europeus com base no índice I de Moran para explorar a estrutura espacial dos metanogéneos pela função “multispati” no pacote R ade4 (Dray e Dufour, 2007). Além disso, o agrupamento de variância mínima da Ward que foi baseado na matriz de distância Jaccard foi implementado nestas 16 amostras usando a função R “hclust” (R Core Team, 2014) e projetamos os resultados do agrupamento em um mapa geográfico. O shapefile europeu para mapeamento a nível estadual está disponível na base de dados GSHHG (v2.3.6)4. O mapa foi gerado usando QGIS v2.18.25.
Resultados
Biogeografia de Arqueia Metanogênica em Ambientes Naturais
As sequências de genes mcrA de 94 ambientes naturais distribuídos globalmente foram recuperadas. A localização e o tipo de ecossistema de cada um destes 94 locais está ilustrado na Figura 1. As frequências de incidência (presença/ausência) das linhagens metanogénicas foram fundidas de acordo com o tipo de ecossistema e ilustradas na Figura 2. Resumidamente, Methanoregula é o taxon mais frequentemente observado nos solos, juntamente com Methanobacterium, Methanosaeta, Methanocella, Methanomassiliicoccus, e Methanosarcina. Em sedimentos estuarinos, sequências de Methanosaeta, Methanobacterium, Methanoregula, e Methanoculleus foram comumente detectadas. Além disso, em sedimentos de lago, Methanoregula e Methanosaeta ocorreram principalmente. Em sedimentos marinhos, Methanoculleus e Methanosaeta são as linhagens mais comuns, seguidas por Methanolinea.
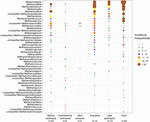
FIGURA 2. Gráfico de bolhas mostrando as frequências de incidência de linhagens metanogênicas em diferentes ambientes naturais. A ordem de classificação ao longo do eixo vertical corresponde à diminuição das freqüências de incidência total das linhagens. A taxonomia é mostrada para o nível do gênero. Se uma atribuição ao nível do gênero não foi possível, foi usado o próximo nível taxonômico atribuível mais alto. O número de amostras (n) é dado para cada habitat.
Even, embora muitos taxa foram detectados em ambientes diferentes, alguns ainda mostram preferências ambientais. Methanoregula, o taxon que ocorre frequentemente em ambientes não marinhos e de transição (solos, sedimentos lacustres e estuários), está ausente dos habitats marinhos (sedimentos marinhos, sedimentos hidrotermais e vulcões de lama). Methanobacterium e Methanocella, que prevalecem nos ambientes não marinhos e de transição, são raramente encontrados em habitats marinhos. Em contraste, Methanococcoides, como uma linhagem predominante em sedimentos marinhos, sedimentos hidrotermais e vulcões de lama, é pouco observado em solos e sedimentos lacustres. Além disso, Methanogenium e Methanolacinia só são observados em sedimentos estuarinos e marinhos, enquanto Methanospirillum e Methanosphaerula só são encontrados em ambientes terrestres. Além disso, alguns taxa específicos são exclusivamente encontrados em sedimentos hidrotermais, incluindo Methanocaldococcus, Methanothermococcus, Methanopyrus, Methanotorris, e Methanococcus. Embora algumas linhagens como Methanosaeta estejam presentes na maioria dos ambientes, nenhuma linhagem pode ser considerada omnipresente.
A maior riqueza de linhagens ocorreu em sedimentos estuarinos que também abrigam frequências de incidência mais uniformes de várias linhagens. Em contraste, vulcões de lama e ecossistemas hidrotermais apresentam uma diversidade metanogênica relativamente baixa. Solos e sedimentos lacustres similares a estuários são caracterizados por diversas montagens metanogênicas.
Alpha-Diversidade de Comunidades Metanogênicas em Ambientes Naturais
A riqueza de arcaicas metanogênicas de acordo com o índice Chao2 variou muito entre os tipos de ecossistemas (Figura 3). Com o propósito de comparar diretamente as diversidades alfa e para obter um trade-off razoável entre as amostras, foi realizada uma subamostragem de 15 seqüências para cada local. O índice de Chao2 mostra que os sedimentos estuarinos englobam a maior riqueza de espécies de metanogéneos ao longo dos seis tipos de ecossistemas (Tabela Complementar S3), o que sublinha os resultados do enredo da bolha (Figura 2). Os solos e sedimentos lacustres, mostrando menor riqueza do que as amostras estuarinas, têm índices de Chao2 significativamente mais altos do que os sedimentos marinhos e hidrotermais e sem diferenças significativas entre sedimentos marinhos e hidrotermais (Tabela Suplementar S3).
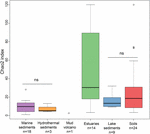
FIGURA 3. Quadro com os índices de Chao2 dos diferentes tipos de ecossistemas. O gráfico é baseado em conjuntos de dados subamostrados contendo 15 seqüências para cada local para tornar a comparação sobre as medidas de diversidade alfa mais robusta. O número de amostras em cada habitat é dado como ‘n’ por baixo do rótulo de habitat. Os ‘ns’ não têm significado estatístico no teste Wilcoxon. O resultado estatístico da diversidade alfa a nível de OTU é dado na Tabela Complementar S3.
Controles Globais sobre Comunidades Metanogênicas em Ambientes Naturais
As 94 comunidades metanogênicas distribuídas globalmente foram agrupadas em um gráfico de ordenação aplicando PCoA baseado na matriz de distância Jaccard. De acordo com a análise do PCoA, o primeiro e segundo eixos juntos explicam 16,3% da variância total. As variações entre as amostras podem assim ser em grande parte explicadas pela salinidade (Figura 4). Como os dados iniciais sobre as concentrações de sal não estavam disponíveis em alguns casos, atribuímos qualitativamente essas amostras como amostras salinas, mistas (intermediárias) e não salinas, como descrito acima. As amostras salinas e não-salinas separam-se efectivamente ao longo do primeiro eixo. As amostras mistas em geral agrupam-se entre as amostras salinas e não-salinas. A MANOVA permutacional baseada na matriz de distância Jaccard também sugere que a salinidade é o fator abiótico primário que controla a distribuição das comunidades metanogênicas globais (R2 = 0,099, P < 0,001) (Tabela 1).
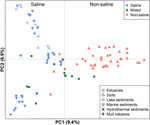
FIGURA 4. Ordenação de coordenadas principais (PCoA) com base na matriz de distância Jaccard de bibliotecas de sequências de genes mcrA metanogénicos comparando 94 amostras. A PCoA é colorida por salinidade: Os símbolos vermelhos indicam ambientes não salinos, os azuis indicam ambientes salinos, e os verdes indicam ambientes intermediários. Os símbolos diferentes descrevem ambientes diferentes. A percentagem da variação explicada pelas coordenadas principais plotadas é indicada nos eixos.

TABLE 1. Análise MANOVA permutacional na matriz de distância Jaccard de todas as amostras dos seis habitats para testar a associação da variância comunitária com diferentes variáveis ambientais.
Adicionalmente, verificamos uma relação potencial entre a fonte de isolamento de culturas metanogênicas puras e a concentração ótima de NaCl para crescimento. A concentração óptima de NaCl das culturas metanogénicas puras demonstrou um declínio desde os marinhos aos estuários, sedimentos lacustres e ecossistemas do solo (Figura Complementar S2). Alguns isolados externos originam-se de sedimentos de lagos de soda ou solos hipersalinos.
Controle Ambiental e Indicador Metanogênico Taxa em Solos Não-Salinos e Sedimentos do Lago
A uma escala global, as comunidades metanogênicas de solos não-salinos e sedimentos lacustres se agrupam de perto (Figura 4) de modo que analisamos mais detalhadamente os controles ambientais das comunidades metanogênicas desses dois habitats que representam 33 locais de estudo no total. A análise de clusters baseada na comunidade para estes dois tipos de habitats revelou quatro clusters baseados na matriz de distância Jaccard (Figura S3 Suplementar). MANOVA permutacional sugere que tanto o pH (R2 = 0,099, P < 0,001) quanto a temperatura (R2 = 0,069, P < 0,001) influenciam o metanogênico β-diversidade em solos não salinos e sedimentos lacustres (Tabela 2). Consequentemente, atribuímos os quatro grupos ao pH e MAAT do local inicial da amostragem e obtivemos subgrupos amplamente consistentes para o agrupamento da comunidade (Figuras 5C,D). A combinação das características ambientais e esses quatro grupos de comunidade nos permite definir esses quatro subgrupos como grupo neutro e frio 1, grupo ácido e frio 2, grupo ácido e moderado 3, e grupo neutro e quente 4 (Figura 5). Uma ordenação PCoA adicional baseada na matriz de diferenças de Jaccard sugere que ao longo do eixo PC1, a maioria das amostras dos grupos 2 e 3 são de solos ácidos e sedimentos lacustres enquanto que os grupos 1 e 4 eram principalmente de ambientes neutros (Figura 5A). Além disso, amostras de locais moderados (grupo 3) foram separadas das amostras de locais quentes e frios ao longo do PC2, enquanto que as amostras dos ambientes quentes (grupo 4) foram separadas das outras amostras ao longo do PC3 (Figura 5B). Assim, os três primeiros eixos da ordenação PCoA explicam 38,8% da variação total.

TABLE 2. MANOVA permutacional baseada em uma matriz de distância Jaccard de amostras de solo não salino e sedimentos lacustres para testar a associação da variância comunitária com diferentes variáveis ambientais.
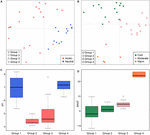
FIGURA 5. Gráfico de análise de coordenadas principais baseado em uma matriz de distância Jaccard para 33 solos não salinos e sedimentos lacustres. Subplot (A) mostra PC1 e PC2 e os símbolos são coloridos por pH, e o subplot (B) mostra PC2 e PC3 e os símbolos são coloridos por temperatura. Os três primeiros componentes explicam 17,1, 11,7, e 10% da variância. Os gráficos da caixa mostram o pH (C) e MAAT (temperatura média anual do ar) (D) de quatro subgrupos identificados. A cor da caixa na figura C corresponde à categoria de pH em (A). As cores dos gráficos de caixa mostram significância estatística baseada em um teste Wilcoxon pareado (P < 0,05), onde as amostras com a mesma cor não diferem significativamente umas das outras. Da mesma forma, a cor em (D) segue o agrupamento de temperatura em (B). Os subgrupos referem-se à análise de agrupamento hierárquico das similaridades da comunidade.
Examinamos a ocorrência de linhagens metanogênicas em cada subgrupo com base nas frequências de incidência (Figura 6). A metanoregula prevalece em todos os tipos de habitats não salinos. Além da Methanoregula, o subgrupo neutro e frio (grupo 1) exibe uma alta abundância de Methanosaeta, Methanobacterium, e Methanosarcina. O grupo ácido e frio 2 é representado por Methanobacterium, Methanocella, e Methanosarcina enquanto Methanosaeta está ausente aqui. Methanocella e Methanosaeta são comuns no grupo ácido e moderado 3. No grupo neutro e quente 4, Methanolinea e Methanosaeta são membros importantes. Este grupo é o único grupo onde Methanoculleus foi identificado.

FIGURA 6. Frequências de incidência de linhagens metanogênicas dentro dos quatro grupos definidos de comunidades metanogênicas em solos e sedimentos lacustres. O eixo vertical está disposto em ordem alfabética. O comprimento da barra corresponde às frequências médias de incidência para cada linhagem dentro do grupo correspondente. As barras de erro representam o desvio padrão de um dado taxon sobre diferentes amostras desse grupo. Os asteriscos mostram os táxons especializados com valor de P< 0,05. A taxonomia é mostrada para o nível do gênero. Se uma atribuição ao nível do gênero não foi possível, foi utilizado o próximo nível taxonômico atribuível mais alto. A descrição para os quatro grupos é dada na Figura 5.
Para todos os quatro grupos, os táxons com alta incidência são Methanoregula, Methanobacterium, Methanosarcina, Methanosaeta, Methanomassiliicoccus, e Methanocella. Os taxa especializados, que são significativamente mais representados na maioria dos locais dentro de um determinado grupo, foram detectados de acordo com a análise de espécies indicadoras, como descrito anteriormente. No total, seis dos 31 taxa mostraram um valor indicador significativo (P < 0,05) (rotulado com asterisco na Figura 6). O grupo 1 (neutro e frio) mostrou o maior número de especialistas com linhagens de Methanosaeta, Methanolobus e Methanometilovorans. Methanobacterium serviu como um taxon especialista no grupo 2 frio e ácido enquanto Methanolinea foi identificado como um especialista no grupo 4 (neutro e quente) mas dificilmente foi observado em outros grupos. Além disso, Methanoregula é largamente representada no grupo ácido e moderado 3,
Dispersal Limitation
A análise de regressão linear (R2= 0,05, P < 0,001) indicou uma fraca correlação entre distância geográfica e estrutura da comunidade metanogénica no conjunto de dados global. Ao mesmo tempo, um teste de Mantel mostrou que as variáveis ambientais têm uma correlação maior com a estrutura da comunidade do que as distâncias geográficas (ver Tabela 3). Esta tendência também é confirmada por um teste parcial de Mantel que controla os efeitos da autocorrelação. O traçado da distância geográfica contra a semelhança da comunidade Jaccard não mostra uma tendência linear clara, mas padrões que resultam principalmente da distribuição global dos pontos de amostragem (ver Figura Suplementar S4).

TABLE 3. Análises de teste de Mantel e Mantel parcial para a determinação da influência das variáveis ambientais e distância geográfica sobre a distribuição microbiana para o conjunto de dados global e uma subamostra de 16 amostras europeias.
Para aprofundar a análise da influência da dispersão, limitámos a nossa análise à Europa, que foi a mais densa e mais homogénea das amostras. Os testes Mantel e os testes Mantel parciais neste subconjunto reproduziram a tendência de que os dados da comunidade estão mais correlacionados com as variáveis ambientais do que com as distâncias geográficas (ver Tabela 3). Os testes parciais de Mantel para controle de variáveis ambientais não conseguiram detectar nenhuma correlação estatisticamente significativa entre a comunidade microbiana e a distância geográfica. Uma análise PCA espacial nestes 16 locais europeus implica uma estrutura espacial da comunidade metanogênica (23,7% da variância total foi explicada por esta estrutura) que corresponde a uma autocorrelação espacial positiva dos locais como indicado pelo índice I de Moran (Moran’s I = 0,4018). Apenas o primeiro valor próprio foi estável e correspondeu a uma separação das amostras entre a Europa Central e os Estados Bálticos (Figura Suplementar S5). O pequeno conjunto de dados, entretanto, complica uma atribuição robusta desta estrutura espacial observada a variáveis geográficas, ambientais ou a ambas. Assim, realizamos uma análise de cluster sobre as comunidades metanogênicas como descrito acima e revelamos três grupos (Figura 7A) que projetamos em um mapa geográfico (Figura 7B). O agrupamento não reproduziu a separação do PCA espacial ao longo do Mar Báltico. Consequentemente, alguns locais que estão geograficamente muito próximos uns dos outros exibem estruturas comunitárias metanogénicas diferentes e reúnem-se com diferentes aglomerados. Por outro lado, alguns locais geograficamente muito distantes apresentam composições comunitárias muito semelhantes e agrupam-se em conjunto (Figura 7B). A dispersão regional dos grupos na Europa não parece corresponder ou ser limitada por uma estrutura geográfica.
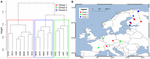
FIGURA 7. Análise hierárquica da matriz de distância Jaccard entre 16 amostras de solo e sedimentos lacustres europeus. O método Ward clustering foi utilizado para a análise. Os locais foram divididos em três grupos coloridos por diferentes retângulos em dendrograma de agrupamento (A) e foram projetados em um mapa europeu (B).
Discussão
Identificar e aplicar conceitos de biogeografia em comunidades microbianas é de grande interesse na ecologia microbiana. Acredita-se que a biogeografia microbiana é governada pela interação evolutiva e ecológica de quatro processos principais: filtragem de habitat, dispersão, deriva e mutação (Hanson et al., 2012). Embora a influência da deriva e da mutação esteja além do poder de interpretação deste estudo, mostramos que existem padrões globais de comunidades metanogênicas em ambientes naturais. Este estudo demonstra um padrão biogeográfico global de comunidades metanogênicas que está mais associado à filtragem do habitat do que à dispersão geográfica. Comunidades metanogênicas de sedimentos de lagos de soda, por exemplo, aglomeram-se estreitamente com amostras marinhas geograficamente distantes (Figura 4) e comunidades metanogênicas muito semelhantes ocorrem em solos e sedimentos de lagos europeus apesar de localizados em grande distância entre si (Figura 7). Em geral, nossas tentativas de revelar uma potencial limitação de dispersão revelaram uma fraca influência da localização geográfica na estrutura da comunidade metanogênica, o que contrasta com uma clara influência das condições ambientais. Uma exclusão conclusiva dos efeitos espaciais nas comunidades microbianas não é possível com os pontos de dados disponíveis. Os pontos de amostragem concentram-se em determinados habitats e/ou áreas enquanto o número total de amostras é baixo. Se houver um efeito, assumimos que os efeitos espaciais ocorrem apenas em escala regional ou local. Foram relatadas limitações de dispersão local para arca hipertermófila causadas por barreiras geográficas (Whitaker et al., 2003), bactérias amonia-oxidantes em pântanos salinos locais, mas não em escalas regionais e continentais (Martiny et al., 2011), microorganismos em sedimentos de águas profundas juntamente com ambientes ambientais (Schauer et al.2009), e para as bactérias de um grande conjunto de ambientes de neve heterogêneos causados principalmente pela disponibilidade de carbono alóctone (Lutz et al., 2016). Uma pequena influência da limitação da dispersão de metanogéneos em ambientes naturais significa que os metanogéneos podem distribuir-se aleatoriamente pelo espaço, são seleccionados com sucesso pelo ambiente local se os seus requisitos fisiológicos forem satisfeitos e podem estabelecer comunidades estáveis (Martiny et al., 2006; von Mering et al., 2007).
O conceito de filtragem de habitats implica que espécies com requisitos ecológicos semelhantes devem co-ocorrer mais frequentemente do que o esperado por acaso (Weiher e Keddy, 2001; Cornwell et al., 2006; Ulrich et al., 2010). Nosso resultado mostra que grandes diferenças entre a composição da comunidade metanogênica ocorrem entre ecossistemas marinhos e de zonas úmidas e lagos, enquanto os estuários se agrupam entre si. Foram também relatados padrões de biogeografia baseados num conjunto de levantamentos genéticos em amostras ambientais para bactérias em geral (Nemergut et al., 2011), o microrganismo metano do mar (Ruff et al., 2015), o arquebactéria amonia-oxidante (Cao et al., 2013), as bactérias pelágicas e bentónicas marinhas (Zinger et al., 2011), e os microrganismos cicladores de azoto (Church et al., 2008). A filtragem de habitats foi especificamente relatada para arcaicas não cultivadas (Auguet et al., 2010), comunidades bacterianas inteiras em ambientes diversos (von Mering et al., 2007; Chaffron et al., 2010) ou em turfeiras sul-americanas em escala regional (Oloo et al., 2016), bem como grupos bacterianos específicos, tais como bactérias metano-oxidantes (Knief, 2015) e bactérias fixadoras de azoto (Nelson et al., 2016).
Os nossos resultados indicam que, à escala global, a salinidade regula substancialmente a composição da comunidade metanogénica e determina grandes diferenças entre os conjuntos metanogénicos marinhos e terrestres. Também os metanogénicos de sedimentos de lagos de soda se agrupam com os de sedimentos marinhos (Figura 4) destacando a influência global da salinidade. Este resultado está de acordo com outros estudos baseados no gene 16S rRNA revelando que a salinidade é um fator primário modelando padrões globais de comunidades bacterianas e arqueológicas em geral (Lozupone e Knight, 2007; Auguet et al., 2010; Caporaso et al., 2011; Cao et al., 2013). Uma baixa influência da separação geográfica mas um forte impacto da salinidade nas comunidades microbianas em geral também foi observada em estudos anteriores (Logares et al., 2013; Yang et al., 2016). Assim, a salinidade determina em grande parte quais linhagens podem sobreviver. Em vários habitats, a atividade de produção de metano foi negativamente correlacionada com a salinidade (Bartlett et al., 1987; Potter et al., 2009; Poffenbarger et al., 2011). A inibição da produção de metano através da salinidade é assim sugerida para coincidir com uma população metanogénica reduzida (Pattnaik et al., 2000). O efeito da salinidade na metanogênese hidrogeno-trófica, acetotrófica e metilotrófica depende do nível de salinidade e é diferente para as diferentes vias de metanogênese (Liu et al., 2016). Atualmente, não há um mecanismo claro para explicar o impacto da salinidade na estrutura da comunidade, mas várias hipóteses podem servir como explicação possível. Fisiologicamente, a salinidade influencia a osmolaridade externa e interna das células. As células metanogênicas não salinas desenvolveram adaptações fisiológicas para combater a pressão turgorífica interna, enquanto as células adaptadas ao sal perderam tal característica (Zinder, 1993). Além disso, o aumento da salinidade pode induzir os metanogénicos a sintetizar ou tomar uma maior proporção de solutos compatíveis a um custo energético e, portanto, metabólico significativo (McGenity, 2010). O traço de tolerância ao sal é mesmo manifestado na concentração ótima de NaCl para o crescimento de culturas metanogênicas puras desde que descobrimos que os isolados dos sedimentos marinhos e sedimentos hidrotermais têm concentração ótima de NaCl significativamente mais alta do que aqueles dos solos (Figura Complementar S2).
Nos ecossistemas terrestres não salinos, especificamente nos solos e sedimentos lacustres, a composição da comunidade metanogênica é controlada pela combinação de temperatura e pH. Assim, os metanogênicos desses ambientes poderiam ser classificados em quatro grupos (Figura 6). Ao contrário dos ecossistemas marinhos, os ecossistemas terrestres não salinos mostram uma grande variabilidade natural tanto de pH como de temperatura. A temperatura pode afectar não só a via metanogénica mas também as próprias populações metanogénicas (Conrad, 2007; Rooney-Varga et al., 2007). A produção de metano pode ser muito aumentada se as temperaturas subirem como consequência das etapas sensíveis à temperatura durante a fermentação e a acetogénese (Megonigal et al., 2005; Kotsyurbenko et al., 2007). Além disso, o baixo pH pode limitar substancialmente a disponibilidade do acetato, impedindo que o acetato se dissocie e, assim, afete negativamente a metanogênese acetoclástica (Fukuzaki et al., 1990; Bridgham et al., 2013). Esta poderia ser uma possível razão para a ausência de Methanosaeta no grupo2, enquanto a Methanosarcina pode alternar entre diferentes fontes e não foi substancialmente influenciada. Além disso, o pH pode regular a eficiência da produção de metano e das vias metanogênicas de turfas ombrotróficas a minerotróficas, através da inibição direta de ambas as vias de metanogênese e indiretamente através de seus efeitos na fermentação (Ye et al., 2012). Portanto, tanto a temperatura como o pH podem regular directa ou indirectamente as etapas metabólicas associadas à metanogénese e à fermentação a montante, que fornece substrato para os metanogéneos.
Methanoregula é omnipresente e muito abundante nos quatro grupos de habitats terrestres (Figura 6) mas virtualmente ausente do sistema marinho e pode assim revelar-se um substituto para a influência da água doce no reino marinho. A sua relevância global foi recentemente reportada noutros locais (Yang et al., 2017). Apesar da sua distribuição omnipresente nos solos e sedimentos lacustres, a Methanoregula ocorre como uma linhagem indicadora em habitats ácidos com temperaturas moderadas. Além disso, (1) Methanolinea parece ter-se adaptado particularmente aos ambientes terrestres quentes e neutros, (2) Methanobacterium aos ambientes frios e ácidos, e (3) Methanosaeta aos ambientes pH neutros, o que é consistente com outros estudos (Rosenberg et al., 2014) e sublinha a robustez da nossa abordagem. Geralmente, as condições geoquímicas que envolvem as comunidades metanogênicas levarão à diferenciação de nichos de mercado. Uma vez que a classificação dos nichos tende a deixar os especialistas adaptativos (Langenheder e Székely, 2011), a seleção ambiental progressiva a longo prazo gerou uma variedade de nichos que foram preenchidos por um conjunto de especialistas em habitats endêmicos, que podem estar menos representados ou ausentes em outras condições ambientais diferentes. A comunidade também é moldada por factores bióticos, tais como interacções ecológicas, dinâmicas, competição e simbiose. Apesar desses fatores biológicos, von Mering et al. (2007) descobriram que as preferências de habitat são freqüentemente notavelmente estáveis ao longo do tempo e a composição taxonômica distintiva das comunidades ambientais, por sua vez, pode ser um indicador importante de sua ecologia e função.
Consistente com a preferência de habitat da arca metanogênica, parece que linhagens metanogênicas intimamente relacionadas foram freqüentemente isoladas de ambientes comparáveis. Por exemplo, Methanoregulaceae parece ser bastante diversificada em ambientes naturais (Yang et al., 2017) e as cepas neutras podem resistir ao culto até agora. As cepas actualmente descritas de Methanoregula são ambas obtidas de ambientes ligeiramente acidófilos, enquanto os dois representantes da Methanolinea são de habitats relativamente quentes, tais como lodos de digestores e solo de campos de arroz, respectivamente (Rosenberg et al., 2014). Embora as linhagens de Methanobacterium foram isoladas de vários ambientes, aproximadamente metade dos isolados existentes deste género exibem pH optima ligeiramente inferior a 7. Isto significa que as linhagens indicadoras, que foram identificadas com base nas sequências ambientais, poderiam reflectir as diferenciações de fisiologia e fontes das cultivares metanogénicas existentes. Por exemplo, a salinidade do habitat, como propriedade geral do habitat, pode progressivamente expor os organismos a uma forte selecção ambiental e filtrar a montagem de um novo conjunto de espécies mais adequadas à salinidade ambiente (Logares et al., 2013).
A conservação e gestão da biodiversidade é um desafio primordial da nossa sociedade actual. Aqui mostramos que a arca metanogénica dos ambientes naturais é mais diversificada nos sedimentos dos estuários. Os estuários são zonas de transição entre os ecossistemas marinhos e terrestres. Isto permite dois grandes processos que podem contribuir para a riqueza das espécies. Em primeiro lugar, os micróbios do mar e da terra misturam-se nos estuários e acabam por abranger uma grande diversidade (McLusky e Elliott, 2004). Por exemplo, a alta diversidade de bactérias estuarinas, archaea, fungos e até mesmo bactérias específicas que desempenham funções únicas foram observadas (Cunliffe et al., 2008; Mosier e Francis, 2008; Crump et al., 2012). Outro aspecto é o alto nível de nutrientes devido às entradas de marés terrestres para os organismos estuarinos se alimentarem (McLusky e Elliott, 2004; Statham, 2012). Neste contexto, os ambientes estuarinos são importantes na recuperação da novidade genérica dos metanogéneos. Até agora, os efeitos da diversidade de espécies nos processos dos ecossistemas têm atraído esforços substanciais de pesquisa. A ligação entre biodiversidade e função do ecossistema ainda está em debate e permanece elusiva para as comunidades microbianas (Loreau et al., 2001; Tilman et al., 2014). Embora os solos e sedimentos lacustres sejam fontes primárias de metano e também habitats com elevada diversidade metanogénica, propomos que a riqueza das espécies não é uma proxy apropriada do potencial de produção de metano e das emissões de metano dos ecossistemas; parece antes reflectir a heterogeneidade e a história do ambiente. Classificar os ambientes de acordo com a riqueza das suas espécies não significa necessariamente o potencial das taxas de emissão de metano que são mais elevadas dos solos e lagos e comparativamente menores dos estuários (Figura 3 e Figura Suplementar S1).
Finalmente, a falta de informação ambiental nas bases de dados públicas pode ter dificultado uma interpretação completa sobre os factores ambientais aqui observados. Embora existam dados seqüenciais crescentes sobre metanogênicos na literatura e bancos de dados públicos, as variáveis abióticas relacionadas fornecidas são frequentemente inconsistentes e esparsas. A quantidade limitada de informação consistente para as variáveis ambientais restringe a aplicação de análises estatísticas multi-variadas. Lembrando que os fatores abióticos neste estudo só podem explicar uma fração limitada das variâncias da comunidade sugere que outras variáveis explicativas estão faltando. De particular importância poderiam ser as concentrações e a disponibilidade de substratos metanogênicos como acetato, hidrogênio e metilaminas. No entanto, os parâmetros abióticos podem nunca ser suficientes para explicar completamente quais as estruturas de conjuntos metanogênicos simplesmente porque os habitats têm histórias diferentes e só podem ser estudados localmente. Além disso, entre os dados mcrA disponíveis, as vastas áreas da subárctica russa e canadense e do Ártico estão mal representadas. Uma melhor cobertura geográfica e mesmo distribuição do conjunto de dados do gene mcrA melhoraria uma avaliação das comunidades metanogênicas em escala global.
Author Contributions
SL e XW projetaram o estudo. XW e SY coletaram e analisaram os dados. XW, SY, e FH realizaram a análise estatística. XW, MW, e SY fizeram a correção filogenética. XW, SY, FH, MW, DW, e SL interpretaram os resultados e escreveram o trabalho. Todos os autores contribuíram para as discussões e revisaram o manuscrito.
Conflict of Interest Statement
Os autores declaram que a pesquisa foi conduzida na ausência de quaisquer relações comerciais ou financeiras que pudessem ser interpretadas como um potencial conflito de interesses.
Acknowledgments
O apoio financeiro para XW (Subsídio No. 201408620031 para XW) fornecido pelo China Scholarship Council (CSC) é reconhecido com gratidão. Este estudo foi apoiado pelo Helmholtz Gemeinschaft (HGF) através do financiamento do Helmholtz Young Investigators Group of SL (VH-NG-919).
Material Suplementar
O Material Suplementar para este artigo pode ser encontrado online em: https://www.frontiersin.org/article/10.3389/fmicb.2017.01339/full#supplementary-material
Pés
- ^ http://metanogen.biotech.uni.wroc.pl/
- ^ http://www.ncbi.nlm.nih.gov/
- ^ http://metanogen.biotech.uni.wroc.pl/
- ^ https://www.ngdc.noaa.gov/mgg/shorelines/gshhs.html
- ^ http://qgis.osgeo.org
Auguet, J. C, Barberan, A., e Casamayor, E. O. (2010). Padrões ecológicos globais na Archaea não cultivada. ISME J. 4, 182-190. doi: 10.1038/ismej.2009.109
PubMed Abstract | CrossRef Full Text | Google Scholar
Bange, H. W., Bartell, U. H., Rapsomanikis, S., and Andreae, M. O. (1994). Metano no Mar Báltico e Mar do Norte e uma reavaliação das emissões marinhas de metano. Globo. Biogeochem. Ciclos 8, 465-480. doi: 10.1029/94GB02181
CrossRef Full Text | Google Scholar
Barreto, D. P., Conrad, R., Klose, M., Claus, P., e Enrich-Prast, A. (2014). Relações distância-decaia e taxa-área para bactérias, arcaia e arcaia metanogênica em um sedimento de lago tropical. PLoS ONE 9:e110128. doi: 10.1371/journal.pone.0110128
PubMed Abstract | CrossRef Full Text | Google Scholar
Bartlett, K. B., Bartlett, D. S., Harriss, R. C., e Sebacher, D. I. (1987). Emissões de metano ao longo de um gradiente de salinidade de pântano salgado. Biogeoquímica 4, 183-202. doi: 10.1007/BF02187365
CrossRef Full Text | Google Scholar
Borrel, G., O’Toole, P. W., Harris, H. M. B., Peyret, P., Brugere, J. F., e Gribaldo, S. (2013). Os dados filogenómicos suportam uma sétima ordem de metanogéneos metilotróficos e fornecem insights sobre a evolução da metanogénese. Biol do Genoma. Evol. 5, 1769-1780. doi: 10.1093/Gbe/Evt128
PubMed Abstract | CrossRef Full Text | Google Scholar
Bousquet, P., Ciais, P., Miller, J., Dlugokencky, E., Hauglustaine, D., Prigent, C., et al. (2006). Contribuição de fontes naturais e antropogénicas para a variabilidade do metano atmosférico. Nature 443, 439-443. doi: 10.1038/nature05132
PubMed Abstract | CrossRef Full Text | Google Scholar
Bridgham, S. D., Cadillo-Quiroz, H., Keller, J. K., and Zhuang, Q. (2013). Emissões de metano de zonas húmidas: perspectivas biogeoquímicas, microbianas e de modelação, da escala local à global. Globo. Chang. Biol. 19, 1325-1346. doi: 10.1111/gcb.12131
PubMed Abstract | CrossRef Full Text | Google Scholar
Cao, H., Auguet, J.-C., e Gu, J.-D. (2013). Padrão ecológico global de arquebactérias amoniacais oxidantes. PLoS ONE 8:e52853. doi: 10.1371/journal.pone.0052853
PubMed Abstract | CrossRef Full Text | Google Scholar
Caporaso, J. G., Lauber, C. L., Walters, W. A., Berg-Lyons, D., Lozupone, C. A., Turnbaugh, P. J., et al. (2011). Padrões globais de 16S rRNA diversidade a uma profundidade de milhões de sequências por amostra. Proc. Natl. Acad. Sci. U.S.A. 108, 4516-4522. doi: 10.1073/pnas.1000080107
PubMed Abstract | CrossRef Full Text | Google Scholar
Chaffron, S., Rehrauer, H., Pernthaler, J., e von Mering, C. (2010). Uma rede global de micróbios coexistentes a partir de dados ambientais e de sequências de todo o genoma. Genome Res. 20, 947-959. doi: 10.1101/gr.104521.109
PubMed Abstract | CrossRef Full Text | Google Scholar
Church, M. J., Bjorkman, K. M., Karl, D. M., Saito, M. A., e Zehr, J. P. (2008). Distribuições regionais de bactérias fixadoras de nitrogênio no Oceano Pacífico. Limnol. Oceanogr. 53, 63-77. doi: 10.4319/lo.2008.53.1.0063
CrossRef Full Text | Google Scholar
Conrad, R. (2007). “Microbial ecology of methanogens and methanotrophs”, em Advances in Agronomy, ed. L. S. Donald (Cambridge, MA: Academic Press), 1-63.
Google Scholar
Cornwell, W. K., Schwilk, D. W., e Ackerly, D. D. (2006). Um teste baseado em características para filtragem de habitat: volume convexo do casco. Ecology 87, 1465-1471. doi: 10.1890/0012-9658(2006)872.0.CO;2
PubMed Abstract | CrossRef Full Text | Google Scholar
Crump, B. C., Amaral-Zettler, L. A., e Kling, G. W. (2012). A diversidade microbiana em águas doces árticas é estruturada pela inoculação de micróbios de solos. ISME J. 6, 1629-1639. doi: 10.1038/ismej.2012.9
PubMed Abstract | CrossRef Full Text | Google Scholar
Cui, M., Ma, A., Qi, H., Zhuang, X., Zhuang, G., and Zhao, G. (2015). A temperatura mais quente acelera as emissões de metano da zona húmida de Zoige no Planalto Tibetano sem alterar a composição da comunidade metanogénica. Sci. Rep. 5:11616. doi: 10.1038/srep11616
PubMed Abstract | CrossRef Full Text | Google Scholar
Cunliffe, M., Schafer, H., Harrison, E., Cleave, S., Upstill-Goddard, R., and Murrell, J. C. (2008). Análise filogenética e funcional das comunidades bacterianas e arqueológicas associadas à micro-camada superficial de um estuário. ISME J. 2, 776-789. doi: 10.1038/ismej.2008.28
PubMed Abstract | CrossRef Full Text | Google Scholar
Dray, S., and Dufour, A. B. (2007). O pacote ade4: implementando o diagrama de dualidade para ecologistas. J. Stat. Softw. 22, 1-20. doi: 10.18637/jss.v022.i04
CrossRef Full Text | Google Scholar
Dufrene, M., and Legendre, P. (1997). Species assemblages and indicator species: the need for a flexible asymmetrical approach. Ecol. Monogr. 67, 345-366. doi: 10.2307/2963459
CrossRef Full Text | Google Scholar
Edgar, R. C., Haas, B. J., Clemente, J. C., Quince, C., e Knight, R. (2011). UCHIME melhora a sensibilidade e a velocidade de detecção de quimeras. Bioinformática 27, 2194-2200. doi: 10.1093/bioinformática/btr381
PubMed Abstract | CrossRef Full Text | Google Scholar
Evans, P. N., Parks, D. H., Chadwick, G. L., Robbins, S. J., Orphan, V. J., Golding, S. D., et al. (2015). Metabolismo do metano no filo archéico Bathyarchaeota revelado pela metagenómica centrada no genoma. Science 350, 434-438. doi: 10.1126/science.aac7745
PubMed Abstract | CrossRef Full Text | Google Scholar
Fish, J. A., Chai, B., Wang, Q., Sun, Y., Brown, C. T., Tiedje, J. M., et al. (2013). FunGene: o gasoduto e repositório de genes funcionais. Frente. Microbiol. 4:291. doi: 10.3389/fmicb.2013.00291
PubMed Abstract | CrossRef Full Text | Google Scholar
Frank-Fahle, B. A., Yergeau, E., Greer, C. W., Lantuit, H., e Wagner, D. (2014). Potencial funcional microbiano e composição da comunidade em solos permafrost-affectados do Noroeste do Ártico Canadense. PLoS ONE 9:e84761. doi: 10.1371/journal.pone.0084761.g001
PubMed Abstract | CrossRef Full Text | Google Scholar
Fukuzaki, S., Nishio, N., and Nagai, S. (1990). Cinética da fermentação metanogénica do acetato. Aplic. Ambiente. Microbiol. 56, 3158-3163.
Google Scholar
Hanson, C. A., Fuhrman, J. A., Horner-Devine, M. C., e Martiny, J. B. (2012). Para além dos padrões biogeográficos: processos que moldam a paisagem microbiana. Nat. Rev. Microbiol. 10, 497-506. doi: 10.1038/Nrmicro2795
PubMed Abstract | CrossRef Full Text | Google Scholar
Holm, G. O., Perez, B. C., McWhorter, D. E., Krauss, K. W., Johnson, D. J., Raynie, R. C., et al. (2016). Ecosystem level methane flowes from tidal freshwater and brackish marshes of the Mississippi River Delta: implications for coastal wetland carbon projects. Wetlands 36, 401-413. doi: 10.1007/s13157-016-0746-7
CrossRef Full Text | Google Scholar
Hunger, S., Schmidt, O., Hilgarth, M., Horn, M. A., Kolb, S., Conrad, R., et al. (2011). Formato concorrente – e dióxido de carbono – utilizando procariotas em um solo de feno emissor de metano anóxico. Aplicar. Ambiente. Microbiol. 77, 3773-3785. doi: 10.1128/AEM.00282-11
PubMed Abstract | CrossRef Full Text | Google Scholar
IPCC (2007). “Climate Change 2007: the Physical Science Basis, em Proceedings of the Contribution of Working Group I to the Fourth Assessment Report of the Intergovernmental Panel on Climate Change, eds D. Qin, S. Solomon, M. Manning, Z. Chen, M. Marquis, K. B. Averyt, et al. (Cambridge: Cambridge University Press).
Google Scholar
Jabłoński, S., Rodowicz, P., e Łukaszewicz, M. (2015). Base de dados de arcaicas metanogênicas contendo características fisiológicas e bioquímicas. Int. J. Syst. Evol. Microbiol. 65, 1360-1368. doi: 10.1099/ijs.0.000065
PubMed Abstract | CrossRef Full Text | Google Scholar
Kirschke, S., Bousquet, P., Ciais, P., Saunois, M., Canadell, J. G., Dlugokencky, E. J., et al. (2013). Três décadas de fontes e sumidouros globais de metano. Nat. Geosci. 6, 813-823. doi: 10.1038/Ngeo1955
CrossRef Full Text | Google Scholar
Knief, C. (2015). Diversidade e preferências de habitat de bactérias metanotróficas aeróbicas cultivadas e não cultivadas avaliadas com base no pmoA como marcador molecular. Frente. Microbiol. 6:1346. doi: 10.3389/fmicb.2015.01346
PubMed Abstract | CrossRef Full Text | Google Scholar
Kotsyurbenko, O., Friedrich, M., Simankova, M., Nozhevnikova, A., Golyshin, P., Timmis, K., et al. (2007). Mudança de acetoclástico para metanogênese dependente de H2 em uma turfeira da Sibéria Ocidental com baixos valores de pH e isolamento de uma cepa acidófila de Methanobacterium. Aplic. ambiente. Microbiol. 73, 2344-2348. doi: 10.1128/AEM.02413-06
PubMed Abstract | CrossRef Full Text | Google Scholar
Langenheder, S., e Székely, A. J. (2011). A separação das espécies e os processos neutros são ambos importantes durante a montagem inicial das comunidades bacterianas. ISME J. 5, 1086-1094. doi: 10.1038/ismej.2010.207
PubMed Abstract | CrossRef Full Text | Google Scholar
Liebner, S., Ganzert, L., Kiss, A., Yang, S., Wagner, D., e Svenning, M. M. (2015). Mudanças na composição da comunidade metanogênica e fluxos de metano ao longo da degradação do permafrost descontínuo. Frente. Microbiol. 6:356. doi: 10.3389/fmicb.2015.00356
PubMed Abstract | CrossRef Full Text | Google Scholar
Liu, Y. Q., Priscu, J. C., Xiong, J. B., Conrad, R., Vick-Majors, T., Chu, H. Y., et al. (2016). A salinidade impulsiona os padrões de distribuição arqueológica nos sedimentos dos lagos de alta altitude no planalto tibetano. FEMS Microbiol. Ecol. 92:fiw033. doi: 10.1093/femsec/fiw033
PubMed Abstract | CrossRef Full Text | Google Scholar
Logares, R., Lindstrom, E. S., Langenheder, S., Logue, J. B., Paterson, H., Laybourn-Parry, J., et al. (2013). Biogeografia de comunidades bacterianas expostas a mudanças ambientais progressivas a longo prazo. ISME J. 7, 937-948. doi: 10.1038/ismej.2012.168
PubMed Abstract | CrossRef Full Text | Google Scholar
Loreau, M., Naeem, S., Inchausti, P., Bengtsson, J., Grime, J. P., Hector, A., et al. (2001). Biodiversidade e funcionamento dos ecossistemas: conhecimentos actuais e desafios futuros. Science 294, 804-808. doi: 10.1126/science.1064088
PubMed Abstract | CrossRef Full Text | Google Scholar
Lozupone, C. A., e Knight, R. (2007). Padrões globais em diversidade bacteriana. Proc. Natl. Acad. Sci. U.S.A. 104, 11436-11440. doi: 10.1073/pnas.0611525104
PubMed Abstract | CrossRef Full Text | Google Scholar
Luton, P. E., Wayne, J. M., Sharp, R. J., and Riley, P. W. (2002). O gene mcrA como alternativa ao 16S rRNA na análise filogenética de populações de metanogênio em aterros sanitários. Microbiologia 148, 3521-3530. doi: 10.1099/00221287-148-11-3521
PubMed Abstract | CrossRef Full Text | Google Scholar
Lutz, S., Anesio, A. M., Raiswell, R., Edwards, A., Newton, R. J., Gill, F., et al. (2016). A biogeografia dos microbiomas da neve vermelha e o seu papel no derretimento dos glaciares árcticos. Nat. Commun. 7:11968. doi: 10.1038/ncomms11968
PubMed Abstract | CrossRef Full Text | Google Scholar
Martiny, J. B. H., Bohannan, B. J. M., Brown, J. H., Colwell, R. K., Fuhrman, J. A., Green, J. L., et al. (2006). Biogeografia microbiana: colocando microrganismos no mapa. Nat. Rev. Microbiol. 4, 102-112. doi: 10.1038/Nrmicro1341
PubMed Abstract | CrossRef Full Text | Google Scholar
Martiny, J. B. H., Eisen, J. A., Penn, K., Allison, S. D., e Horner-Devine, M. C. (2011). Os condutores de beta-diversidade bacteriana dependem da escala espacial. Proc. Natl. Acad. Sci. U.S.A. 108, 7850-7854. doi: 10.1073/pnas.1016308108
PubMed Abstract | CrossRef Full Text | Google Scholar
McArdle, B. H., and Anderson, M. J. (2001). Adaptando modelos multivariados aos dados da comunidade: um comentário sobre a análise de redundância baseada na distância. Ecology 82, 290-297. doi: 10.1890/0012-9658(2001)0822.0.CO;2
CrossRef Full Text | Google Scholar
McCalley, C. K., Woodcroft, B. J., Hodgkins, S. B., Wehr, R. A., Kim, E.-H., Mondav, R., et al. (2014). Dinâmica do metano regulada pela resposta da comunidade microbiana ao degelo do permafrost. Nature 514, 478-481. doi: 10.1038/nature13798
PubMed Abstract | CrossRef Full Text | Google Scholar
McGenity, T. J. (2010). “Methanogens and Methanogenesis in Hypersaline Environments,” in Handbook of Hydrocarbon and Lipid Microbiology, ed. K. N. Timmis (Berlin: Springer), 665-680. doi: 10.1007/978-3-540-77587-4_53
CrossRef Full Text | Google Scholar
McLusky, D. S., e Elliott, M. (2004). O Ecossistema Estuarino: Ecologia, Ameaças e Gestão. Nova York, NY: Oxford University Press. doi: 10.1093/acprof:oso/9780198525080.001.0001
CrossRef Full Text | Google Scholar
Megonigal, J. P., Mines, M. E., e Visscher, P. T. (2005). “Metabolismo anaeróbico: ligações a gases vestigiais e processos aeróbicos”, em Biogeoquímica, ed., 2005. W. H. Schlesinger (Oxford: Elsevier), 350-362.
Google Scholar
Milferstedt, K., Youngblut, N. D., e Whitaker, R. J. (2010). Estrutura espacial e persistência de populações de metanogênio em pântanos húmicos. ISME J. 4, 764-776. doi: 10.1038/Ismej.2010.7
PubMed Abstract | CrossRef Full Text | Google Scholar
Mosier, A. C., e Francis, C. A. (2008). A abundância e diversidade relativas de bactérias e arcáceas amonia-oxidantes no estuário da Baía de São Francisco. Ambiente. Microbiol. 10, 3002-3016. doi: 10.1111/j.1462-2920.2008.01764.x
PubMed Abstract | CrossRef Full Text | Google Scholar
Nazaries, L., Murrell, J. C., Millard, P., Baggs, L., and Singh, B. K. (2013). Metano, micróbios e modelos: compreensão fundamental do ciclo do metano do solo para previsões futuras. Ambiente. Microbiol. 15, 2395-2417. doi: 10.1111/1462-2920.12149
PubMed Abstract | CrossRef Full Text | Google Scholar
Nelson, M. B., Martiny, A. C., and Martiny, J. B. H. (2016). Biogeografia global dos traços de ciclagem microbiana de nitrogênio no solo. Proc. Natl. Acad. Sci. U.S.A. 113, 8033-8040. doi: 10.1073/pnas.1601070113
PubMed Abstract | CrossRef Full Text | Google Scholar
Nemergut, D. R., Costello, E. K., Hamady, M., Lozupone, C., Jiang, L., Schmidt, S. K., et al. (2011). Padrões globais na biogeografia de taxa bacteriana. Ambiente. Microbiol. 13, 135-144. doi: 10.1111/j.1462-2920.2010.02315.x
PubMed Abstract | CrossRef Full Text | Google Scholar
Nobu, M. K., Narihiro, T., Kuroda, K., Mei, R., and Liu, W. T. (2016). Chasing the elusive Euryarchaeota class WSA2: genomes reveal a uniquely fastidious methyl-reducing methanogen. ISME J. 10, 2478-2487. doi: 10.1038/ismej.2016.33
PubMed Abstract | CrossRef Full Text | Google Scholar
Oksanen, J., Blanchet, F. G., Kindt, R., Legendre, P., Minchin, P. R., O’Hara, R. B., et al. (2015). vegan: Pacote de Ecologia Comunitária. R Pacote Versão 2.2.0. Disponível em: https://CRAN.R-project.org/package=vegan
Google Scholar
Oloo, F., Valverde, A., Quiroga, M. V., Vikram, S., Cowan, D., e Mataloni, G. (2016). A heterogeneidade do habitat e a conectividade formam comunidades microbianas em turfeiras sul-americanas. Sci. Rep. 6:25712. doi: 10.1038/srep25712
PubMed Abstract | CrossRef Full Text | Google Scholar
Pattnaik, P., Mishra, S. R., Bharati, K., Mohanty, S. R., Sethunathan, N., e Adhya, T. K. (2000). Influência da salinidade na metanogênese e microflora associada em solos tropicais de arroz. Microbiol. Res. 155, 215-220. doi: 10.1016/S0944-5013(00)80035-X
PubMed Abstract | CrossRef Full Text | Google Scholar
Poffenbarger, H. J., Needelman, B. A., e Megonigal, J. P. (2011). Influência da salinidade nas emissões de metano dos pântanos das marés. Wetlands 31, 831-842. doi: 10.1007/s13157-011-0197-0
PubMed Abstract | CrossRef Full Text | Google Scholar
Potter, E. G., Bebout, B. M., e Kelley, C. A. (2009). Composição isotópica do metano e dos substratos metanogénicos inferidos ao longo de um gradiente de salinidade num sistema hipersalino de tapetes microbianos. Astrobiologia 9, 383-390. doi: 10.1089/ast.2008.0260
PubMed Abstract | CrossRef Full Text | Google Scholar
R Core Team (2014). R: A Language and Environment for Statistical Computing (Versão 3.0.3). Viena: R Fundação para a Computação Estatística.
Google Scholar
Roberts, D. (2016). labdsv: Ordenação e Análise Multivariada para a Ecologia. Pacote R Versão 1.8-0. Disponível em: https://CRAN.R-project.org/package=labdsv
Google Scholar
Rooney-Varga, J. N., Giewat, M. W., Duddleston, K. N., Chanton, J. P., and Hines, M. E. (2007). Links entre a estrutura da comunidade arqueal, tipo de vegetação e caminho metanogênico em turfeiras do Alasca. FEMS Microbiol. Ecol. 60, 240-251. doi: 10.1111/j.1574-6941.2007.00278.x
PubMed Abstract | CrossRef Full Text | Google Scholar
Rosenberg, E., DeLong, E. F., Lory, S., Stackebrandt, E., e Thompson, F. (2014). Os Prokaryotes: Outras Linhagens Principais de Bactérias e da Archaea. Berlim: Springer.
Google Scholar
Ruff, S. E., Biddle, J. F., Teske, A. P., Knittel, K., Boetius, A., e Ramette, A. (2015). Dispersão global e diversificação local do microbioma do infiltrado de metano. Proc. Natl. Acad. Sci. U.S.A. 112, 4015-4020. doi: 10.1073/pnas.1421865112
PubMed Abstract | CrossRef Full Text | Google Scholar
Schauer, R., Bienhold, C., Ramette, A., and Harder, J. (2009). Diversidade bacteriana e biogeografia em sedimentos de superfície de mar profundo do Oceano Atlântico Sul. ISME J. 4, 159-170. doi: 10.1038/ismej.2009.106
PubMed Abstract | CrossRef Full Text | Google Scholar
Schloss, P. D., Westcott, S. L., Ryabin, T., Hall, J. R., Hartmann, M., Hollister, E. B., et al. (2009). Introduzindo mothur: software de código aberto, independente de plataforma, apoiado pela comunidade para descrever e comparar comunidades microbianas. Appl. Environ. Microbiol. 75, 7537-7541. doi: 10.1128/Aem.01541-09
PubMed Abstract | CrossRef Full Text | Google Scholar
Statham, P. J. (2012). Nutrientes em estuários – uma visão geral e os impactos potenciais das mudanças climáticas. Sci. Total Environ. 434, 213-227. doi: 10.1016/j.scitotenv.2011.09.088
PubMed Abstract | CrossRef Full Text | Google Scholar
Thauer, R. K., Kaster, A.-K., Seedorf, H., Buckel, W., e Hedderich, R. (2008). Metanogenic archaea: differences ecologically relevant in energy conservation. Nat. Rev. Microbiol. 6, 579-591. doi: 10.1038/Nrmicro1931
PubMed Abstract | CrossRef Full Text | Google Scholar
Tilman, D., Isbell, F., and Cowles, J. M. (2014). Biodiversidade e funcionamento dos ecossistemas. Annu. Rev. Ecol. Evol. Syst. 45, 471-493. doi: 10.1146/annurev-ecolsys-120213-091917
CrossRef Full Text | Google Scholar
Ulrich, W., Hajdamowicz, I., Zalewski, M., Stañska, M., Ciurzycki, W., e Tykarski, P. (2010). Sortimento de espécies ou filtragem de habitat: um estudo de caso das comunidades de aranhas nas ilhas dos lagos. Ecol. Res. 25, 375-381. doi: 10.1007/s11284-009-0661-y
CrossRef Full Text | Google Scholar
Vanwonterghem, I., Evans, P. N., Parks, D. H., Jensen, P. D., Woodcroft, B. J., Hugenholtz, P., et al. (2016). Metanogênese metilotrófica descoberta no filo arquebactéria Verstraetearchaeota. Nat. Microbiol. 1:16170. doi: 10.1038/nmicrobiol.2016.170
PubMed Abstract | CrossRef Full Text | Google Scholar
von Mering, C., Hugenholtz, P., Raes, J., Tringe, S. G., Doerks, T., Jensen, L. J., et al. (2007). Avaliação filogenética quantitativa de comunidades microbianas em diversos ambientes. Science 315, 1126-1130. doi: 10.1126/science.1133420
PubMed Abstract | CrossRef Full Text | Google Scholar
Wagner, D., and Liebner, S. (2009). “Aquecimento global e dinâmica do carbono em solos permafrost: produção de metano e oxidação”, em Permafrost Soils, ed. R. Margesin (Berlin: Springer), 219-236.
Google Scholar
Weiher, E., e Keddy, P. (2001). Regras da Assembléia Ecológica: Perspectivas, Avanços, Retiros. Cambridge: Cambridge University Press.
Google Scholar
Whitaker, R. J., Grogan, D. W., e Taylor, J. W. (2003). Barreiras geográficas isolam populações endêmicas de arquebactérias hipertermofílicas. Science 301, 976-978. doi: 10.1126/science.1086909
PubMed Abstract | CrossRef Full Text | Google Scholar
Wickham, H. (2009). ggplot2: Gráficos Elegantes para Análise de Dados. Nova York, NY: Springer-Verlag. doi: 10.1007/978-0-387-98141-3
CrossRef Full Text | Google Scholar
Yang, J., Ma, L. A., Jiang, H., Wu, G., and Dong, H. (2016). A salinidade forma a diversidade microbiana e a estrutura comunitária nos sedimentos superficiais dos Lagos Qinghai-Tibetanos. Sci. Rep. 6:25078. doi: 10.1038/srep25078
PubMed Abstract | CrossRef Full Text | Google Scholar
Yang, S., Liebner, S., Alawi, M., Ebenhöh, O., e Wagner, D. (2014). Taxonomic database and cut-off value for processing mcrA gene 454 pyrosequencing data by MOTHUR. J. Microbiol. Métodos 103, 3-5. doi: 10.1016/j.mimet.2014.05.006
PubMed Abstract | CrossRef Full Text | Google Scholar
Yang, S., Liebner, S., Winkel, M., Alawi, M., Horn, F., Dörfer, C., et al. (2017). Análise aprofundada de comunidades metanogênicas centrais de zonas úmidas de alta elevação permafrost-afetadas. Biol do Solo. Bioquímica. 111, 66-77. doi: 10.1016/j.soilbio.2017.03.007
CrossRef Full Text | Google Scholar
Ye, R. Z., Jin, Q. S., Bohannan, B., Keller, J. K., McAllister, S. A., e Bridgham, S. D. (2012). O pH controla a mineralização anaeróbica do carbono, a eficiência da produção de metano, e as vias metanogénicas em turfeiras através de um gradiente ombrotrófico-minerotrofio. Biol do Solo. Bioquímica. 54, 36-47. doi: 10.1016/j.soilbio.2012.05.015
CrossRef Full Text | Google Scholar
Zinder, S. H. (1993). “Fisiological ecology of methanogens”, em Methanogenesis: Ecologia, Fisiologia, Bioquímica & Genetics, ed. J. G. Ferry (Boston, MA: Springer), 128-206.
Google Scholar
Zinger, L., Amaral-Zettler, L. A., Fuhrman, J. A., Horner-Devine, M. C., Huse, S. M., Welch, D. B. M., et al. (2011). Padrões globais de beta-diversidade bacteriana em ecossistemas de fundos marinhos e de água do mar. PLoS ONE 6:e24570. doi: 10.1371/journal.pone.0024570
PubMed Abstract | CrossRef Full Text | Google Scholar