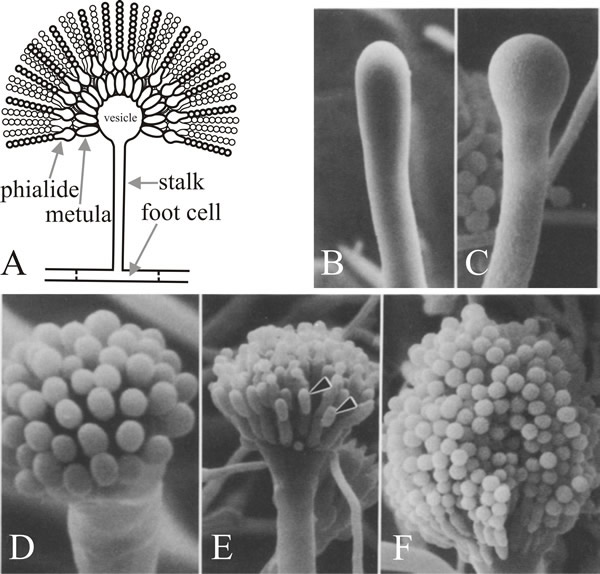
Die Zelle, aus der der Zweig hervorgeht, ist die Konidiophorenfußzelle, die sich von anderen vegetativen Zellen dadurch unterscheidet, dass sie eine braun pigmentierte sekundäre Wandverdickung auf der Innenseite ihrer ursprünglichen Wand hat. Der Stiel wächst apikal, bis er eine Länge von ca. 100 µm erreicht, wenn der Apex anschwillt und die Konidiophore mit einem Durchmesser von ca. 10 µm bildet. Aus dem Vesikel sprießen dann eine einzelne Reihe zahlreicher primärer Sterigmata, Metulae genannt, und sekundäre Sterigmata, die Phialiden, aus den exponierten Spitzen der Metulae. Die Phialiden sind die Stammzellen, die sich anschließend wiederholt asymmetrisch teilen, um die langen Ketten enteroblastischer Konidien zu bilden, die einen Durchmesser von etwa 3 µm haben (Abb. 7).
Die klassische genetische Analyse durch Isolierung und Analyse von Mutanten hat den grundlegenden genetischen Aufbau der Konidiation von Aspergillus festgelegt. Man schätzte, dass zwischen 300 und 1.000 Genorte an der Konidiation beteiligt sind, indem man die Mutationshäufigkeiten an Orten, die die Konidiation beeinflussen, mit denen für andere Funktionen verglich (Martinelli & Clutterbuck, 1971). Die Analyse der mRNA-Arten ergab, dass etwa 6.000 im vegetativen Myzel exprimiert wurden und weitere 1.200 in Kulturen, die Konidiophoren und Konidien enthielten; 200 dieser zusätzlichen mRNAs wurden in den Konidien selbst gefunden. Nur etwa 2 % der Mutanten von A. nidulans, denen Konidien fehlten, wiesen Defekte in den Stadien auf, die mit dem Wachstum und der Entwicklung der Konidiophore zusammenhängen, und 85 % der Konidienmutanten waren auch beim vegetativen Hyphenwachstum und bei der Erlangung der Kompetenz defekt.
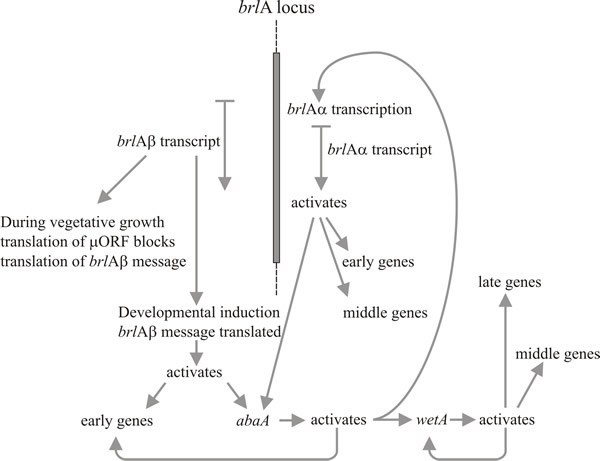
Vor allem zwei Gene spielen eine Schlüsselrolle bei der Morphogenese der Konidiophore: das „Borsten“-Gen (brlA), das Defekte bei der Vesikel- und Metula-Bildung aufweist, und das „Abakus“-Gen (abaA), bei dem die Konidien durch wulstige Hyphenabschnitte ersetzt werden, so dass es vermutlich zu Defekten bei der Konidienknospung aus der Phialide und der endgültigen Septierung kommt. Ein drittes Gen, wetA, ist in einem frühen Stadium der Sporenreifung defekt. Den Konidien der wetA-Mutanten fehlt es an Pigment und Hydrophobie; sie autolytisieren nach einigen Stunden und exprimieren eine Reihe sporenspezifischer mRNAs nicht. Das Transkript des wetA-Gens fehlt in brlA- und abaA-Mutanten (d. h. brlA und abaA sind epistatisch zu wetA), und Untersuchungen von Doppelmutanten zeigen, dass diese drei Gene in der Reihenfolge brlA → abaA → wetA wirken.
Ein auffälliges Merkmal der Mutationsanalyse der Konidiophorenentwicklung in A. nidulans ist, dass Mutanten nur dieser drei Gene Defekte in der Konidiophoren- und Sporenmorphogenese verursachen, während bis zu 1.000 andere Genloci bei Mutation eine fehlende Konidiation verursachen. Dies deutet darauf hin, dass brlA, abaA und wetA Regulatoren sind, die die Expression anderer Gene integrieren, die für die Konidiation benötigt werden, aber nicht selbst dafür verantwortlich sind. Viele der Aspergillus-Konidiationsmutanten waren auch bei der sexuellen Fortpflanzung defekt, so dass eine weitere Schlussfolgerung darin besteht, dass morphogenetische Gene bei verschiedenen Entwicklungsprozessen in gewissem Umfang ökonomisch genutzt werden. Vermutlich werden bei den verschiedenen Entwicklungsmodi Strukturgene eingesetzt, die nicht ausschließlich für die Entwicklung zuständig sind, sondern in zahlreichen Pfaden funktionieren, wobei ihnen ihre Entwicklungsspezifität durch die Regulatoren verliehen wird, auf die sie reagieren. Dies wird in der Vorstellung verkörpert, dass der Schlüssel zur Entwicklung von Eukaryonten in der Fähigkeit liegt, relativ wenige regulatorische Gene zu nutzen, um die Aktivitäten vieler anderer zu integrieren.
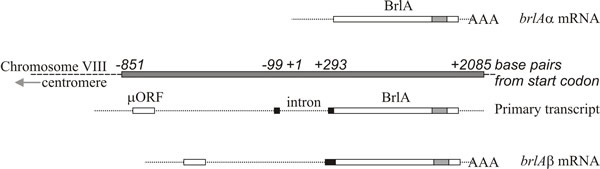
Die molekulare Analyse unterstützt die Interpretation, dass brlA, abaA und wetA Regulatoren sind; die brlA-Sequenz kodiert für ein Zinkfingerprotein, das ein sequenzspezifischer DNA-bindender Transkriptionsaktivator von entwicklungsregulierten Zielgenen ist (Abb. 8). Das ist jedoch noch nicht alles, denn das brlA-Produkt hat unterschiedliche Affinitäten für verschiedene Zielgene. Tatsächlich besteht der brlA-Locus aus sich überlappenden Transkriptionseinheiten (Abb. 9), wobei die stromabwärts gelegene Einheit als brlAα und die stromaufwärts gelegene Einheit als brlAβ bezeichnet wird; zwischen ihnen lösen ihre Produkte zwei klassische Entwicklungsprobleme, nämlich wie man auf ein Signal reagiert und wie man diese Reaktion aufrechterhält, wenn das Signal verpufft ist.
Die beiden brlA-Transkriptionseinheiten haben über den größten Teil ihrer Länge das gleiche Leseraster, aber brlAβ hat zusätzliche 23 Aminosäurereste am aminoterminalen Ende dieses Leserasters, und sein Transkript besitzt auch ein separates Leseraster von 41 Aminosäureresten (µORF genannt) in der Nähe seines 5′-Terminus. Das µORF-Produkt unterdrückt die Translation aus dem stromabwärts gelegenen Leseraster (BrlA), und obwohl das BrlAβ-Transkript in vegetativen Hyphen nachgewiesen werden kann, wird das BrlA-Peptid nicht produziert. Die durch µORF verursachte Unterdrückung wird nur überwunden, wenn das Myzel kompetent ist, was wahrscheinlich als Stickstofflimitierung signalisiert wird (ein übliches Umweltsignal für die Einleitung der Sporulation bei Ascomycota), wodurch die Aminoacyl-tRNA-Pools reduziert und die Translationsregulation durch µORF gestört wird. Wenn die Wirkung von µORF abgewürgt wird, kann das BrlA-Peptid aus dem vorhandenen Transkript übersetzt werden.
Der Entdecker dieses Prozesses beschrieb die Aktivierung des Konidiationsweges als „translationales Triggern“ (Timberlake, 1993), was darauf hindeutet, dass der translationale Trigger eine Möglichkeit ist, die Differenzierung auf den Ernährungszustand der Hyphe empfindlich zu machen. Die kompetente Hyphe ist also für die Entwicklung der Konidiophore vorbereitet, aber die irreversible Aktivierung des Konidiationsweges wird durch eine Translationsunterdrückung verhindert, die das vegetative Wachstum aufrechterhält, bis die Sporulationsbedingungen ideal sind.
Die Aktivierung von brlA wird daher als der erste Schritt in der Konidiophorenentwicklung angesehen, und sein Produkt aktiviert wiederum eine Reihe von konidiationsspezifischen Genen, zu denen auch der nächste Regulator, abaA, gehört. Das abaA-Produkt ist ebenfalls ein DNA-bindendes Transkriptionsregulatorprotein, das die Expression von brlA-induzierten Strukturgenen verstärkt. Die brlA- und abaA-Gene sind wechselseitige Aktivatoren, da abaA auch brlA aktiviert. Natürlich muss brlA exprimiert werden, bevor abaA exprimiert werden kann, aber die daraus resultierende abaA-Aktivierung von brlA verstärkt dessen Expression und macht den Fortschritt des Weges effektiv unabhängig von äußeren Ereignissen. Das abaA-Produkt aktiviert auch zusätzliche Strukturgene und das letzte Regulierungsgen, wetA, das sporenspezifische Strukturgene aktiviert. Da brlA und abaA in sich differenzierenden Konidien nicht exprimiert werden, ist wetA wahrscheinlich an der Inaktivierung ihrer Expression in den Sporen beteiligt (und vielleicht auch im Phialid; wenn der aktuelle Sporenkern das Reifestadium erreicht, für das wetA-regulierte Gene benötigt werden, müssen die Phialidenkerne in den Konidien-Initiationszustand „zurückgedreht“ werden, um die Bildung der nächsten Spore zu beginnen). Die Expression von wetA wird im Phialid zunächst durch die sequentielle Wirkung von brlA und abaA aktiviert und dann autoreguliert (das wetA-Produkt aktiviert die wetA-Transkription). Eine positive Autoregulation von wetA hält die Expression auch nach der (physischen und zytologischen) Trennung des Konidiums vom Phialid aufrecht.
Timberlake (1993) bezeichnete diesen Mechanismus als Feedback-Fixierung: die wechselseitige Aktivierung, die Feedback-Aktivierung und die Autoregulation der zentralen regulatorischen Sequenzen verstärken die Expression des gesamten Weges, machen ihn unabhängig von den externen Umweltfaktoren, die ihn ausgelöst haben, und ermöglichen es der Spore, ihre Reifung auch nach der Trennung vom Phialid fortzusetzen. Die Entwicklung der Konidiophore ist wie viele andere morphogenetische Prozesse natürlich in sequenzielle Schritte unterteilt. Dieses regulatorische Netzwerk zeigt, wie translatorische Triggerung einen morphogenetischen Pfad einerseits mit der Entwicklung von Kompetenz und andererseits mit der Initiierung als Reaktion auf Umweltreize verbinden kann. Im Anschluss an die Initiierung führt die Rückkopplungsfixierung zu einer Entwicklungsbestimmung im klassischen embryologischen Sinne, bei der die Morphogenese auch dann fortgesetzt wird, wenn sie aus der initiierenden Umgebung entfernt wird.
Wir haben uns in diesem Abschnitt bewusst dafür entschieden, unsere Beschreibung auf Forschungen zu stützen, die vor etwa 50 Jahren durchgeführt wurden und die mit der Isolierung einer großen Anzahl von Mutationen mit veränderten Konidiationsphänotypen begannen und mit „klassischen“ Studien zu Genseigerungen und Genkomplementation fortgesetzt wurden (Martinelli & Clutterbuck, 1971). Dies zeigt nicht nur, wie sich unser Verständnis der Sporulation entwickelt hat, sondern auch, wie wertvoll es ist, diese „altmodischen“ Techniken zur Untersuchung solcher Phänomene einzusetzen.
Allerdings können wir die umfangreichen Forschungsarbeiten nicht ignorieren, die zu den molekularen Mechanismen durchgeführt wurden, die dem Wachstum und der Entwicklung von Aspergillus zugrunde liegen. Diese Studien haben bestätigt, dass das Schlüsselereignis bei der Sporulation die Aktivierung des Zinkfinger-Transkriptionsfaktors ist, der von brlA kodiert wird, und dass die abaA- und wetA-Gene notwendige Regulatoren der Konidiation sind. Der von abaA kodierte Transkriptionsfaktor wird von brlA nach der Differenzierung der Metulae und während der mittleren Stadien der Konidiophorenentwicklung aktiviert; das von abaA aktiviertewetA-Gen wirkt in der späten Phase der Konidiation, indem es die Synthese wichtiger Zellwandkomponenten steuert und den Stoffwechsel des reifenden Konidiums verändert. In Aspergillus nidulans bilden diese drei Gene den zentralen Regulationsweg, der zusammen mit anderen Genen die konidiationsspezifische Genexpression und die Sequenz der Genaktivierung steuert, die am Erwerb der Entwicklungskompetenz, der Konidiophorenentwicklung und der Sporenreifung beteiligt ist (Lee et al., 2016).
Viele Aspekte dieser Entwicklungswege sind in anderen Aspergillen konserviert worden. Mitglieder der Gattung Aspergillus gehören zu den am häufigsten vorkommenden Pilzen und vermehren sich alle ungeschlechtlich, indem sie lange Ketten von Konidien bilden. Mehrere Arten, darunter Aspergillus oryzae und Aspergillus niger, werden in der Industrie für die Enzymproduktion und die Lebensmittelverarbeitung verwendet, während Aspergillus flavus für den Verderb von Lebensmitteln verantwortlich ist, indem er die stärksten bekannten natürlich vorkommenden Karzinogene, die Aflatoxine, produziert. Eine weitere besorgniserregende Spezies ist der opportunistische Humanpathogen Aspergillus fumigatus, der eine große Anzahl kleiner hydrophober Konidien als primäres Verbreitungsmedium produziert und in den Industrieländern zu einem weit verbreiteten Pilzerreger geworden ist, der über die Luft übertragen wird. Bei immungeschwächten Patienten verursacht A. fumigatus eine invasive Aspergillose, die eine hohe Sterblichkeitsrate aufweist. Der BrlA-AbaA-WetA-Entwicklungssignalweg ist bei der Konidiation dieser Aspergillus-Arten konserviert worden (Yu, 2010; Tao & Yu, 2011; Krijgsheld et al., 2013). Darüber hinaus haben proteomische, transkriptomische und metabolomische Studien ein detailliertes Bild der dynamischen Veränderungen geliefert, die in vielen Tausenden von Genen, Transkripten, Enzymen und Stoffwechselreaktionen während der Bildung, Reifung, Dormanz und Keimung von Aspergillus-Konidien auftreten (van Leeuwen et al., 2013; Novodvorska et al., 2016; Teertstra et al., 2017).
Aktualisiert Juli, 2019