Introduction
Methan (CH4) ist ein wichtiges Treibhausgas. Seine Emissionen aus natürlichen Umgebungen wie Feuchtgebieten, Ozeanen und Sedimenten machen weltweit über 70 % des atmosphärischen Methans aus (IPCC, 2007). Eine Auswertung der veröffentlichten Daten ergab unterschiedliche Methanemissionsraten für Feuchtgebiete, Seen, Flüsse, Flussmündungen und Ozeane (in abnehmender Reihenfolge, siehe ergänzende Abbildung S1). Natürliche Feuchtgebiete allein sind für 62 % der biogenen CH4-Produktion verantwortlich (Kirschke et al., 2013; Nazaries et al., 2013), und die Emissionen aus Feuchtgebieten dominieren die interannuelle Variabilität der Methanquellen (Bousquet et al., 2006). Im Gegensatz dazu tragen die riesigen Flächen der Meeresökosysteme nur etwa 8 % zu den natürlichen CH4-Quellen bei (Nazaries et al., 2013). Von den globalen ozeanischen Methanemissionen entfallen weniger als 10 % auf Ästuare (Bange et al., 1994).
Methanogenese, die biologische Bildung von Methan, wird von methanogenen Archaeen durchgeführt, die Methan unter anoxischen Bedingungen hauptsächlich aus H2/CO2, Methylgruppen oder Acetat produzieren (Thauer et al., 2008). Diese Reaktion wird durch die Methyl-Coenzym-M-Reduktase (MCR) katalysiert. Das mcrA-Gen, das für eine Untereinheit dieses Enzyms kodiert, ist ein häufig verwendeter Genmarker bei molekularen Untersuchungen (Conrad, 2007; Bridgham et al., 2013). Der Vorteil des mcrA-Genmarkers besteht darin, dass er sowohl die phylogenetischen als auch die funktionellen Signaturen von Methanogenen erfasst und eine hohe Sequenzierungstiefe für diese spezielle Funktion bietet (Luton et al., 2002; Borrel et al., 2013; Yang et al., 2014). Es wurde eine große Anzahl von mcrA-Sequenzen aus einer Vielzahl von natürlichen Umgebungen abgerufen. Der öffentliche mcrA-Datensatz ermöglicht es, allgemeine ökologische Muster zu extrahieren und die Gestaltung von Umweltgradienten auf globaler und regionaler Ebene zu untersuchen. Darüber hinaus ist eine Datenbank verfügbar, in der die physiologischen Eigenschaften von 152 methanogenen Isolaten zusammengefasst sind1 (Jabłoński et al., 2015). Kürzlich wurden durch Genom-Binning ungewöhnliche mcrA-Sequenzen in der neuen Klasse der Methanofastidiosa (Nobu et al., 2016) und in den neuen Phyla der Bathyarchaeota (Evans et al., 2015) und Verstraetearchaeota (Vanwonterghem et al., 2016) entdeckt. Diese neuen Erkenntnisse haben unser Wissen über die Vielfalt potenzieller Methanogene erweitert, aber die Anwendbarkeit des mcrA-Gens als molekularer Marker für die große Mehrheit der methanogenen Gemeinschaften nicht in Frage gestellt.
Bis heute wurden methanogene Gemeinschaften in Feuchtgebieten, Sedimenten, Permafrostgebieten, Reisfeldern, Fermentern, geothermischen Quellen und hydrothermalen Schloten nachgewiesen (Conrad, 2007; Thauer et al., 2008; Wagner und Liebner, 2009). Es wurde festgestellt, dass die Struktur der methanogenen Gemeinschaft mit dem pH-Wert der Umwelt, der Temperatur, dem Salzgehalt, dem Grundwasserspiegel und der Vegetationsdynamik auf verschiedenen räumlichen und zeitlichen Ebenen zusammenhängt (Megonigal et al., 2005; Milferstedt et al., 2010; Frank-Fahle et al., 2014; McCalley et al., 2014; Cui et al., 2015; Liebner et al., 2015). So wird beispielsweise die acetoklastische Methanogenese im Allgemeinen durch einen niedrigen pH-Wert behindert, da dieser die Acetatdissoziation verringert (Megonigal et al., 2005; Kotsyurbenko et al., 2007). Die Vegetation kann labilen, hochwertigen organischen Kohlenstoff als Brennstoff für Methanogene in Form von Wurzelexsudaten oder Detritus liefern, so dass Pflanzenexsudate im Allgemeinen acetoklastische Methanogene vor allem in Niedermooren begünstigen (Bridgham et al., 2013). Sulfat aus dem Meerwasser hemmt die Methanproduktion in Gezeitenfeuchtgebieten, und der Salzgehalt wurde daher als allgemeiner Prädiktor für Methanemissionen verwendet (Holm et al., 2016). Eine Studie an Sedimenten tibetischer Seen zeigte, dass ein steigender Salzgehalt hydrogenotrophe Methanogene hemmt, aber die acetoklastische Methanogenese fördert (Liu et al., 2016). Diese Studien weisen auf umweltbedingte Faktoren für methanogene Gemeinschaften hin, konzentrierten sich jedoch auf einzelne Lebensräume oder begrenzte räumliche Skalen.
Um die Anpassung von Methanogenen an verschiedene Umweltveränderungen zu verstehen, ist jedoch eine systematische und globale Erforschung der Zusammenhänge zwischen der Zusammensetzung mikrobieller Gemeinschaften und den Umweltbedingungen erforderlich (Lozupone und Knight, 2007). Derzeit gibt es nur wenige Studien, die sich mit der Ausbreitung und Habitatfilterung von Methanogengemeinschaften befassen (Auguet et al., 2010; Barreto et al., 2014). Wir stellen die Hypothese auf, dass methanogene Gemeinschaften hauptsächlich durch die Filterung des Lebensraums beeinflusst werden und dass dies durch globale Umweltkontrollen gesteuert wird. In Anbetracht der Tatsache, dass sich die Methanemissionsraten in natürlichen Ökosystemen stark unterscheiden, kann die explizite Integration der Zusammensetzung, Vielfalt und Biogeografie methanogener Assemblagen in diesen Ökosystemen von grundlegender Bedeutung sein, um die Reaktion der Methanproduktion auf aktuelle und künftige Klimaänderungen zu bestimmen. Diese Metastudie wird durchgeführt, um die Lücken zu schließen, die mit der Biogeografie, der Diversität und den Umweltkontrollen von Methanogenen verbunden sind, indem öffentlich verfügbare mcrA-Sequenzdaten und Literatur verwendet werden, die durch physiologische Daten von Methanogen-Isolaten ergänzt werden.
Materialien und Methoden
Datensammlung
Wir haben mcrA-Sequenzen abgerufen, die in GenBank (Januar 2016)2 verfügbar sind. Für jeden Treffer wurde die Originalarbeit überprüft und die entsprechenden mcrA-Sequenzen wurden von einem benutzerdefinierten Perl-Skript geparst. Da wir uns auf natürliche Umgebungen konzentrierten, wurden methanogene mcrA-Sequenzen aus natürlichen Lebensräumen gewonnen und als Boden, See-, Ästuar-, Meeres- und hydrothermale Sedimente sowie Schlammvulkane klassifiziert. Zusätzlich zu den Sequenzen der Klonbibliotheken wurden fünf Bibliotheken aus dem Next Generation Sequencing (NGS) einbezogen. Die Sequenzen wurden heruntergeladen, ohne die relative Häufigkeit im ursprünglichen Datensatz zu berücksichtigen. Da die Sequenzen von Klonbibliotheken hauptsächlich die reichlich vorhandenen Phylotypen abdecken, während NGS eine viel größere Vielfalt erfassen kann, sind wir einen Kompromiss eingegangen, um die NGS-Daten zu verwenden, haben aber gleichzeitig einen möglichen Fehler aufgrund der unterschiedlichen Auflösung der Sequenzierungsmethoden abgeschwächt. Daher wählten wir nur die repräsentativen Sequenzen von häufigen OTUs mit einer relativen Häufigkeit von mehr als 1 % aus. Außerdem haben wir diejenigen NGS-Sequenzen verworfen, die bei der Übersetzungsprüfung von Nukleotid- zu Proteinsequenzen durchgefallen sind oder eine niedrige Qualität aufwiesen (Sequenzen < 250 bps). Schließlich erstellten wir einen Datensatz mit 4466 einzigartigen mcrA-Sequenzen von 94 weltweit verteilten Standorten (Abbildung 1 und ergänzende Tabellen S1, S2). Darüber hinaus haben wir die mcrA-Sequenzen potenzieller archaeischer Methanotropher nicht vom Datensatz abgezogen, die bei der genomischen Untersuchung zwangsläufig entdeckt wurden (Conrad, 2007). Dieser Teil liegt nicht im Fokus dieser Studie.
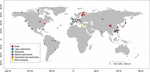
Abbildung 1. Lage der für diese Studie verwendeten Standorte. Die Standorte wurden nach ihren Lebensräumen in sechs verschiedene Kategorien eingeteilt und mit unterschiedlichen Farben gekennzeichnet. Eine Zusammenfassung der sechs Lebensräume ist in den ergänzenden Tabellen S1 und S2 zu finden.
Die geografischen Koordinaten und Umweltparameter wie pH-Wert, Salzgehalt, Höhe, mittlere jährliche Lufttemperatur (MAAT) und mittlerer jährlicher Niederschlag (MAP) wurden für jeden in dieser Studie untersuchten Standort aus den entsprechenden Veröffentlichungen entnommen, sofern die Daten verfügbar sind (siehe ergänzende Tabelle S1). Um fehlende Umweltparameter in mehreren Studien zu berücksichtigen, definierten wir einige Umweltvariablen qualitativ gemäß den Standortbeschreibungen in der einschlägigen Literatur und wandelten diese Kategoriedaten dann in semi-metrische numerische Werte um. So definierten wir beispielsweise marine Sedimente, hydrothermale Sedimente, vulkanischen Schlamm und Sedimentproben aus Sodaseen als „salzhaltig“, Boden- und Süßwasserseesedimentproben als „nicht salzhaltig“ und Mangroven- und Ästuarproben als „gemischte“ Proben.
Raw Sequence Processing
Die Sequenzverarbeitung wurde mit der Softwareplattform Mothur (Schloss et al., 2009). Sequenzen aus verschiedenen Bibliotheken wurden vor der Verarbeitung gepoolt. Sequenzen mit einer Länge von weniger als 350 bp oder mehr als 8 mehrdeutigen Basen wurden verworfen. Anschließend wurden diese Sequenzen mit einer bereits ausgerichteten Teilmenge von mcrA-Sequenzen aligniert, die aus der FunGene-Datenbank unter http://fungene.cme.msu.edu/ (Fish et al., 2013) abgerufen wurden. Chimäre Sequenzen wurden mit der Mothur-Software unter Verwendung der uchime-Methode (Edgar et al., 2011) identifiziert, wobei der Datensatz selbst als Referenz diente. Anschließend wurden die gültigen Nukleotidsequenzen des mcrA-Gens verwendet, um die unkorrigierten paarweisen Abstände zwischen den ausgerichteten DNA-Sequenzen zu berechnen, und anschließend mit einem Cutoff von 84 %, der 97 % für das 16S rRNA-Gen entspricht, in operative taxonomische Einheiten (OTU) eingeteilt (Yang et al., 2014). Die Häufigkeit jeder mcrA-OTU wurde nur als Vorhandensein oder Fehlen berücksichtigt. Wir haben die Genauigkeit der taxonomischen Klassifizierung der OTUs erhöht, indem wir sowohl Nukleotid- als auch Aminosäuresequenzen berücksichtigt haben. Auf DNA-Ebene wurde die taxonomische Identität von der Mothur-Plattform gemäß einer Referenzdatenbank (Yang et al., 2014) zugewiesen. Auf Proteinebene wurden die ausgerichteten Proteinsequenzen verwendet, um einen Baum in ARB zu konstruieren, und dann wurde die taxonomische Zuordnung auf der Grundlage der entsprechenden Datenbank vorgenommen. Wenn die Zuordnung einer OTU nicht konsistent war, haben wir sowohl die Nukleotid- als auch die Proteinsequenz im NCBI manuell geblastet und die endgültige taxonomische Identität unter Berücksichtigung der Abfrageabdeckung (>95 %), der Identität (>84 %) und des e-Werts (<1E-5) bestimmt. Für Proteinsequenzen bezog sich der Cutoff auf der Gattungsebene auf den Schwellenwert von 83,5% (Hunger et al., 2011).
Ökologische und statistische Analyse
Die statistische Analyse wurde mit verschiedenen R-Paketen durchgeführt. Die Ordinationen der Hauptkoordinatenanalyse (PCoA) wurden auf der Grundlage von Jaccard-Distanzmatrizen erstellt, die mit dem vegan-Paket v2.2.0 (Oksanen et al., 2015) konstruiert wurden. Eine permutative MANOVA (multivariate Varianzanalyse) wurde durchgeführt, um die Quelle der Variation in der Jaccard-Matrix (McArdle und Anderson, 2001) in vegan mit 104 Permutationen zu bewerten. Die Jaccard-Distanzmaße basieren auf dem Vorhandensein/Abwesenheit der Arten, was für unseren Datensatz besser geeignet ist, da die meisten Studien nur die repräsentativen Sequenzen lieferten, während die Informationen über die Häufigkeit fehlen. Die Häufigkeit der taxonomischen Vorkommen in den verschiedenen Lebensräumen wurde mit dem ggplot2-Paket (v1.0.0) (Wickham, 2009) in Form von Blasenplots dargestellt. Die hierarchische Clustering-Analyse der nicht-salzhaltigen Boden- und Seesediment-Gemeinschaften wurde mit der R-Funktion „hclust“ (R Core Team, 2014) durchgeführt. Die erhaltenen Gemeinschaftscluster wurden entsprechend dem pH- und Temperaturregime der ursprünglichen Proben beschrieben, da wir mit Hilfe von PCoA zuvor festgestellt hatten, dass die beiden Parameter pH und Temperatur die Zusammensetzung der methanogenen Gemeinschaft in nicht-salinen Böden und Seesedimenten beeinflussen. Die Assoziation jeder methanogenen Linie mit jedem dieser Cluster wurde mit Hilfe einer korrelationsbasierten Indikatorartenanalyse (Dufrene und Legendre, 1997) bestimmt. Indikatorarten werden hier als solche definiert, die sowohl in einem bestimmten Lebensraumtyp häufig (Spezifität) als auch überwiegend in diesem Lebensraumtyp (Treue) vorkommen. In dieser Studie wurden die Indikatortaxa, ähnlich dem Konzept der Indikatorarten, für nicht-salzhaltige Böden und Seesedimente nach einem Indikatorwert (IndVal-Wert) mit dem R-Paket labdsv (Roberts, 2016) ausgewählt, wenn die Wahrscheinlichkeit, einen Indikatorwert zu erhalten, der so hoch ist wie der über die angegebenen Iterationen beobachtete, weniger als 0,05 beträgt. Die Chao2-Indizes wurden für jede Probe mit dem Vegan-Paket berechnet. Der Wilcoxon-Rangsummentest der Chao2-Indizes zwischen Lebensräumen wurde mit der R-Funktion „wilcox.test“ (R Core Team, 2014) durchgeführt. Um die Auswirkungen der Habitatfilterung auf methanogene Archaeen-Isolate zu konsolidieren, wurden die physiologischen und biochemischen Merkmale der beschriebenen methanogenen Kulturen aus der3 „Methanogenic archaea database“ (Jabłoński et al., 2015) abgerufen. Darunter wurden die Isolate mit Informationen zum optimalen NaCl-Bedarf gefiltert, kategorisiert und entsprechend ihrer Isolationsquelle dargestellt.
Um den Einfluss der Ausbreitungsbeschränkung auf die Struktur der methanogenen Gemeinschaft zu untersuchen, wurde eine lineare Regressionsanalyse auf der Grundlage einer geografischen Distanzmatrix und einer Jaccard-Distanzmatrix der Gemeinschaft mit der R-Funktion „lm“ (R Core Team, 2014) durchgeführt. Wir führten Mantel- und partielle Mantel-Tests durch, um die Auswirkungen der Ausbreitungsbeschränkung anhand der beiden Matrizen zu bewerten, wiederum unter Verwendung des Vegan-Pakets in R (Oksanen et al., 2015). Des Weiteren wurde eine multivariate räumliche Analyse (räumliche PCA) auf 16 europäische Boden- und Seesedimentproben auf der Grundlage des Moran’s I-Index angewandt, um die räumliche Struktur von Methanogenen mit der Funktion „multispati“ im R ade4-Paket (Dray und Dufour, 2007) zu untersuchen. Darüber hinaus wurde das Ward’s Minimum variance clustering, das auf der Jaccard-Distanzmatrix basiert, mit der R-Funktion „hclust“ (R Core Team, 2014) für diese 16 Proben implementiert, und wir projizierten die Clustering-Ergebnisse auf eine geografische Karte. Das europäische Shapefile für die Kartierung auf Staatsebene ist in der GSHHG-Datenbank (v2.3.6)4 verfügbar. Die Karte wurde mit QGIS v2.18.25 erstellt.
Ergebnisse
Biogeographie von methanogenen Archaeen in natürlichen Umgebungen
Die mcrA-Gensequenzen wurden aus 94 weltweit verteilten natürlichen Umgebungen abgerufen. Die Lage und der Ökosystemtyp jedes dieser 94 Standorte ist in Abbildung 1 dargestellt. Die Häufigkeit des Auftretens (Vorhandensein/Abwesenheit) der methanogenen Linien wurde je nach Ökosystemtyp zusammengefasst und in Abbildung 2 dargestellt. Kurz gesagt, Methanoregula ist das am häufigsten beobachtete Taxon in Böden, zusammen mit Methanobacterium, Methanosaeta, Methanocella, Methanomassiliicoccus und Methanosarcina. In Mündungssedimenten wurden häufig Sequenzen von Methanosaeta, Methanobacterium, Methanoregula und Methanoculleus nachgewiesen. In Seesedimenten kamen hauptsächlich Methanoregula und Methanosaeta vor. In Meeressedimenten sind Methanoculleus und Methanosaeta die häufigsten Linien, gefolgt von Methanolinea.
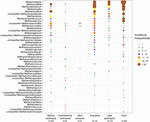
Abbildung 2. Bubble-Plot, der die Häufigkeit der methanogenen Linien in verschiedenen natürlichen Umgebungen zeigt. Die Rangfolge entlang der vertikalen Achse entspricht der abnehmenden Gesamthäufigkeit der Linien. Die Taxonomie ist für die Gattungsebene dargestellt. War eine Zuordnung zur Gattungsebene nicht möglich, wurde die nächsthöhere zuordenbare taxonomische Ebene verwendet. Die Anzahl der Proben (n) ist für jeden Lebensraum angegeben.
Auch wenn viele Taxa in verschiedenen Lebensräumen nachgewiesen wurden, zeigen einige dennoch Umweltpräferenzen. Methanoregula, das Taxon, das häufig in nichtmarinen und Übergangsumgebungen (Böden, Seesedimente und Flussmündungen) vorkommt, fehlt in marinen Lebensräumen (marine Sedimente, hydrothermale Sedimente und Schlammvulkane). Methanobacterium und Methanocella, die in nichtmarinen und Übergangsumgebungen vorherrschen, werden in marinen Lebensräumen nur selten gefunden. Im Gegensatz dazu wird Methanococcoides, das in marinen Sedimenten, hydrothermalen Sedimenten und Schlammvulkanen vorherrscht, in Böden und Seesedimenten kaum beobachtet. Darüber hinaus werden Methanogenium und Methanolacinia nur in Ästuaren und Meeressedimenten beobachtet, während Methanospirillum und Methanosphaerula nur in terrestrischen Umgebungen vorkommen. Darüber hinaus gibt es einige spezifische Taxa, die ausschließlich in hydrothermalen Sedimenten vorkommen, darunter Methanocaldococcus, Methanothermococcus, Methanopyrus, Methanotorris und Methanococcus. Obwohl einige Linien wie Methanosaeta in den meisten Umgebungen vorkommen, kann keine Linie als allgegenwärtig angesehen werden.
Der größte Reichtum an Linien trat in Mündungssedimenten auf, die auch eine gleichmäßigere Häufigkeit der verschiedenen Linien beherbergen. Im Gegensatz dazu weisen Schlammvulkane und hydrothermale Ökosysteme eine relativ geringe methanogene Vielfalt auf. Böden und Seesedimente, die den Ästuaren ähnlich sind, zeichnen sich durch eine vielfältige methanogene Assemblierung aus.
Alpha-Diversität methanogener Gemeinschaften in natürlichen Umgebungen
Der Reichtum methanogener Archaeen gemäß dem Chao2-Index variierte stark zwischen den Ökosystemtypen (Abbildung 3). Um die Alpha-Diversität direkt zu vergleichen und einen angemessenen Kompromiss zwischen den Proben zu erzielen, wurde für jeden Standort eine Unterstichprobe von 15 Sequenzen durchgeführt. Der Chao2-Index zeigt, dass Ästuarsedimente den höchsten Artenreichtum an Methanogenen entlang der sechs Ökosystemtypen aufweisen (ergänzende Tabelle S3), was die Ergebnisse des Bubble Plots (Abbildung 2) unterstreicht. Böden und Seesedimente, die einen geringeren Artenreichtum als Ästuarproben aufweisen, haben signifikant höhere Chao2-Indizes als marine Sedimente und hydrothermale Sedimente, wobei es keine signifikanten Unterschiede zwischen marinen Sedimenten und hydrothermalen Sedimenten gibt (ergänzende Tabelle S3).
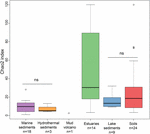
Abbildung 3: Boxplot der Chao2-Indizes der verschiedenen Ökosystemtypen. Das Diagramm basiert auf unterbeprobten Datensätzen mit 15 Sequenzen für jeden Standort, um den Vergleich der Alpha-Diversitätsmaße robuster zu machen. Die Anzahl der Proben in jedem Habitat ist als „n“ unter der Habitatbezeichnung angegeben. Das „ns“ bedeutet keine statistische Signifikanz im Wilcoxon-Test. Das statistische Ergebnis der Alpha-Diversität auf OTU-Ebene ist in der ergänzenden Tabelle S3 angegeben.
Globale Kontrollen von Methanogen-Gemeinschaften in natürlichen Umgebungen
Die 94 global verteilten Methanogen-Gemeinschaften wurden durch Anwendung von PCoA auf der Grundlage der Jaccard-Distanzmatrix in einem Ordinationsdiagramm geclustert. Nach der PCoA-Analyse erklären die erste und zweite Achse zusammen 16,3 % der Gesamtvarianz. Die Unterschiede zwischen den Proben lassen sich somit weitgehend durch den Salzgehalt erklären (Abbildung 4). Da in einigen Fällen keine Ausgangsdaten zu den Salzkonzentrationen vorlagen, haben wir diese Proben wie oben beschrieben qualitativ als salzhaltige, gemischte (intermediäre) und nicht salzhaltige Proben eingestuft. Die salzhaltigen und die nicht salzhaltigen Proben sind entlang der ersten Achse effektiv getrennt. Die Mischproben liegen insgesamt zwischen den salzhaltigen und den nicht salzhaltigen Proben. Die Permutations-MANOVA auf der Grundlage der Jaccard-Distanzmatrix deutet ebenfalls darauf hin, dass der Salzgehalt der primäre abiotische Faktor ist, der die Verteilung der globalen methanogenen Gemeinschaften steuert (R2 = 0,099, P < 0,001) (Tabelle 1).
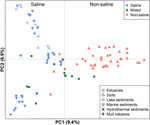
Abbildung 4. Hauptkoordinatenanalyse (PCoA) Ordination basierend auf der Jaccard-Distanzmatrix von methanogenen mcrA-Gen-Sequenzbibliotheken, die 94 Proben vergleichen. Die PCoA ist nach dem Salzgehalt eingefärbt: Die roten Symbole stehen für nicht-salzhaltige Umgebungen, die blauen für salzhaltige Umgebungen und die grünen für intermediäre Umgebungen. Verschiedene Symbole stehen für unterschiedliche Umgebungen. Der prozentuale Anteil der Variation, der durch die aufgezeichneten Hauptkoordinaten erklärt wird, ist auf den Achsen angegeben.

TABLE 1. Permutations-MANOVA-Analyse der Jaccard-Distanzmatrix aller Proben aus den sechs Habitaten, um die Assoziation der Gemeinschaftsvarianz mit verschiedenen Umweltvariablen zu testen.
Zusätzlich wurde eine mögliche Beziehung zwischen der Isolationsquelle der methanogenen Reinkulturen und der optimalen NaCl-Konzentration für das Wachstum untersucht. Die optimale NaCl-Konzentration der methanogenen Reinkulturen zeigte einen Rückgang von marinen zu Ästuaren, Seesedimenten und Bodenökosystemen (ergänzende Abbildung S2). Einige wenige Ausreißerisolate stammen aus Natronseesedimenten oder hypersalinen Böden.
Umweltkontrollen und methanogene Indikatortaxa in nicht-salinen Böden und Seesedimenten
Auf globaler Ebene sind die methanogenen Gemeinschaften aus nicht-salinen Böden und Seesedimenten eng geclustert (Abbildung 4), so dass wir die Umweltkontrollen der methanogenen Gemeinschaften aus diesen beiden Lebensräumen, die insgesamt 33 Untersuchungsstandorte ausmachen, weiter analysiert haben. Die gemeinschaftsbasierte Clusteranalyse für diese beiden Lebensraumtypen ergab vier Cluster auf der Grundlage der Jaccard-Abstandsmatrix (ergänzende Abbildung S3). Die Permutations-MANOVA deutet darauf hin, dass sowohl der pH-Wert (R2 = 0,099, P < 0,001) als auch die Temperatur (R2 = 0,069, P < 0,001) die methanogene β-Diversität in nicht-salzhaltigen Böden und Seesedimenten beeinflussen (Tabelle 2). Dementsprechend ordneten wir die vier Cluster dem pH-Wert und dem MAAT-Wert der ursprünglichen Probenahmestelle zu und erhielten weitgehend konsistente Untergruppen für die Clusterung der Gemeinschaft (Abbildungen 5C und D). Die Kombination von Umweltmerkmalen und diesen vier Gemeinschaftsclustern ermöglicht es uns, diese vier Untergruppen als neutrale und kalte Gruppe 1, saure und kalte Gruppe 2, saure und moderate Gruppe 3 und neutrale und warme Gruppe 4 zu definieren (Abbildung 5). Eine weitere PCoA-Ordnung auf der Grundlage der Jaccard-Dissimilaritätsmatrix deutet darauf hin, dass entlang der PC1-Achse die meisten Proben aus Gruppe 2 und Gruppe 3 aus sauren Böden und Seesedimenten stammen, während Gruppe 1 und Gruppe 4 hauptsächlich aus neutralen Umgebungen stammen (Abbildung 5A). Darüber hinaus trennten sich die Proben aus gemäßigten Standorten (Gruppe 3) von denen aus warmen und kalten Standorten entlang der PC2-Achse, während sich die Proben aus warmen Umgebungen (Gruppe 4) von den anderen Proben entlang der PC3-Achse trennten (Abbildung 5B). Dadurch erklären die ersten drei Achsen der PCoA-Ordnung 38,8 % der Gesamtvariation.

TABLE 2. Permutations-MANOVA basierend auf einer Jaccard-Distanzmatrix von nicht-salzhaltigen Boden- und Seesedimentproben, um die Assoziation der Gemeinschaftsvarianz mit verschiedenen Umweltvariablen zu testen.
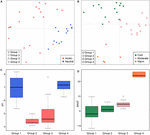
Abbildung 5. Plot der Hauptkoordinatenanalyse basierend auf einer Jaccard-Distanzmatrix für 33 nicht-saline Böden und Seesedimente. Subplot (A) zeigt PC1 und PC2 und die Symbole sind nach pH-Wert gefärbt, und Subplot (B) zeigt PC2 und PC3 und die Symbole sind nach Temperatur gefärbt. Die ersten drei Komponenten erklären 17,1, 11,7 und 10 % der Varianz. Die Boxplots zeigen den pH-Wert (C) und die MAAT (mittlere jährliche Lufttemperatur) (D) von vier identifizierten Untergruppen. Die Farbe der Box in Abbildung C entspricht der pH-Kategorie in (A). Die Farben der Boxplots zeigen die statistische Signifikanz auf der Grundlage eines paarweisen Wilcoxon-Tests (P < 0,05), wobei sich die Proben mit der gleichen Farbe nicht signifikant voneinander unterscheiden. In ähnlicher Weise folgt die Farbe in (D) der Temperaturgruppierung in (B). Die Untergruppen beziehen sich auf die hierarchische Clusteranalyse der Gemeinschaftsähnlichkeiten.
Wir untersuchten das Vorkommen methanogener Linien in jeder Untergruppe auf der Grundlage der Häufigkeit des Auftretens (Abbildung 6). Methanoregula überwiegt in allen Arten von nicht-salzhaltigen Habitaten. Zusätzlich zu Methanoregula weist die neutrale und kalte Untergruppe (Gruppe 1) eine hohe Häufigkeit von Methanosaeta, Methanobacterium und Methanosarcina auf. Die saure und kalte Gruppe 2 wird von Methanobacterium, Methanocella und Methanosarcina repräsentiert, während Methanosaeta hier nicht vorkommt. Methanocella und Methanosaeta sind in der sauren und gemäßigten Gruppe 3 verbreitet. In der neutralen und warmen Gruppe 4 sind Methanolinea und Methanosaeta wichtige Mitglieder. Diese Gruppe ist die einzige, in der Methanoculleus identifiziert wurde.

Abbildung 6: Häufigkeit der methanogenen Linien innerhalb der vier definierten Gruppen von methanogenen Gemeinschaften in Böden und Seesedimenten. Die vertikale Achse ist in alphabetischer Reihenfolge angeordnet. Die Balkenlänge entspricht den durchschnittlichen Häufigkeiten für jede Linie innerhalb der entsprechenden Gruppe. Die Fehlerbalken stellen die Standardabweichung eines bestimmten Taxons über verschiedene Proben in dieser Gruppe dar. Die Sternchen zeigen die spezialisierten Taxa mit einem P-Wert < 0,05. Die Taxonomie ist für die Gattungsebene angegeben. Wenn eine Zuordnung zur Gattungsebene nicht möglich war, wurde die nächsthöhere zuordenbare taxonomische Ebene verwendet. Die Beschreibung für die vier Gruppen findet sich in Abbildung 5.
Für alle vier Gruppen sind die Taxa mit hohem Vorkommen Methanoregula, Methanobacterium, Methanosarcina, Methanosaeta, Methanomassiliicoccus und Methanocella. Die spezialisierten Taxa, die an den meisten Standorten innerhalb einer bestimmten Gruppe deutlich stärker vertreten sind, wurden wie zuvor beschrieben anhand der Analyse der Indikatorarten ermittelt. Insgesamt zeigten sechs der 31 Taxa einen signifikanten Indikatorwert (P < 0,05) (in Abbildung 6 mit einem Sternchen gekennzeichnet). Gruppe 1 (neutral und kalt) wies die größte Anzahl von Spezialisten mit Linien von Methanosaeta, Methanolobus und Methanomethylovorans auf. Methanobacterium diente als spezialisiertes Taxon in der kalten und sauren Gruppe 2, während Methanolinea als Spezialist in Gruppe 4 (neutral und warm) identifiziert wurde, aber in den anderen Gruppen kaum zu finden war. Darüber hinaus ist Methanoregula in der sauren und gemäßigten Gruppe 3 stark vertreten.
Dispersionsbeschränkung
Eine lineare Regressionsanalyse (R2= 0,05, P < 0,001) deutete auf eine schwache Korrelation zwischen der geografischen Entfernung und der Struktur der methanogenen Gemeinschaft im globalen Datensatz hin. Gleichzeitig zeigte ein Mantel-Test, dass die Umweltvariablen eine höhere Korrelation mit der Gemeinschaftsstruktur aufweisen als die geografischen Entfernungen (siehe Tabelle 3). Dieser Trend wird auch durch einen partiellen Mantel-Test bestätigt, der die Autokorrelationseffekte kontrolliert. Das Auftragen der geographischen Entfernung gegen die Jaccard-Gemeinschaftsähnlichkeit zeigt keinen klaren linearen Trend, sondern Muster, die hauptsächlich aus der globalen Verteilung der Probenahmepunkte resultieren (siehe ergänzende Abbildung S4).

TABELLE 3. Mantel- und partielle Mantel-Test-Analysen zur Bestimmung des Einflusses von Umweltvariablen und geografischer Entfernung auf die mikrobielle Verteilung für den globalen Datensatz und eine Teilstichprobe von 16 europäischen Proben.
Um den Einfluss der Ausbreitung weiter zu analysieren, beschränkten wir unsere Analyse auf Europa, das am dichtesten und gleichmäßigsten beprobt wurde. Mantel-Tests und partielle Mantel-Tests für diese Untergruppe bestätigten den Trend, dass die Gemeinschaftsdaten stärker mit den Umweltvariablen als mit den geografischen Entfernungen korreliert sind (siehe Tabelle 3). Der partielle Mantel-Test, der die Umweltvariablen kontrolliert, konnte keine statistisch signifikante Korrelation zwischen der mikrobiellen Gemeinschaft und der geografischen Entfernung feststellen. Eine räumliche PCA-Analyse dieser 16 europäischen Standorte deutet auf eine räumliche Struktur der methanogenen Gemeinschaft hin (23,7 % der Gesamtvarianz wurde durch diese Struktur erklärt), die einer positiven räumlichen Autokorrelation der Standorte entspricht, wie der Moran’s I-Index zeigt (Moran’s I = 0,4018). Nur der erste Eigenwert war stabil und entsprach einer Trennung der Proben zwischen Mitteleuropa und den baltischen Staaten (ergänzende Abbildung S5). Der kleine Datensatz erschwert jedoch eine robuste Zuordnung dieser beobachteten räumlichen Struktur zu geografischen oder Umweltvariablen oder zu beiden. Wir haben daher wie oben beschrieben eine Clusteranalyse der methanogenen Gemeinschaften durchgeführt und drei Gruppen ermittelt (Abbildung 7A), die wir auf eine geografische Karte projiziert haben (Abbildung 7B). Die Clusterbildung gab die Trennung der räumlichen PCA entlang der Ostsee nicht wieder. Dementsprechend weisen einige Standorte, die geografisch sehr nahe beieinander liegen, unterschiedliche Strukturen der methanogenen Gemeinschaft auf und werden in verschiedenen Clustern zusammengefasst. Andererseits weisen einige geografisch sehr weit entfernte Standorte sehr ähnliche Gemeinschaftsstrukturen auf und gruppieren sich zusammen (Abbildung 7B). Die regionale Ausbreitung der Gruppen in Europa scheint nicht einer geographischen Struktur zu entsprechen oder durch diese begrenzt zu sein.
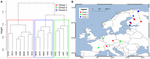
Abbildung 7. Hierarchische Clusteranalyse der Jaccard-Distanzmatrix zwischen 16 europäischen Boden- und Seesedimentproben. Für die Analyse wurde die Ward-Clustermethode verwendet. Die Standorte wurden in drei Gruppen eingeteilt, die durch unterschiedliche Rechtecke im Cluster-Dendrogramm (A) gefärbt und auf eine europäische Karte (B) projiziert wurden.
Diskussion
Die Identifizierung und Anwendung von Konzepten der Biogeographie auf mikrobielle Gemeinschaften ist von großem Interesse in der mikrobiellen Ökologie. Es wird angenommen, dass die mikrobielle Biogeografie durch das evolutionäre und ökologische Zusammenspiel von vier Hauptprozessen bestimmt wird: Habitatfilterung, Ausbreitung, Drift und Mutation (Hanson et al., 2012). Auch wenn der Einfluss von Drift und Mutation die Interpretationsmöglichkeiten dieser Studie übersteigt, zeigen wir, dass es globale Muster methanogener Gemeinschaften in natürlichen Umgebungen gibt. Diese Studie zeigt ein globales biogeografisches Muster methanogener Gemeinschaften, das eher mit der Filterung von Lebensräumen als mit geografischer Ausbreitung zusammenhängt. Methanogene Gemeinschaften aus Sedimenten von Sodaseen sind beispielsweise eng mit geografisch weit entfernten Meeresproben verbunden (Abbildung 4), und in europäischen Boden- und Seesedimenten treten sehr ähnliche methanogene Gemeinschaften auf, obwohl sie weit voneinander entfernt sind (Abbildung 7). Insgesamt ergaben unsere Versuche, mögliche Ausbreitungsbeschränkungen aufzudecken, einen schwachen Einfluss der geografischen Lage auf die Struktur der methanogenen Gemeinschaften, dem ein deutlicher Einfluss der Umweltbedingungen gegenübersteht. Ein schlüssiger Ausschluss von räumlichen Effekten auf die mikrobiellen Gemeinschaften ist mit den verfügbaren Datenpunkten nicht möglich. Die Probenahmepunkte konzentrieren sich auf bestimmte Lebensräume und/oder Gebiete, während die Gesamtzahl der Proben gering ist. Falls es einen Effekt gibt, gehen wir davon aus, dass räumliche Effekte nur auf regionaler oder lokaler Ebene auftreten. Lokale Ausbreitungsbeschränkungen wurden für hyperthermophile Archaeen aufgrund geografischer Barrieren (Whitaker et al., 2003), für ammoniakoxidierende Bakterien in lokalen Salzwiesen, aber nicht auf regionaler oder kontinentaler Ebene (Martiny et al., 2011), für Mikroorganismen in Tiefseesedimenten in Verbindung mit Umweltbedingungen (Schauer et al., 2009), und für Bakterien einer großen Anzahl heterogener Schneeumgebungen, die hauptsächlich durch die Verfügbarkeit von allochthonem Kohlenstoff verursacht werden (Lutz et al., 2016). Ein geringer Einfluss der Ausbreitungsbeschränkung auf Methanogene in natürlichen Umgebungen bedeutet, dass sich Methanogene zufällig über den Raum verteilen können, von der lokalen Umgebung erfolgreich selektiert werden, wenn ihre physiologischen Anforderungen erfüllt sind, und stabile Gemeinschaften bilden können (Martiny et al., 2006; von Mering et al., 2007).
Das Konzept der Habitatfilterung impliziert, dass Arten mit ähnlichen ökologischen Anforderungen häufiger als zufällig erwartet gemeinsam vorkommen sollten (Weiher und Keddy, 2001; Cornwell et al., 2006; Ulrich et al., 2010). Unsere Ergebnisse zeigen, dass große Unterschiede in der Zusammensetzung der Methanogen-Gemeinschaften zwischen Meeres-, Feuchtgebiets- und Seeökosystemen bestehen, während Ästuare dazwischen liegen. Biogeografische Muster, die auf einer Reihe von Genuntersuchungen an Umweltproben basieren, wurden auch für allgemeine Bakterien (Nemergut et al., 2011), das Mikrobiom von Methanquellen (Ruff et al., 2015), ammoniakoxidierende Archaeen (Cao et al., 2013), marine pelagische und benthische Bakterien (Zinger et al., 2011) und stickstoffzyklische Mikroorganismen (Church et al., 2008) berichtet. Die Filterung von Lebensräumen wurde speziell für nicht kultivierte Archaeen (Auguet et al., 2010), ganze bakterielle Gemeinschaften in verschiedenen Umgebungen (von Mering et al., 2007; Chaffron et al., 2010) oder in südamerikanischen Torfgebieten auf regionaler Ebene (Oloo et al., 2016) sowie spezifische Bakteriengruppen wie methanoxidierende (Knief, 2015) und stickstofffixierende Bakterien (Nelson et al., 2016).
Unsere Ergebnisse deuten darauf hin, dass der Salzgehalt auf globaler Ebene die Zusammensetzung der Methanogen-Gemeinschaft wesentlich beeinflusst und große Unterschiede zwischen marinen und terrestrischen Methanogen-Gemeinschaften bestimmt. Auch die Methanogene aus den Sedimenten von Sodaseen sind in einer Gruppe mit denen aus Meeressedimenten vertreten (Abbildung 4), was den globalen Einfluss des Salzgehalts verdeutlicht. Dieses Ergebnis steht im Einklang mit anderen Studien auf der Grundlage des 16S rRNA-Gens, die zeigen, dass der Salzgehalt ein Hauptfaktor ist, der die globalen Muster der gesamten bakteriellen und archäologischen Gemeinschaften prägt (Lozupone und Knight, 2007; Auguet et al., 2010; Caporaso et al., 2011; Cao et al., 2013). Ein geringer Einfluss der geografischen Trennung, aber ein starker Einfluss des Salzgehalts auf die allgemeinen mikrobiellen Gemeinschaften wurde auch in früheren Studien beobachtet (Logares et al., 2013; Yang et al., 2016). Demnach bestimmt der Salzgehalt weitgehend, welche Linien überleben können. In verschiedenen Lebensräumen war die Aktivität der Methanproduktion negativ mit dem Salzgehalt korreliert (Bartlett et al., 1987; Potter et al., 2009; Poffenbarger et al., 2011). Es wird angenommen, dass die Hemmung der Methanproduktion durch den Salzgehalt mit einer geringeren Größe der methanogenen Population einhergeht (Pattnaik et al., 2000). Die Wirkung des Salzgehalts auf die hydrogenotrophe, acetotrophe und methylotrophe Methanogenese hängt dabei von der Höhe des Salzgehalts ab und ist für die verschiedenen Wege der Methanogenese unterschiedlich (Liu et al., 2016). Derzeit gibt es keinen eindeutigen Mechanismus zur Erklärung der Auswirkungen des Salzgehalts auf die Struktur der Gemeinschaft, aber mehrere Hypothesen können als mögliche Erklärung dienen. Physiologisch gesehen beeinflusst der Salzgehalt die externe und interne Osmolarität der Zellen. Die nicht salzhaltigen methanogenen Zellen haben physiologische Anpassungen entwickelt, um dem internen Turgordruck entgegenzuwirken, während die salzangepassten Zellen diese Eigenschaft verloren haben (Zinder, 1993). Darüber hinaus kann ein zunehmender Salzgehalt Methanogene dazu veranlassen, einen größeren Anteil kompatibler gelöster Stoffe zu synthetisieren oder aufzunehmen, was mit erheblichen energetischen und damit metabolischen Kosten verbunden ist (McGenity, 2010). Die Eigenschaft der Salztoleranz zeigt sich sogar in der optimalen NaCl-Konzentration für das Wachstum methanogener Reinkulturen, da wir festgestellt haben, dass die Isolate aus marinen Sedimenten und hydrothermalen Sedimenten eine deutlich höhere optimale NaCl-Konzentration aufweisen als die aus Böden (ergänzende Abbildung S2).
In nicht-salzhaltigen terrestrischen Ökosystemen, insbesondere in Böden und Seesedimenten, wird die Zusammensetzung der methanogenen Gemeinschaft durch die Kombination von Temperatur und pH-Wert gesteuert. Dementsprechend lassen sich die Methanogene in diesen Ökosystemen in vier Gruppen einteilen (Abbildung 6). Im Gegensatz zu den marinen Ökosystemen weisen die nicht-salzhaltigen terrestrischen Ökosysteme eine große natürliche Variabilität sowohl des pH-Werts als auch der Temperatur auf. Die Temperatur kann nicht nur den Methanbildungsweg, sondern auch die methanbildenden Populationen selbst beeinflussen (Conrad, 2007; Rooney-Varga et al., 2007). Die Methanproduktion kann erheblich gesteigert werden, wenn die Temperatur als Folge der temperaturempfindlichen Schritte während der Fermentation und Acetogenese steigt (Megonigal et al., 2005; Kotsyurbenko et al., 2007). Darüber hinaus kann ein niedriger pH-Wert die Verfügbarkeit von Acetat erheblich einschränken, indem er die Dissoziation von Acetat verhindert und somit die acetoklastische Methanogenese negativ beeinflusst (Fukuzaki et al., 1990; Bridgham et al., 2013). Dies könnte ein möglicher Grund dafür sein, dass Methanosaeta in der Gruppe 2 fehlte, während Methanosarcina zwischen verschiedenen Quellen wechseln kann und nicht wesentlich beeinflusst wurde. Darüber hinaus kann der pH-Wert die Effizienz der Methanproduktion und der methanogenen Wege von ombrotrophen zu minerotrophen Mooren regulieren, und zwar durch direkte Hemmung beider Methanogenesewege und indirekt durch seine Auswirkungen auf die Fermentation (Ye et al., 2012). Daher können sowohl die Temperatur als auch der pH-Wert direkt oder indirekt die mit der Methanogenese verbundenen Stoffwechselschritte und die vorgelagerte Gärung regulieren, die das Substrat für Methanogene liefert.
Methanoregula ist in allen vier Gruppen terrestrischer Lebensräume allgegenwärtig und sehr häufig (Abbildung 6), aber im marinen System praktisch nicht vorhanden und könnte sich daher als Stellvertreter für den Einfluss von Süßwasser im marinen Bereich erweisen. Über seine globale Bedeutung wurde kürzlich an anderer Stelle berichtet (Yang et al., 2017). Trotz ihrer ubiquitären Verbreitung in Böden und Seesedimenten tritt Methanoregula als Indikatorlinie in sauren Lebensräumen mit moderaten Temperaturen auf. Darüber hinaus scheint sich (1) Methanolinea besonders an warme, neutrale terrestrische Umgebungen angepasst zu haben, (2) Methanobacterium an kalte, saure Umgebungen und (3) Methanosaeta an pH-neutrale Umgebungen, was mit anderen Studien übereinstimmt (Rosenberg et al., 2014) und die Robustheit unseres Ansatzes unterstreicht. Im Allgemeinen führen die geochemischen Bedingungen in der Umgebung von methanogenen Gemeinschaften zu einer Nischendifferenzierung. Da die Nischensortierung dazu neigt, die anpassungsfähigen Spezialisten zu verlassen (Langenheder und Székely, 2011), hat die fortschreitende langfristige Umweltselektion eine Vielzahl von Nischen hervorgebracht, die von einer Reihe von endemischen Habitatspezialisten besetzt wurden, die unter anderen, unterschiedlichen Umweltbedingungen weniger stark vertreten oder nicht vorhanden sind. Die Gemeinschaft wird auch durch biotische Faktoren wie ökologische Interaktionen, Dynamik, Wettbewerb und Symbiose geprägt. Trotz dieser biologischen Faktoren stellten von Mering et al. (2007) fest, dass die Habitatpräferenzen im Laufe der Zeit oft bemerkenswert stabil sind und dass die charakteristische taxonomische Zusammensetzung von Umweltgemeinschaften wiederum ein wichtiger Indikator für ihre Ökologie und Funktion sein kann.
In Übereinstimmung mit der Habitatpräferenz von methanogenen Archaeen scheint es, dass eng verwandte methanogene Stämme oft aus vergleichbaren Umgebungen isoliert wurden. Zum Beispiel scheinen Methanoregulaceae in natürlichen Umgebungen recht vielfältig zu sein (Yang et al., 2017), und neutrale Stämme können der Kultivierung bisher widerstehen. Die derzeit beschriebenen Stämme von Methanoregula stammen beide aus leicht acidophilen Umgebungen, während die beiden Vertreter von Methanolinea aus relativ warmen Lebensräumen wie Faulschlamm bzw. Reisfeldboden stammen (Rosenberg et al., 2014). Obwohl Methanobacterium-Stämme aus verschiedenen Umgebungen isoliert wurden, weist etwa die Hälfte der vorhandenen Isolate dieser Gattung pH-Optima von etwas weniger als 7 auf. Das bedeutet, dass die Indikatorlinien, die anhand der Umgebungssequenzen identifiziert wurden, die Unterschiede in der Physiologie und den Quellen der vorhandenen methanogenen Kulturen widerspiegeln könnten. Beispielsweise kann der Salzgehalt des Lebensraums als allgemeine Eigenschaft des Lebensraums die Organismen nach und nach einer starken Umweltselektion aussetzen und den Aufbau einer neuen Reihe von Arten herausfiltern, die am besten für den Salzgehalt der Umgebung geeignet sind (Logares et al., 2013).
Die Erhaltung und Bewirtschaftung der biologischen Vielfalt ist eine der wichtigsten Herausforderungen unserer heutigen Gesellschaft. Hier zeigen wir, dass methanogene Archaeen in natürlichen Umgebungen in Ästuarsedimenten am vielfältigsten sind. Ästuare sind Übergangszonen zwischen marinen und terrestrischen Ökosystemen. Dies ermöglicht zwei wichtige Prozesse, die zum Artenreichtum beitragen können. Erstens vermischen sich in den Ästuaren Mikroben aus dem Meer und vom Land und bilden schließlich eine große Vielfalt (McLusky und Elliott, 2004). So wurde beispielsweise eine hohe Vielfalt von Ästuarbakterien, Archaeen, Pilzen und sogar spezifischen Bakterien mit einzigartigen Funktionen beobachtet (Cunliffe et al., 2008; Mosier und Francis, 2008; Crump et al., 2012). Ein weiterer Aspekt ist der hohe Nährstoffgehalt aufgrund der terrestrischen, gezeitenbedingten Einträge, von denen sich die Organismen des Ästuars ernähren (McLusky und Elliott, 2004; Statham, 2012). In diesem Zusammenhang sind die Mündungsgebiete von Bedeutung für die Wiederherstellung der Gattungsneutralität von Methanogenen. Die Auswirkungen der Artenvielfalt auf die Ökosystemprozesse sind bisher Gegenstand umfangreicher Forschungsarbeiten gewesen. Der Zusammenhang zwischen Biodiversität und Ökosystemfunktion wird immer noch diskutiert und bleibt für mikrobielle Gemeinschaften schwer fassbar (Loreau et al., 2001; Tilman et al., 2014). Obwohl Böden und Seesedimente primäre Methanquellen und auch Lebensräume mit hoher methanogener Vielfalt sind, schlagen wir vor, dass der Artenreichtum kein geeigneter Indikator für das Methanproduktionspotenzial und die Methanemissionen des Ökosystems ist; er scheint vielmehr die Heterogenität und Geschichte der Umwelt widerzuspiegeln. Die Einstufung der Umgebungen nach ihrem Artenreichtum bedeutet nicht notwendigerweise das Potenzial der Methanemissionsraten, die in Böden und Seen am höchsten und in Ästuaren vergleichsweise gering sind (Abbildung 3 und ergänzende Abbildung S1).
Schließlich könnte der Mangel an Umweltinformationen in den öffentlichen Datenbanken eine vollständige Interpretation der hier beobachteten Umweltfaktoren erschwert haben. Obwohl in der Literatur und in öffentlichen Datenbanken immer mehr Sequenzierungsdaten zu Methanogenen zu finden sind, sind die damit verbundenen abiotischen Variablen oft uneinheitlich und spärlich vorhanden. Die begrenzte Menge an konsistenten Informationen für Umweltvariablen schränkt die Anwendung multivariater statistischer Analysen ein. Wenn man bedenkt, dass die abiotischen Faktoren in dieser Studie nur einen begrenzten Teil der Varianz der Lebensgemeinschaften erklären können, liegt der Schluss nahe, dass andere erklärende Variablen fehlen. Von besonderer Bedeutung könnten die Konzentrationen und die Verfügbarkeit von methanogenen Substraten wie Acetat, Wasserstoff und Methylaminen sein. Dennoch reichen abiotische Parameter möglicherweise nie aus, um die Struktur der methanogenen Assemblagen vollständig zu erklären, einfach weil die Lebensräume eine unterschiedliche Geschichte haben und nur lokal untersucht werden können. Außerdem sind in den verfügbaren mcrA-Daten die riesigen Gebiete der russischen und kanadischen Subarktis und Arktis nur unzureichend vertreten. Eine bessere geografische Abdeckung und gleichmäßige Verteilung der mcrA-Gendaten würde die Bewertung der methanogenen Gemeinschaften auf globaler Ebene verbessern.
Beiträge der Autoren
SL und XW entwarfen die Studie. XW und SY sammelten und analysierten die Daten. XW, SY und FH führten die statistische Analyse durch. XW, MW und SY führten die phylogenetische Korrektur durch. XW, SY, FH, MW, DW und SL interpretierten die Ergebnisse und schrieben die Arbeit. Alle Autoren trugen zu den Diskussionen bei und überprüften das Manuskript.
Erklärung zu Interessenkonflikten
Die Autoren erklären, dass die Forschung in Abwesenheit jeglicher kommerzieller oder finanzieller Beziehungen durchgeführt wurde, die als potenzieller Interessenkonflikt ausgelegt werden könnten.
Danksagungen
Die finanzielle Unterstützung für XW (Stipendium Nr. 201408620031 für XW) durch den China Scholarship Council (CSC) wird dankend anerkannt. Diese Studie wurde von der Helmholtz-Gemeinschaft (HGF) durch die Förderung der Helmholtz-Nachwuchsgruppe der SL (VH-NG-919) unterstützt.
Supplementary Material
Das Supplementary Material zu diesem Artikel finden Sie online unter: https://www.frontiersin.org/article/10.3389/fmicb.2017.01339/full#supplementary-material
Fußnoten
- ^ http://metanogen.biotech.uni.wroc.pl/
- ^ http://www.ncbi.nlm.nih.gov/
- ^ http://metanogen.biotech.uni.wroc.pl/
- ^ https://www.ngdc.noaa.gov/mgg/shorelines/gshhs.html
- ^ http://qgis.osgeo.org
Auguet, J. C., Barberan, A., und Casamayor, E. O. (2010). Globale ökologische Muster in unkultivierten Archaea. ISME J. 4, 182-190. doi: 10.1038/ismej.2009.109
PubMed Abstract | CrossRef Full Text | Google Scholar
Bange, H. W., Bartell, U. H., Rapsomanikis, S., and Andreae, M. O. (1994). Methan in der Ost- und Nordsee und eine Neubewertung der marinen Methanemissionen. Glob. Biogeochem. Cycles 8, 465-480. doi: 10.1029/94GB02181
CrossRef Full Text | Google Scholar
Barreto, D. P., Conrad, R., Klose, M., Claus, P., and Enrich-Prast, A. (2014). Distance-decay and taxa-area relationships for bacteria, archaea and methanogenic archaea in a tropical lake sediment. PLoS ONE 9:e110128. doi: 10.1371/journal.pone.0110128
PubMed Abstract | CrossRef Full Text | Google Scholar
Bartlett, K. B., Bartlett, D. S., Harriss, R. C., and Sebacher, D. I. (1987). Methanemissionen entlang eines Salzwiesen-Salzgehaltsgradienten. Biogeochemistry 4, 183-202. doi: 10.1007/BF02187365
CrossRef Full Text | Google Scholar
Borrel, G., O’Toole, P. W., Harris, H. M. B., Peyret, P., Brugere, J. F., and Gribaldo, S. (2013). Phylogenomische Daten belegen eine siebte Ordnung der methylotrophen Methanogene und geben Einblicke in die Evolution der Methanogenese. Genome Biol. Evol. 5, 1769-1780. doi: 10.1093/Gbe/Evt128
PubMed Abstract | CrossRef Full Text | Google Scholar
Bousquet, P., Ciais, P., Miller, J., Dlugokencky, E., Hauglustaine, D., Prigent, C., et al. (2006). Beitrag anthropogener und natürlicher Quellen zur Variabilität des atmosphärischen Methans. Nature 443, 439-443. doi: 10.1038/nature05132
PubMed Abstract | CrossRef Full Text | Google Scholar
Bridgham, S. D., Cadillo-Quiroz, H., Keller, J. K., and Zhuang, Q. (2013). Methanemissionen aus Feuchtgebieten: biogeochemische, mikrobielle und Modellierungsperspektiven vom lokalen bis zum globalen Maßstab. Glob. Chang. Biol. 19, 1325-1346. doi: 10.1111/gcb.12131
PubMed Abstract | CrossRef Full Text | Google Scholar
Cao, H., Auguet, J.-C., and Gu, J.-D. (2013). Globales ökologisches Muster von Ammoniak-oxidierenden Archaeen. PLoS ONE 8:e52853. doi: 10.1371/journal.pone.0052853
PubMed Abstract | CrossRef Full Text | Google Scholar
Caporaso, J. G., Lauber, C. L., Walters, W. A., Berg-Lyons, D., Lozupone, C. A., Turnbaugh, P. J., et al. (2011). Globale Muster der 16S rRNA-Diversität bei einer Tiefe von Millionen von Sequenzen pro Probe. Proc. Natl. Acad. Sci. U.S.A. 108, 4516-4522. doi: 10.1073/pnas.1000080107
PubMed Abstract | CrossRef Full Text | Google Scholar
Chaffron, S., Rehrauer, H., Pernthaler, J., and von Mering, C. (2010). Ein globales Netzwerk von koexistierenden Mikroben aus Umwelt- und Ganzgenomsequenzdaten. Genome Res. 20, 947-959. doi: 10.1101/gr.104521.109
PubMed Abstract | CrossRef Full Text | Google Scholar
Church, M. J., Bjorkman, K. M., Karl, D. M., Saito, M. A., and Zehr, J. P. (2008). Regionale Verteilung von stickstofffixierenden Bakterien im Pazifischen Ozean. Limnol. Oceanogr. 53, 63-77. doi: 10.4319/lo.2008.53.1.0063
CrossRef Full Text | Google Scholar
Conrad, R. (2007). „Microbial ecology of methanogens and methanotrophs“, in Advances in Agronomy, ed. L. S. Donald (Cambridge, MA: Academic Press), 1-63.
Google Scholar
Cornwell, W. K., Schwilk, D. W., und Ackerly, D. D. (2006). Ein eigenschaftsbasierter Test für Habitatfilterung: Konvexes Hüllenvolumen. Ecology 87, 1465-1471. doi: 10.1890/0012-9658(2006)872.0.CO;2
PubMed Abstract | CrossRef Full Text | Google Scholar
Crump, B. C., Amaral-Zettler, L. A., and Kling, G. W. (2012). Mikrobielle Vielfalt in arktischen Süßgewässern wird durch Beimpfung von Mikroben aus Böden strukturiert. ISME J. 6, 1629-1639. doi: 10.1038/ismej.2012.9
PubMed Abstract | CrossRef Full Text | Google Scholar
Cui, M., Ma, A., Qi, H., Zhuang, X., Zhuang, G., and Zhao, G. (2015). Wärmere Temperaturen beschleunigen die Methanemissionen aus dem Zoige-Feuchtgebiet auf dem Tibetischen Plateau, ohne die Zusammensetzung der methanogenen Gemeinschaft zu verändern. Sci. Rep. 5:11616. doi: 10.1038/srep11616
PubMed Abstract | CrossRef Full Text | Google Scholar
Cunliffe, M., Schafer, H., Harrison, E., Cleave, S., Upstill-Goddard, R., and Murrell, J. C. (2008). Phylogenetische und funktionelle Genanalyse der bakteriellen und archaeischen Gemeinschaften, die mit der Oberflächenmikroschicht eines Ästuars verbunden sind. ISME J. 2, 776-789. doi: 10.1038/ismej.2008.28
PubMed Abstract | CrossRef Full Text | Google Scholar
Dray, S., and Dufour, A. B. (2007). The ade4 package: implementing the duality diagram for ecologists. J. Stat. Softw. 22, 1-20. doi: 10.18637/jss.v022.i04
CrossRef Full Text | Google Scholar
Dufrene, M., and Legendre, P. (1997). Artengemeinschaften und Indikatorarten: die Notwendigkeit eines flexiblen asymmetrischen Ansatzes. Ecol. Monogr. 67, 345-366. doi: 10.2307/2963459
CrossRef Full Text | Google Scholar
Edgar, R. C., Haas, B. J., Clemente, J. C., Quince, C., and Knight, R. (2011). UCHIME verbessert die Empfindlichkeit und Geschwindigkeit der Chimärenerkennung. Bioinformatics 27, 2194-2200. doi: 10.1093/bioinformatics/btr381
PubMed Abstract | CrossRef Full Text | Google Scholar
Evans, P. N., Parks, D. H., Chadwick, G. L., Robbins, S. J., Orphan, V. J., Golding, S. D., et al. (2015). Methan-Stoffwechsel im Archaeen-Phylum Bathyarchaeota offenbart durch genomzentrierte Metagenomik. Science 350, 434-438. doi: 10.1126/science.aac7745
PubMed Abstract | CrossRef Full Text | Google Scholar
Fish, J. A., Chai, B., Wang, Q., Sun, Y., Brown, C. T., Tiedje, J. M., et al. (2013). FunGene: die funktionale Gen-Pipeline und das Repository. Front. Microbiol. 4:291. doi: 10.3389/fmicb.2013.00291
PubMed Abstract | CrossRef Full Text | Google Scholar
Frank-Fahle, B. A., Yergeau, E., Greer, C. W., Lantuit, H., and Wagner, D. (2014). Microbial functional potential and community composition in permafrost-affected soils of the NW Canadian Arctic. PLoS ONE 9:e84761. doi: 10.1371/journal.pone.0084761.g001
PubMed Abstract | CrossRef Full Text | Google Scholar
Fukuzaki, S., Nishio, N., and Nagai, S. (1990). Kinetik der methanogenen Fermentation von Acetat. Appl. Environ. Microbiol. 56, 3158-3163.
Google Scholar
Hanson, C. A., Fuhrman, J. A., Horner-Devine, M. C., and Martiny, J. B. (2012). Jenseits biogeografischer Muster: Prozesse, die die mikrobielle Landschaft formen. Nat. Rev. Microbiol. 10, 497-506. doi: 10.1038/Nrmicro2795
PubMed Abstract | CrossRef Full Text | Google Scholar
Holm, G. O., Perez, B. C., McWhorter, D. E., Krauss, K. W., Johnson, D. J., Raynie, R. C., et al. (2016). Ecosystem level methane fluxes from tidal freshwater and brackish marshes of the Mississippi River Delta: implications for coastal wetland carbon projects. Wetlands 36, 401-413. doi: 10.1007/s13157-016-0746-7
CrossRef Full Text | Google Scholar
Hunger, S., Schmidt, O., Hilgarth, M., Horn, M. A., Kolb, S., Conrad, R., et al. (2011). Konkurrierende Formatum- und Kohlendioxid-verwertende Prokaryoten in einem anoxischen, Methan emittierenden Moorboden. Appl. Environ. Microbiol. 77, 3773-3785. doi: 10.1128/AEM.00282-11
PubMed Abstract | CrossRef Full Text | Google Scholar
IPCC (2007). „Climate Change 2007: the Physical Science Basis,“ in Proceedings of the Contribution of Working Group I to the Fourth Assessment Report of the Intergovernmental Panel on Climate Change, eds D. Qin, S. Solomon, M. Manning, Z. Chen, M. Marquis, K. B. Averyt, et al. (Cambridge: Cambridge University Press).
Google Scholar
Jabłoński, S., Rodowicz, P., and Łukaszewicz, M. (2015). Methanogene Archaea-Datenbank mit physiologischen und biochemischen Merkmalen. Int. J. Syst. Evol. Microbiol. 65, 1360-1368. doi: 10.1099/ijs.0.000065
PubMed Abstract | CrossRef Full Text | Google Scholar
Kirschke, S., Bousquet, P., Ciais, P., Saunois, M., Canadell, J. G., Dlugokencky, E. J., et al. (2013). Drei Jahrzehnte der globalen Methanquellen und -senken. Nat. Geosci. 6, 813-823. doi: 10.1038/Ngeo1955
CrossRef Full Text | Google Scholar
Knief, C. (2015). Diversität und Habitatpräferenzen von kultivierten und nicht kultivierten aeroben methanotrophen Bakterien anhand von pmoA als molekularem Marker bewertet. Front. Microbiol. 6:1346. doi: 10.3389/fmicb.2015.01346
PubMed Abstract | CrossRef Full Text | Google Scholar
Kotsyurbenko, O., Friedrich, M., Simankova, M., Nozhevnikova, A., Golyshin, P., Timmis, K., et al. (2007). Wechsel von acetoklastischer zu H2-abhängiger Methanogenese in einem westsibirischen Torfmoor bei niedrigen pH-Werten und Isolierung eines acidophilen Methanobakterium-Stammes. Appl. Environ. Microbiol. 73, 2344-2348. doi: 10.1128/AEM.02413-06
PubMed Abstract | CrossRef Full Text | Google Scholar
Langenheder, S., and Székely, A. J. (2011). Species sorting and neutral processes are both important during the initial assembly of bacterial communities. ISME J. 5, 1086-1094. doi: 10.1038/ismej.2010.207
PubMed Abstract | CrossRef Full Text | Google Scholar
Liebner, S., Ganzert, L., Kiss, A., Yang, S., Wagner, D., and Svenning, M. M. (2015). Shifts in methanogenic community composition and methane fluxes along the degradation of discontinuous permafrost. Front. Microbiol. 6:356. doi: 10.3389/fmicb.2015.00356
PubMed Abstract | CrossRef Full Text | Google Scholar
Liu, Y. Q., Priscu, J. C., Xiong, J. B., Conrad, R., Vick-Majors, T., Chu, H. Y., et al. (2016). Der Salzgehalt beeinflusst die Verteilungsmuster von Archaeen in hoch gelegenen Seesedimenten auf dem tibetischen Plateau. FEMS Microbiol. Ecol. 92:fiw033. doi: 10.1093/femsec/fiw033
PubMed Abstract | CrossRef Full Text | Google Scholar
Logares, R., Lindstrom, E. S., Langenheder, S., Logue, J. B., Paterson, H., Laybourn-Parry, J., et al. (2013). Biogeographie bakterieller Gemeinschaften, die fortschreitenden langfristigen Umweltveränderungen ausgesetzt sind. ISME J. 7, 937-948. doi: 10.1038/ismej.2012.168
PubMed Abstract | CrossRef Full Text | Google Scholar
Loreau, M., Naeem, S., Inchausti, P., Bengtsson, J., Grime, J. P., Hector, A., et al. (2001). Biologische Vielfalt und die Funktionsweise von Ökosystemen: Aktuelle Erkenntnisse und zukünftige Herausforderungen. Science 294, 804-808. doi: 10.1126/science.1064088
PubMed Abstract | CrossRef Full Text | Google Scholar
Lozupone, C. A., und Knight, R. (2007). Globale Muster in der bakteriellen Vielfalt. Proc. Natl. Acad. Sci. U.S.A. 104, 11436-11440. doi: 10.1073/pnas.0611525104
PubMed Abstract | CrossRef Full Text | Google Scholar
Luton, P. E., Wayne, J. M., Sharp, R. J., und Riley, P. W. (2002). Das mcrA-Gen als Alternative zur 16S rRNA in der phylogenetischen Analyse von Methanogenpopulationen in Mülldeponien. Microbiology 148, 3521-3530. doi: 10.1099/00221287-148-11-3521
PubMed Abstract | CrossRef Full Text | Google Scholar
Lutz, S., Anesio, A. M., Raiswell, R., Edwards, A., Newton, R. J., Gill, F., et al. (2016). Die Biogeographie von Mikrobiomen aus rotem Schnee und ihre Rolle beim Schmelzen arktischer Gletscher. Nat. Commun. 7:11968. doi: 10.1038/ncomms11968
PubMed Abstract | CrossRef Full Text | Google Scholar
Martiny, J. B. H., Bohannan, B. J. M., Brown, J. H., Colwell, R. K., Fuhrman, J. A., Green, J. L., et al. (2006). Mikrobielle Biogeographie: Mikroorganismen auf die Landkarte setzen. Nat. Rev. Microbiol. 4, 102-112. doi: 10.1038/Nrmicro1341
PubMed Abstract | CrossRef Full Text | Google Scholar
Martiny, J. B. H., Eisen, J. A., Penn, K., Allison, S. D., and Horner-Devine, M. C. (2011). Einflussfaktoren der bakteriellen Beta-Diversität hängen von der räumlichen Ausdehnung ab. Proc. Natl. Acad. Sci. U.S.A. 108, 7850-7854. doi: 10.1073/pnas.1016308108
PubMed Abstract | CrossRef Full Text | Google Scholar
McArdle, B. H., and Anderson, M. J. (2001). Fitting multivariate Modelle zu Gemeinschaft Daten: ein Kommentar zu Distanz-basierte Redundanz-Analyse. Ecology 82, 290-297. doi: 10.1890/0012-9658(2001)0822.0.CO;2
CrossRef Full Text | Google Scholar
McCalley, C. K., Woodcroft, B. J., Hodgkins, S. B., Wehr, R. A., Kim, E.-H., Mondav, R., et al. (2014). Die Methandynamik wird durch die Reaktion der mikrobiellen Gemeinschaft auf das Auftauen des Permafrostes reguliert. Nature 514, 478-481. doi: 10.1038/nature13798
PubMed Abstract | CrossRef Full Text | Google Scholar
McGenity, T. J. (2010). „Methanogens and Methanogenesis in Hypersaline Environments“, in Handbook of Hydrocarbon and Lipid Microbiology, ed. K. N. Timmis (Berlin: Springer), 665-680. doi: 10.1007/978-3-540-77587-4_53
CrossRef Full Text | Google Scholar
McLusky, D. S., and Elliott, M. (2004). The Estuarine Ecosystem: Ökologie, Bedrohungen und Management. New York, NY: Oxford University Press. doi: 10.1093/acprof:oso/9780198525080.001.0001
CrossRef Full Text | Google Scholar
Megonigal, J. P., Mines, M. E., and Visscher, P. T. (2005). „Anaerobic metabolism: linkages to trace gases and aerobic processes“, in Biogeochemistry, ed. W. H. Schlesinger (Oxford: Elsevier), 350-362.
Google Scholar
Milferstedt, K., Youngblut, N. D., and Whitaker, R. J. (2010). Räumliche Struktur und Persistenz von Methanogenpopulationen in humosen Moorseen. ISME J. 4, 764-776. doi: 10.1038/Ismej.2010.7
PubMed Abstract | CrossRef Full Text | Google Scholar
Mosier, A. C., and Francis, C. A. (2008). Relative Häufigkeit und Vielfalt von ammoniakoxidierenden Archaeen und Bakterien im Ästuar der San Francisco Bay. Environ. Microbiol. 10, 3002-3016. doi: 10.1111/j.1462-2920.2008.01764.x
PubMed Abstract | CrossRef Full Text | Google Scholar
Nazaries, L., Murrell, J. C., Millard, P., Baggs, L., und Singh, B. K. (2013). Methan, Mikroben und Modelle: Grundlegendes Verständnis des Methankreislaufs im Boden für zukünftige Vorhersagen. Environ. Microbiol. 15, 2395-2417. doi: 10.1111/1462-2920.12149
PubMed Abstract | CrossRef Full Text | Google Scholar
Nelson, M. B., Martiny, A. C., and Martiny, J. B. H. (2016). Global biogeography of microbial nitrogen-cycling traits in soil. Proc. Natl. Acad. Sci. U.S.A. 113, 8033-8040. doi: 10.1073/pnas.1601070113
PubMed Abstract | CrossRef Full Text | Google Scholar
Nemergut, D. R., Costello, E. K., Hamady, M., Lozupone, C., Jiang, L., Schmidt, S. K., et al. (2011). Globale Muster in der Biogeographie von Bakterientaxa. Environ. Microbiol. 13, 135-144. doi: 10.1111/j.1462-2920.2010.02315.x
PubMed Abstract | CrossRef Full Text | Google Scholar
Nobu, M. K., Narihiro, T., Kuroda, K., Mei, R., and Liu, W. T. (2016). Auf der Jagd nach der schwer fassbaren Euryarchaeota-Klasse WSA2: Genome enthüllen ein einzigartig anspruchsvolles Methyl-reduzierendes Methanogen. ISME J. 10, 2478-2487. doi: 10.1038/ismej.2016.33
PubMed Abstract | CrossRef Full Text | Google Scholar
Oksanen, J., Blanchet, F. G., Kindt, R., Legendre, P., Minchin, P. R., O’Hara, R. B., et al. (2015). vegan: Community Ecology Package. R Package Version 2.2.0. Available at: https://CRAN.R-project.org/package=vegan
Google Scholar
Oloo, F., Valverde, A., Quiroga, M. V., Vikram, S., Cowan, D., and Mataloni, G. (2016). Habitat-Heterogenität und Konnektivität prägen mikrobielle Gemeinschaften in südamerikanischen Mooren. Sci. Rep. 6:25712. doi: 10.1038/srep25712
PubMed Abstract |Ref Full Text | Google Scholar
Pattnaik, P., Mishra, S. R., Bharati, K., Mohanty, S. R., Sethunathan, N., and Adhya, T. K. (2000). Einfluss des Salzgehalts auf die Methanogenese und die zugehörige Mikroflora in tropischen Reisböden. Microbiol. Res. 155, 215-220. doi: 10.1016/S0944-5013(00)80035-X
PubMed Abstract | CrossRef Full Text | Google Scholar
Poffenbarger, H. J., Needelman, B. A., and Megonigal, J. P. (2011). Einfluss des Salzgehalts auf Methanemissionen aus Gezeitensümpfen. Wetlands 31, 831-842. doi: 10.1007/s13157-011-0197-0
PubMed Abstract | CrossRef Full Text | Google Scholar
Potter, E. G., Bebout, B. M., and Kelley, C. A. (2009). Isotopische Zusammensetzung von Methan und daraus abgeleiteten methanogenen Substraten entlang eines Salinitätsgradienten in einem hypersalinen mikrobiellen Mattensystem. Astrobiology 9, 383-390. doi: 10.1089/ast.2008.0260
PubMed Abstract | CrossRef Full Text | Google Scholar
R Core Team (2014). R: A Language and Environment for Statistical Computing (Version 3.0.3). Vienna: R Foundation for Statistical Computing.
Google Scholar
Roberts, D. (2016). labdsv: Ordination and Multivariate Analysis for Ecology. R Package Version 1.8-0. Verfügbar unter: https://CRAN.R-project.org/package=labdsv
Google Scholar
Rooney-Varga, J. N., Giewat, M. W., Duddleston, K. N., Chanton, J. P., and Hines, M. E. (2007). Zusammenhänge zwischen der Struktur der Archaeengemeinschaft, dem Vegetationstyp und dem Methanbildungsweg in Torfgebieten in Alaska. FEMS Microbiol. Ecol. 60, 240-251. doi: 10.1111/j.1574-6941.2007.00278.x
PubMed Abstract | CrossRef Full Text | Google Scholar
Rosenberg, E., DeLong, E. F., Lory, S., Stackebrandt, E., and Thompson, F. (2014). The Prokaryotes: Other Major Lineages of Bacteria and the Archaea. Berlin: Springer.
Google Scholar
Ruff, S. E., Biddle, J. F., Teske, A. P., Knittel, K., Boetius, A., and Ramette, A. (2015). Globale Ausbreitung und lokale Diversifizierung des Mikrobioms von Methanquellen. Proc. Natl. Acad. Sci. U.S.A. 112, 4015-4020. doi: 10.1073/pnas.1421865112
PubMed Abstract | CrossRef Full Text | Google Scholar
Schauer, R., Bienhold, C., Ramette, A., and Harder, J. (2009). Bakterielle Vielfalt und Biogeographie in Tiefsee-Oberflächensedimenten des Südatlantiks. ISME J. 4, 159-170. doi: 10.1038/ismej.2009.106
PubMed Abstract | CrossRef Full Text | Google Scholar
Schloss, P. D., Westcott, S. L., Ryabin, T., Hall, J. R., Hartmann, M., Hollister, E. B., et al. (2009). Einführung von mothur: quelloffene, plattformunabhängige, gemeinschaftsgestützte Software zur Beschreibung und zum Vergleich mikrobieller Gemeinschaften. Appl. Environ. Microbiol. 75, 7537-7541. doi: 10.1128/Aem.01541-09
PubMed Abstract | CrossRef Full Text | Google Scholar
Statham, P. J. (2012). Nährstoffe in Ästuaren – ein Überblick und die möglichen Auswirkungen des Klimawandels. Sci. Total Environ. 434, 213-227. doi: 10.1016/j.scitotenv.2011.09.088
PubMed Abstract | CrossRef Full Text | Google Scholar
Thauer, R. K., Kaster, A.-K., Seedorf, H., Buckel, W., and Hedderich, R. (2008). Methanogene Archaeen: Ökologisch relevante Unterschiede in der Energieerhaltung. Nat. Rev. Microbiol. 6, 579-591. doi: 10.1038/Nrmicro1931
PubMed Abstract | CrossRef Full Text | Google Scholar
Tilman, D., Isbell, F., and Cowles, J. M. (2014). Biodiversität und das Funktionieren von Ökosystemen. Annu. Rev. Ecol. Evol. Syst. 45, 471-493. doi: 10.1146/annurev-ecolsys-120213-091917
CrossRef Full Text | Google Scholar
Ulrich, W., Hajdamowicz, I., Zalewski, M., Stañska, M., Ciurzycki, W., and Tykarski, P. (2010). Artensortiment oder Habitatfilterung: eine Fallstudie über Spinnengemeinschaften auf See-Inseln. Ecol. Res. 25, 375-381. doi: 10.1007/s11284-009-0661-y
CrossRef Full Text | Google Scholar
Vanwonterghem, I., Evans, P. N., Parks, D. H., Jensen, P. D., Woodcroft, B. J., Hugenholtz, P., et al. (2016). Methylotrophe Methanogenese im Archaeenstamm Verstraetearchaeota entdeckt. Nat. Microbiol. 1:16170. doi: 10.1038/nmicrobiol.2016.170
PubMed Abstract | CrossRef Full Text | Google Scholar
von Mering, C., Hugenholtz, P., Raes, J., Tringe, S. G., Doerks, T., Jensen, L. J., et al. (2007). Quantitative phylogenetische Bewertung von mikrobiellen Gemeinschaften in verschiedenen Umgebungen. Science 315, 1126-1130. doi: 10.1126/science.1133420
PubMed Abstract | CrossRef Full Text | Google Scholar
Wagner, D., and Liebner, S. (2009). „Global warming and carbon dynamics in permafrost soils: methane production and oxidation,“ in Permafrost Soils, ed. R. Margesin (Berlin: Springer), 219-236.
Google Scholar
Weiher, E., and Keddy, P. (2001). Ecological Assembly Rules: Perspectives, Advances, Retreats. Cambridge: Cambridge University Press.
Google Scholar
Whitaker, R. J., Grogan, D. W., and Taylor, J. W. (2003). Geografische Barrieren isolieren endemische Populationen von hyperthermophilen Archaeen. Science 301, 976-978. doi: 10.1126/science.1086909
PubMed Abstract | CrossRef Full Text | Google Scholar
Wickham, H. (2009). ggplot2: Elegant Graphics for Data Analysis. New York, NY: Springer-Verlag. doi: 10.1007/978-0-387-98141-3
CrossRef Full Text | Google Scholar
Yang, J., Ma, L. A., Jiang, H., Wu, G., and Dong, H. (2016). Der Salzgehalt beeinflusst die mikrobielle Diversität und Gemeinschaftsstruktur in Oberflächensedimenten der Qinghai-Tibet-Seen. Sci. Rep. 6:25078. doi: 10.1038/srep25078
PubMed Abstract | CrossRef Full Text | Google Scholar
Yang, S., Liebner, S., Alawi, M., Ebenhöh, O., and Wagner, D. (2014). Taxonomische Datenbank und Cut-off-Wert für die Verarbeitung von mcrA-Gen 454 Pyrosequenzierungsdaten durch MOTHUR. J. Microbiol. Methods 103, 3-5. doi: 10.1016/j.mimet.2014.05.006
PubMed Abstract | CrossRef Full Text | Google Scholar
Yang, S., Liebner, S., Winkel, M., Alawi, M., Horn, F., Dörfer, C., et al. (2017). In-depth analysis of core methanogenic communities from high elevation permafrost-affected wetlands. Soil Biol. Biochem. 111, 66-77. doi: 10.1016/j.soilbio.2017.03.007
CrossRef Full Text | Google Scholar
Ye, R. Z., Jin, Q. S., Bohannan, B., Keller, J. K., McAllister, S. A., and Bridgham, S. D. (2012). pH controls over anaerobic carbon mineralization, the efficiency of methane production, and methanogenic pathways in peatlands across an ombrotrophic-minerotrophic gradient. Soil Biol. Biochem. 54, 36-47. doi: 10.1016/j.soilbio.2012.05.015
CrossRef Full Text | Google Scholar
Zinder, S. H. (1993). „Physiological ecology of methanogens,“ in Methanogenesis: Ecology, Physiology, Biochemistry & Genetics, ed. J. G. Ferry (Boston, MA: Springer), 128-206.
Google Scholar
Zinger, L., Amaral-Zettler, L. A., Fuhrman, J. A., Horner-Devine, M. C., Huse, S. M., Welch, D. B. M., et al. (2011). Globale Muster der bakteriellen Beta-Diversität in Ökosystemen am Meeresboden und im Meerwasser. PLoS ONE 6:e24570. doi: 10.1371/journal.pone.0024570
PubMed Abstract | CrossRef Full Text | Google Scholar