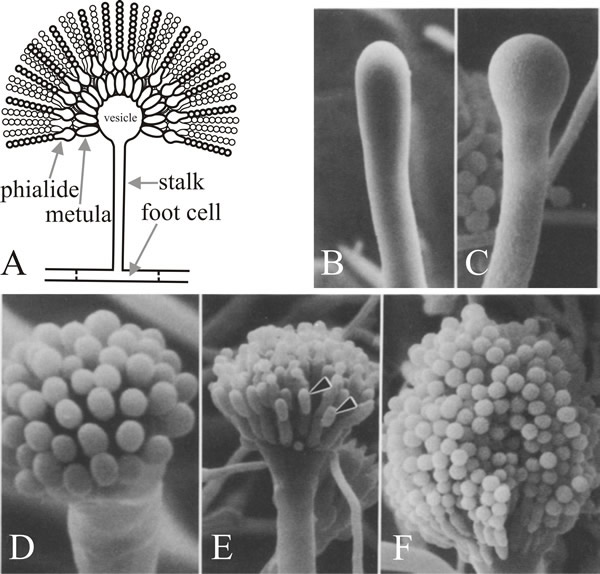
Komórką, z której wyłania się gałązka, jest komórka podstawna konidioforu, którą od innych komórek wegetatywnych odróżnia posiadanie brązowego, pigmentowanego zgrubienia ściany wtórnej po wewnętrznej stronie ściany pierwotnej. Łodyga rośnie apikalnie aż do osiągnięcia długości ok. 100 µm, kiedy to wierzchołek pęcznieje tworząc pęcherzyk konidioforu o średnicy ok. 10 µm. Następnie z pęcherzyka wyrastają pojedyncze warstwy licznych pierwotnych sterigmatów, zwanych metulami, a z odsłoniętych wierzchołków metuli wyrastają wtórne sterigmaty, czyli fialidy. Fiolidy są komórkami macierzystymi, które następnie ulegają powtarzającym się asymetrycznym podziałom, tworząc długie łańcuchy enteroblastycznych konidiów o średnicy około 3 µm (ryc. 7).
Klasyczna analiza genetyczna, poprzez izolację i analizę mutantów, ustaliła podstawowy zarys genetyczny konidiozy Aspergillus. Oszacowano, że od 300 do 1000 loci genowych jest związanych z tworzeniem konidiów poprzez porównanie częstotliwości mutacji w loci wpływających na tworzenie konidiów z tymi dla innych funkcji (Martinelli & Clutterbuck, 1971). Analiza gatunków mRNA wskazała, że około 6,000 ulegało ekspresji w wegetatywnej grzybni i dodatkowe 1,200 znajdowało się w kulturach, które zawierały konidiofory i konidia; 200 z tych dodatkowych mRNA znajdowało się w samych konidiach. Tylko około 2% mutantów A. nidulans pozbawionych konidiów miało defekty na etapach związanych ze wzrostem i rozwojem konidioforów, a 85% mutantów pozbawionych konidiów było również wadliwych w wegetatywnym wzroście hyfusów i w osiąganiu kompetencji.
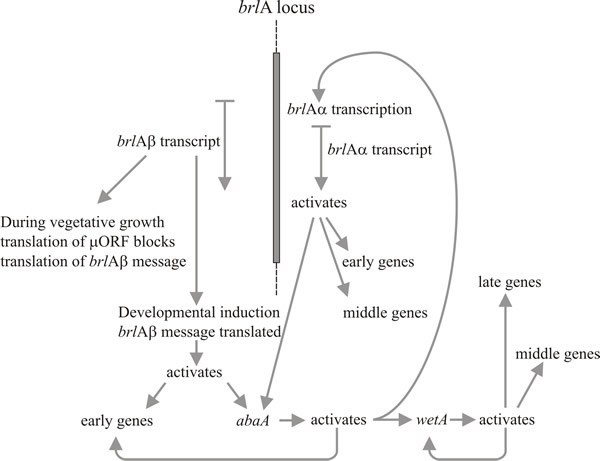
Dwa geny, w szczególności, odgrywają kluczową rolę w morfogenezie konidioforu: są to gen „szczeciny” (brlA), który ma defekty w tworzeniu pęcherzyków i metuli, oraz gen „liczydła” (abaA), w którym konidia są zastępowane przez paciorkowate odcinki hyphy, więc przypuszczalnie jest on wadliwy w pączkowaniu konidiów z fialidu i końcowej septacji. Trzeci gen, wetA, jest wadliwy we wczesnym etapie dojrzewania zarodników. Konidia mutantów wetA są pozbawione pigmentu i hydrofobowości; ulegają autolizie po kilku godzinach i nie wykazują ekspresji szeregu mRNA specyficznego dla zarodników. Transkryptu genu wetA brakuje u mutantów brlA i abaA (tzn. brlA i abaA są epistatyczne w stosunku do wetA), a badania podwójnych mutantów pokazują, że te trzy geny działają w kolejności: brlA → abaA → wetA.
Uderzającą cechą mutacyjnej analizy rozwoju konidioforów u A. nidulans jest to, że mutanty tylko tych trzech genów powodują defekty w morfogenezie konidioforów i zarodników, podczas gdy do 1000 innych loci genowych powoduje brak konidiacji, gdy są zmutowane. Sugeruje to, że brlA, abaA i wetA s± regulatorami integruj±cymi ekspresję innych genów, które s± potrzebne do konidiacji, ale same nie s± do niej dedykowane. Wiele mutantów Aspergillus wykazuje również defekt w rozmnażaniu płciowym, więc kolejny wniosek, jaki można wyciągnąć, jest taki, że istnieje pewna ekonomia wykorzystania genów morfogenetycznych w różnych procesach rozwojowych. Przypuszczalnie różne tryby rozwojowe wykorzystują geny strukturalne, które nie są wyłącznie rozwojowe, ale funkcjonują w wielu ścieżkach, a ich specyficzność rozwojowa jest nadawana im przez regulatory, na które reagują. Uosabia to idea, że kluczem do rozwoju eukariotów jest zdolność wykorzystania stosunkowo niewielu genów regulatorowych do zintegrowania aktywności wielu innych.
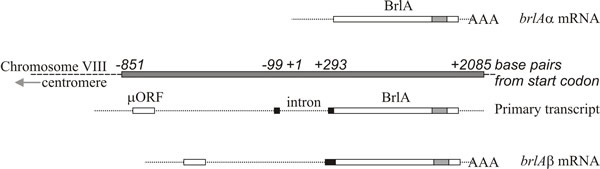
Analiza molekularna wspiera interpretację, że brlA, abaA i wetA są regulatorami; sekwencja brlA koduje białko palca cynkowego, które jest specyficznym dla sekwencji DNA aktywatorem transkrypcji wiążącym geny docelowe regulowane rozwojowo (ryc. 8). Nie jest to jednak cała historia, ponieważ produkt brlA ma różne powinowactwa do różnych genów docelowych. W rzeczywistości locus brlA składa się z nakładających się jednostek transkrypcyjnych (Ryc. 9), z których jednostka downstream jest oznaczona jako brlAα, a jednostka upstream brlAβ; pomiędzy nimi, ich produkty rozwiązują dwa klasyczne problemy rozwojowe, jak odpowiedzieć na sygnał i jak utrzymać tę odpowiedź, gdy sygnał się rozproszył.
Dwie jednostki transkrypcyjne brlA dzielą tę samą ramkę odczytu przez większą część swojej długości, ale brlAβ ma dodatkowe 23 reszty aminokwasowe na aminokońcowym końcu tej ramki odczytu, a jego transkrypt posiada również oddzielną ramkę odczytu 41 reszt aminokwasowych (zwaną µORF) w pobliżu swojego 5′ terminusa. Produkt µORF represjonuje translację z ramki odczytu BrlA i chociaż transkrypt brlAβ może być wykryty w wegetatywnych hyfusach, peptyd BrlA nie jest produkowany. Represja wywołana przez µORF jest przezwyciężana tylko wtedy, gdy grzybnia jest kompetentna, co jest prawdopodobnie sygnalizowane ograniczeniem azotu (powszechny sygnał środowiskowy dla inicjacji sporulacji u Ascomycota), które zmniejsza pule aminoacylo-tRNA i zaburza regulację translacji przez µORF. Gdy działanie µORF zostaje zahamowane, peptyd BrlA może zostać przetłumaczony z istniejącego transkryptu.
Odkrywca tego procesu określił aktywację szlaku konidiacji jako „wyzwalanie translacji” (Timberlake, 1993), sugerując, że wyzwalanie translacji jest sposobem na uwrażliwienie różnicowania na status odżywczy hyfy. Tak więc kompetentna hiefa jest przygotowana do podjęcia rozwoju konidioforu, ale nieodwracalna aktywacja szlaku konidialnego jest uniemożliwiona przez represję translacyjną, która utrzymuje wzrost wegetatywny do czasu, gdy warunki sporulacji będą idealne.
Aktywacja brlA jest zatem postrzegana jako pierwszy krok w rozwoju konidioforu, a jej produkt z kolei aktywuje panel genów specyficznych dla konidioforu, wśród których znajduje się kolejny regulator, abaA. Produkt abaA jest również wi±ż±cym się z DNA białkiem regulatora transkrypcji, które zwiększa ekspresję genów strukturalnych indukowanych przez brlA. Geny brlA i abaA s± wzajemnymi aktywatorami, ponieważ abaA aktywuje również brlA. Oczywiście ekspresja brlA musi nastąpić przed ekspresją abaA, ale konsekwentna aktywacja brlA przez abaA wzmacnia ekspresję tego ostatniego i skutecznie uniezależnia postęp szlaku od zdarzeń zewnętrznych. Produkt abaA aktywuje także dodatkowe geny strukturalne oraz ostatni gen regulatorowy, wetA, który aktywuje geny strukturalne specyficzne dla zarodników. Ponieważ brlA i abaA nie ulegaj± ekspresji w różnicuj±cych się konidiach, wetA prawdopodobnie bierze udział w inaktywacji ich ekspresji w zarodnikach (i być może także w fialidach; gdy j±dro bież±cego zarodnika osi±ga etap dojrzewania, który wymaga genów regulowanych przez wetA, j±dra fialidów musz± być „zawrócone” do stanu inicjacji konidium, aby rozpocz±ć formowanie kolejnego zarodnika). Ekspresja wetA jest początkowo aktywowana w fialidach przez sekwencyjne działanie brlA i abaA, a następnie ulega autoregulacji (produkt wetA aktywuje transkrypcję wetA). Pozytywna autoregulacja wetA podtrzymuje następnie jego ekspresję po oddzieleniu konidium (fizycznie i cytologicznie) od fialidu.
Timberlake (1993) nazwał ten mechanizm utrwalaniem zwrotnym: wzajemna aktywacja, aktywacja zwrotna i autoregulacja podstawowych sekwencji regulacyjnych wzmacnia ekspresję całego szlaku, uniezależniając ją od zewnętrznych czynników środowiskowych, które ją zainicjowały i umożliwiając zarodnikowi kontynuowanie dojrzewania nawet po oddzieleniu od fialidu. Rozwój konidioforu, jak wiele innych procesów morfogenetycznych, jest naturalnie podzielony na sekwencyjne etapy. Ta sieć regulacyjna pokazuje, jak translacyjne wyzwalanie może powiązać ścieżkę morfogenetyczną z jednej strony z rozwojem kompetencji, a z drugiej z inicjacją w odpowiedzi na wskazówki środowiskowe. Po inicjacji, utrwalenie sprzężenia zwrotnego skutkuje determinacją rozwojową w klasycznym embriologicznym sensie kontynuowania morfogenezy nawet po usunięciu ze środowiska inicjującego.
W tej sekcji celowo wybraliśmy oparcie naszego opisu na badaniach przeprowadzonych około 50 lat temu, które rozpoczęły się od wyizolowania dużej liczby mutacji ze zmienionymi fenotypami konidacji i kontynuowały „klasyczne” badania segregacji genów i komplementacji genów (Martinelli & Clutterbuck, 1971). Poza pokazaniem, w jaki sposób nasze zrozumienie sporulacji zostało osiągnięte, pokazuje to wartość używania tych „staroświeckich” technik do badania takich zjawisk.
Nie możemy jednak ignorować rozległych badań, które zostały zakończone nad molekularnymi mechanizmami leżącymi u podstaw wzrostu i rozwoju Aspergillus. Badania te potwierdziły, że kluczowym wydarzeniem w sporulacji jest aktywacja czynnika transkrypcyjnego z palcem cynkowym kodowanego przez brlA, oraz że geny abaA i wetA są niezbędnymi regulatorami konidiacji. Czynnik transkrypcyjny kodowany przez abaA jest aktywowany przez brlA po zróżnicowaniu metuli i podczas środkowych etapów rozwoju konidioforu; natomiast gen wetA, aktywowany przez abaA, funkcjonuje w późnej fazie konidiacji, kierując syntezą kluczowych składników ściany komórkowej i przekształcając metabolizm dojrzewającego konidium. U Aspergillus nidulans te trzy geny tworzą centralny szlak regulacyjny, który wraz z innymi genami kontroluje ekspresję genów specyficznych dla konidiów oraz sekwencję aktywacji genów zaangażowanych w nabywanie kompetencji rozwojowej, rozwój konidioforów i dojrzewanie zarodników (Lee i in., 2016).
Wiele aspektów tych szlaków rozwojowych zostało zachowanych u innych aspergilli. Członkowie rodzaju Aspergillus należą do najczęściej spotykanych grzybów, a wszystkie rozmnażają się bezpłciowo poprzez tworzenie długich łańcuchów konidiów. Kilka gatunków, w tym Aspergillus oryzae i Aspergillus niger, jest wykorzystywanych w przemyśle do produkcji enzymów i przetwarzania żywności; natomiast Aspergillus flavus jest odpowiedzialny za psucie się żywności poprzez wytwarzanie najsilniejszych znanych naturalnie występujących substancji rakotwórczych, aflatoksyn. Innym niepokojącym gatunkiem jest oportunistyczny patogen człowieka Aspergillus fumigatus, który wytwarza ogromną liczbę małych hydrofobowych konidiów, będących jego głównym środkiem rozprzestrzeniania się i stał się szeroko rozpowszechnionym patogenem grzybiczym przenoszonym drogą powietrzną w krajach rozwiniętych. U pacjentów z obniżoną odpornością A. fumigatus wywołuje inwazyjną aspergilozę, która charakteryzuje się wysoką śmiertelnością. Szlak sygnalizacji rozwojowej BrlA-AbaA-WetA został zachowany w konidiacji tych gatunków Aspergillus (Yu, 2010; Tao & Yu, 2011; Krijgsheld i in., 2013). Dalsze badania proteomiczne, transkryptomiczne i metabolomiczne dostarczyły szczegółowego obrazu dynamicznych zmian, jakie zachodzą w wielu tysiącach genów, transkryptów, enzymów i reakcji metabolicznych podczas formowania, dojrzewania, spoczynku i kiełkowania konidiów Aspergillus (van Leeuwen i in., 2013; Novodvorska i in., 2016; Teertstra i in., 2017).
Uaktualnione lipiec, 2019
.