Introduction
Metan (CH4) jest głównym gazem cieplarnianym. Jego emisja ze środowisk naturalnych, takich jak mokradła, oceany i osady, stanowi ponad 70% metanu atmosferycznego w skali globalnej (IPCC, 2007). Ocena opublikowanych danych wykazała różne wskaźniki emisji metanu dla mokradeł, jezior, rzek, estuariów i oceanów (w kolejności malejącej, Supplementary Figure S1). Same naturalne tereny podmokłe odpowiadają za 62% produkcji biogenicznego CH4 (Kirschke i in., 2013; Nazaries i in., 2013), a emisje z terenów podmokłych dominują w międzyrocznej zmienności źródeł metanu (Bousquet i in., 2006). Z kolei rozległy obszar ekosystemów morskich wnosi jedynie około 8% do naturalnych źródeł CH4 (Nazaries i in., 2013). W ramach globalnej oceanicznej emisji metanu, mniej niż 10% przypada na estuaria (Bange i in., 1994).
Metanogeneza, czyli biologiczne tworzenie metanu, jest przeprowadzana przez archaea metanogenne, które w warunkach anoksycznych wytwarzają metan głównie z H2/CO2, grup metylowych lub octanu (Thauer i in., 2008). Reakcja ta jest katalizowana przez reduktazę metylo-koenzymu M (MCR). Gen mcrA kodujący podjednostkę tego enzymu jest powszechnie stosowanym markerem genowym w badaniach molekularnych (Conrad, 2007; Bridgham i in., 2013). Zaletą markera genowego mcrA jest uchwycenie zarówno sygnatur filogenetycznych, jak i funkcjonalnych metanogenów, oferując dużą głębokość sekwencjonowania dla tej konkretnej funkcji (Luton i in., 2002; Borrel i in., 2013; Yang i in., 2014). Duża liczba sekwencji mcrA została pobrana z różnych środowisk naturalnych. Publiczny zbiór danych mcrA pozwala na wyodrębnienie ogólnych wzorców ekologicznych i zbadanie kształtujących się gradientów środowiskowych w skalach globalnych i regionalnych. Dodatkowo, dostępna jest baza danych podsumowująca właściwości fizjologiczne 152 izolatów metanogennych1 (Jabłoński i in., 2015). Ostatnio binning genomów ujawnił niezwykłe sekwencje mcrA w nowej klasie Methanofastidiosa (Nobu i in., 2016) oraz w nowych filiach Bathyarchaeota (Evans i in., 2015) i Verstraetearchaeota (Vanwonterghem i in., 2016). Te nowe odkrycia poszerzyły naszą wiedzę na temat różnorodności potencjalnych metanogenów, ale nie przesłoniły możliwości zastosowania genu mcrA jako markera molekularnego dla znacznej większości zbiorowisk metanogennych.
Do tej pory zbiorowiska metanogenne wykryto na terenach podmokłych, w osadach, na obszarach wiecznej zmarzliny, na polach ryżowych, w komorach fermentacyjnych, w źródłach geotermalnych i w kominkach hydrotermalnych (Conrad, 2007; Thauer i in., 2008; Wagner i Liebner, 2009). Stwierdzono, że struktura zbiorowiska metanogenów jest związana z pH środowiska, temperaturą, zasoleniem, poziomem wód gruntowych i dynamiką roślinności w różnych skalach przestrzennych i czasowych (Megonigal i in., 2005; Milferstedt i in., 2010; Frank-Fahle i in., 2014; McCalley i in., 2014; Cui i in., 2015; Liebner i in., 2015). Na przykład, metanogenezę acetoklastyczną hamuje na ogół niskie pH, ponieważ zmniejsza ono dysocjację octanową (Megonigal i in., 2005; Kotsyurbenko i in., 2007). Roślinność może dostarczać labilnego, wysokiej jakości węgla organicznego do zasilania metanogenów w postaci wysięków korzeniowych lub detrytusu, tak więc wysięki roślinne generalnie sprzyjają metanogenom acetoklastycznym głównie na torfowiskach (Bridgham i in., 2013). Siarczany pochodzące z wody morskiej hamują produkcję metanu na mokradłach pływowych, a zasolenie było w konsekwencji wykorzystywane jako ogólny predyktor emisji metanu (Holm i in., 2016). Badania osadów z jezior tybetańskich wykazały, że rosnące zasolenie hamuje metanogeny hydrogenotroficzne, ale zwiększa metanogenezę acetoklastyczną (Liu i in., 2016). Badania te wskazały na czynniki środowiskowe dla zbiorowisk metanogennych, ale koncentrowały się na pojedynczych siedliskach lub ograniczonych skalach przestrzennych.
Zrozumienie adaptacji metanogenów do różnych zmian środowiskowych wymaga jednak systematycznego i globalnego zbadania korelacji między składem zbiorowisk mikrobiologicznych a warunkami środowiskowymi (Lozupone i Knight, 2007). Obecnie, tylko kilka badań dotyczy dyspersji i filtracji siedliskowej zbiorowisk metanogennych (Auguet i in., 2010; Barreto i in., 2014). Postawiliśmy hipotezę, że na zespoły metanogeniczne wpływa głównie filtrowanie siedlisk i że jest ono sterowane przez globalne czynniki środowiskowe. Biorąc pod uwagę, że wskaźniki emisji metanu różnią się znacznie w poszczególnych naturalnych ekosystemach, wyraźne uwzględnienie składu, różnorodności i biogeografii zespołów metanogenicznych w tych ekosystemach może mieć zasadnicze znaczenie dla określenia odpowiedzi produkcji metanu na obecne i przyszłe zmiany klimatu. Niniejsze meta-badanie przeprowadzono w celu wypełnienia luk związanych z biogeografią metanogenów, ich różnorodnością i kontrolą środowiskową, wykorzystując publicznie dostępne dane sekwencji mcrA i literaturę uzupełnioną danymi fizjologicznymi izolatów metanogenów.
Materiały i Metody
Gromadzenie danych
Wybraliśmy sekwencje mcrA dostępne w GenBank (styczeń 2016)2. Dla każdego trafienia sprawdzono oryginalny papier, a odpowiadające usystematyzowane sekwencje mcrA zostały sparsowane przez niestandardowy skrypt Perla. Ponieważ skupiliśmy się na środowiskach naturalnych, metanogenne sekwencje mcrA uzyskano ze środowisk naturalnych i sklasyfikowano je jako glebę, osady jeziorne, estuaryjne, morskie i hydrotermalne oraz wulkany błotne. Oprócz sekwencji z bibliotek klonów uwzględniono pięć bibliotek pochodzących z sekwencjonowania następnej generacji (NGS). Sekwencje zostały pobrane bez uwzględnienia względnej liczebności w oryginalnym zbiorze danych. Ponieważ sekwencje z bibliotek klonów obejmują głównie obfite filotypy, podczas gdy NGS może uchwycić znacznie głębszą różnorodność, poszliśmy na kompromis, aby wykorzystać dane NGS, ale złagodzić potencjalny błąd wynikający z różnej rozdzielczości metod sekwencjonowania. Dlatego wybraliśmy tylko reprezentatywne sekwencje obfitych OTU, których względna liczebność była wyższa niż 1%. Odrzuciliśmy również te sekwencje NGS, które nie przeszły testu translacji z sekwencji nukleotydowych na białkowe lub charakteryzowały się niską jakością (sekwencje < 250 bps). Ostatecznie skonstruowaliśmy zbiór danych zawierający 4466 unikalnych sekwencji mcrA z 94 globalnie rozmieszczonych miejsc (Rysunek 1 i Tabele Uzupełniające S1, S2). Dodatkowo, nie odjęliśmy od zbioru danych sekwencji mcrA potencjalnych archetypowych metanotrofów, które były nieuchronnie wykrywane w badaniach genomicznych (Conrad, 2007). Ta część wykracza poza zakres niniejszego opracowania.
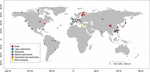
FIGURE 1. Lokalizacja stanowisk wykorzystanych w niniejszym opracowaniu. Stanowiska pogrupowano na sześć różnych kategorii w zależności od siedlisk i oznaczono różnymi kolorami. Podsumowanie sześciu siedlisk podano w tabelach uzupełniających S1, S2.
Współrzędne geograficzne i ustawienia środowiskowe, w tym pH, zasolenie, wysokość, średnia roczna temperatura powietrza (MAAT) i średnie roczne opady (MAP) zostały pobrane dla każdego miejsca badawczego uwzględnionego w tym badaniu z odpowiednich publikacji, o ile dane te są dostępne (patrz tabela uzupełniająca S1). Aby uwzględnić brakujące parametry środowiskowe w wielu badaniach, zdefiniowaliśmy jakościowo niektóre zmienne środowiskowe zgodnie z opisami miejsc w odpowiedniej literaturze, a następnie przekształciliśmy te dane kategorii w semiometryczne wartości liczbowe, na przykład zdefiniowaliśmy osady morskie, osady hydrotermalne, błoto wulkaniczne i próbki osadów jeziora sodowego jako „słone”, glebę i próbki osadów jeziora słodkowodnego jako „niesolne”, a próbki namorzynowe i estuarium jako próbki „mieszane”.
Raw Sequence Processing
Przetwarzanie sekwencji zostało zaimplementowane za pomocą platformy programowej Mothur (Schloss i in., 2009). Sekwencje z różnych bibliotek były łączone przed przetwarzaniem. Sekwencje o długości mniejszej niż 350 bp lub zawierające więcej niż 8 niejednoznacznych zasad były odrzucane. Następnie, sekwencje te zostały wyrównane do wstępnie wyrównanego podzbioru sekwencji mcrA, które zostały pobrane z bazy FunGene pod adresem http://fungene.cme.msu.edu/ (Fish i in., 2013). Sekwencje chimeryczne identyfikowano za pomocą programu Mothur metodą uchime (Edgar i in., 2011) z wykorzystaniem samego zbioru danych jako referencji. Następnie, prawidłowe sekwencje nukleotydowe genu mcrA zostały wykorzystane do obliczenia nieskorygowanych odległości parami pomiędzy wyrównanymi sekwencjami DNA i dalej przypisane do operacyjnych jednostek taksonomicznych (OTU) przy odcięciu 84%, co odpowiada 97% dla genu 16S rRNA (Yang i in., 2014). Obfitość każdego OTU mcrA była ujmowana tylko jako obecność lub brak. Zwiększyliśmy dokładność klasyfikacji taksonomicznej OTU, biorąc pod uwagę zarówno sekwencje nukleotydowe, jak i aminokwasowe. Na poziomie DNA, tożsamość taksonomiczna została przypisana przez platformę Mothur zgodnie z referencyjną bazą danych (Yang i in., 2014). Na poziomie białka, wyrównane sekwencje białkowe zostały użyte do skonstruowania drzewa w ARB, a następnie przypisanie taksonomiczne zostało oparte na odpowiedniej bazie danych. Jeśli przypisanie OTU było niespójne, ręcznie blastowaliśmy zarówno sekwencję nukleotydową, jak i białkową w NCBI i określaliśmy ostateczną tożsamość taksonomiczną, biorąc pod uwagę pokrycie zapytania (>95%), tożsamość (>84%) i wartość e- (<1E-5). Dla sekwencji białkowych, odcięcie na poziomie rodzaju odnosiło się do progu 83,5% (Hunger i in., 2011).
Analiza ekologiczna i statystyczna
Analizę statystyczną przeprowadzono za pomocą różnych pakietów R. Rzędne analizy współrzędnych głównych (PCoA) zostały wygenerowane na podstawie macierzy odległości Jaccarda skonstruowanych przy użyciu pakietu vegan v2.2.0 (Oksanen i in., 2015). Permutacyjną MANOVA (multivariate analysis of variance) przeprowadzono w celu oceny źródła zmienności macierzy Jaccarda (McArdle i Anderson, 2001) w weganach przy 104 permutacjach. Miary odległości Jaccarda oparte są na obecności/nieobecności gatunków, co jest bardziej odpowiednie dla naszego zbioru danych, ponieważ większość badań dostarczyła jedynie reprezentatywne sekwencje, podczas gdy brakuje informacji o liczebności. Częstotliwości występowania taksonomicznego w siedliskach były wizualizowane za pomocą wykresów bąbelkowych przy użyciu pakietu ggplot2 (v1.0.0) (Wickham, 2009). Hierarchiczną analizę skupień zbiorowisk gleb niesolankowych i osadów jeziornych przeprowadzono za pomocą funkcji R 'hclust’ (R Core Team, 2014). Uzyskane skupienia zbiorowisk opisano zgodnie z reżimem pH i temperatury oryginalnych próbek, ponieważ stosując wcześniej PCoA zidentyfikowaliśmy oba parametry pH i temperaturę jako wpływające na skład zbiorowisk metanogennych w niesolankowych glebach i osadach jeziornych. Powiązanie każdej linii metanogenicznej z każdym z tych skupisk określono za pomocą analizy gatunków wskaźnikowych opartej na korelacji (Dufrene i Legendre, 1997). Gatunki wskaźnikowe definiuje się tutaj jako te, które występują zarówno licznie w określonym typie siedliska (specyficzność), jak i przeważają w tym typie siedliska (wierność). W niniejszej pracy taksony wskaźnikowe, podobnie jak w przypadku koncepcji gatunków wskaźnikowych, dla gleb niesolankowych i osadów jeziornych były wybierane według wartości wskaźnikowej (wartość IndVal) przez pakiet R labdsv (Roberts, 2016), jeśli prawdopodobieństwo uzyskania wartości wskaźnikowej tak wysokiej jak obserwowana w ciągu określonych iteracji jest mniejsze niż 0,05. Indeksy Chao2 zostały obliczone dla każdej próby za pomocą pakietu vegan. Test sumy rang Wilcoxona indeksów Chao2 pomiędzy siedliskami wykonano za pomocą funkcji R 'wilcox.test’ (R Core Team, 2014). W celu utrwalenia wpływu filtracji siedliskowej na izolaty archaea metanogennych, z bazy danych3 'Methanogenic archaea database’ (Jabłoński i in., 2015) pobrano charakterystykę fizjologiczną i biochemiczną opisanych kultur metanogennych. Spośród nich izolaty z informacją o optymalnym zapotrzebowaniu na NaCl zostały przefiltrowane, skategoryzowane i wykreślone zgodnie ze źródłem izolacji.
W celu zbadania wpływu ograniczenia dyspersji na strukturę zbiorowiska metanogenów wykonano analizę regresji liniowej w oparciu o macierz odległości geograficznej i macierz odległości Jaccarda zbiorowiska za pomocą funkcji R 'lm’ (R Core Team, 2014). Przeprowadziliśmy testy Mantela i częściowe testy Mantela w celu oceny efektów ograniczenia dyspersji według tych dwóch macierzy ponownie przy użyciu pakietu vegan w R (Oksanen i in., 2015). Następnie zastosowano wielowariantową analizę przestrzenną (spatial PCA) dla 16 europejskich próbek gleby i osadów jeziornych w oparciu o indeks Morana I w celu zbadania struktury przestrzennej metanogenów za pomocą funkcji „multispati” w pakiecie R ade4 (Dray i Dufour, 2007). Ponadto, na tych 16 próbkach zaimplementowano grupowanie według minimalnej wariancji Warda, oparte na macierzy odległości Jaccarda, przy użyciu funkcji R „hclust” (R Core Team, 2014), a następnie wyniki grupowania wyświetlono na mapie geograficznej. Europejski shapefile do mapowania na poziomie państw jest dostępny w GSHHG Database (v2.3.6)4. Mapa została wygenerowana przy użyciu QGIS v2.18.25.
Results
Biogeography of Methanogenic Archaea in Natural Environments
Sekwencje genu mcrA zostały pobrane z 94 globalnie rozmieszczonych środowisk naturalnych. Lokalizacja i typ ekosystemu każdego z tych 94 miejsc jest przedstawiony na Rysunku 1. Częstotliwości występowania (obecność/nieobecność) linii metanogennych zostały połączone zgodnie z typem ekosystemu i zilustrowane na Rysunku 2. W skrócie, Methanoregula jest najczęściej obserwowanym taksonem w glebie, wraz z Methanobacterium, Methanosaeta, Methanocella, Methanomassiliicoccus i Methanosarcina. W osadach estuarium powszechnie wykrywano sekwencje z Methanosaeta, Methanobacterium, Methanoregula i Methanoculleus. Ponadto w osadach jeziornych występowały głównie Methanoregula i Methanosaeta. W osadach morskich najczęściej występowały Methanoculleus i Methanosaeta, a następnie Methanolinea.
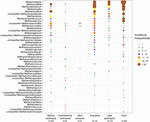
FIGURE 2. Wykres bąbelkowy przedstawiający częstości występowania linii metanogennych w różnych środowiskach naturalnych. Porządek rangowy wzdłuż osi pionowej odpowiada malejącym całkowitym częstościom występowania poszczególnych linii. Taksonomia jest przedstawiona dla poziomu rodzaju. Jeśli przypisanie do poziomu rodzaju nie było możliwe, użyto następnego wyższego poziomu taksonomicznego, który można przypisać. Liczba próbek (n) jest podana dla każdego siedliska.
Mimo, że wiele taksonów zostało wykrytych w różnych środowiskach, niektóre z nich nadal wykazują preferencje środowiskowe. Methanoregula, takson często występujący w środowiskach niemorskich i przejściowych (gleby, osady jeziorne i estuaria), jest nieobecny w siedliskach morskich (osady morskie, osady hydrotermalne i wulkany błotne). Methanobacterium i Methanocella, które dominują w środowiskach niemorskich i przejściowych, są rzadko spotykane w siedliskach morskich. W przeciwieństwie do nich, Methanococcoides, jako dominująca linia w osadach morskich, osadach hydrotermalnych i wulkanach błotnych, jest ledwie obserwowana w glebie i osadach jeziornych. Co więcej, Methanogenium i Methanolacinia obserwowane są tylko w osadach estuariowych i morskich, podczas gdy Methanospirillum i Methanosphaerula występują tylko w środowiskach lądowych. Ponadto niektóre specyficzne taksony występują wyłącznie w osadach hydrotermalnych, w tym Methanocaldococcus, Methanothermococcus, Methanopyrus, Methanotorris i Methanococcus. Chociaż niektóre linie, takie jak Methanosaeta, są obecne w większości środowisk, żadna linia nie może być uznana za wszechobecną.
Największe bogactwo linii występowało w osadach estuarium, które również charakteryzują się bardziej wyrównaną częstotliwością występowania różnych linii. Z kolei ekosystemy wulkanów błotnych i hydrotermalne wykazują stosunkowo niską różnorodność metanogeniczną. Gleby i osady jeziorne podobne do estuariów charakteryzują się zróżnicowanymi zespołami metanogennymi.
Alpha-Diversity of Methanogenic Communities in Natural Environments
Różnorodność archai metanogennych według wskaźnika Chao2 różniła się znacznie pomiędzy typami ekosystemów (Rysunek 3). W celu bezpośredniego porównania zróżnicowania alfa i uzyskania rozsądnego kompromisu między próbkami, dokonano podpróbkowania do 15 sekwencji dla każdego stanowiska. Indeks Chao2 pokazuje, że osady estuarium obejmują najwyższe bogactwo gatunkowe metanogenów w obrębie sześciu typów ekosystemów (Tabela uzupełniająca S3), co podkreśla wyniki wykresu bąbelkowego (Rysunek 2). Gleby i osady jeziorne, wykazujące niższe bogactwo gatunkowe niż próbki z estuarium, mają znacznie wyższe wskaźniki Chao2 niż osady morskie i osady hydrotermalne oraz nie wykazują istotnych różnic między osadami morskimi i osadami hydrotermalnymi (Tabela uzupełniająca S3).
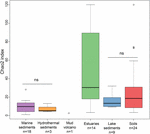
RYSUNEK 3. Wykres pudełkowy wskaźników Chao2 dla różnych typów ekosystemów. Wykres oparty jest na podpróbkowanych zestawach danych zawierających 15 sekwencji dla każdego siedliska, aby porównanie miar różnorodności alfa było bardziej wiarygodne. Liczba próbek w każdym siedlisku jest podana jako „n” pod etykietą siedliska. Litera „ns” oznacza brak istotności statystycznej w teście Wilcoxona. Statystyczny wynik różnorodności alfa na poziomie OTU podano w Tabeli S3.
Global Controls on Methanogenic Communities in Natural Environments
Koncentracja 94 globalnie rozmieszczonych zbiorowisk metanogennych została dokonana poprzez zastosowanie PCoA w oparciu o macierz odległości Jaccarda. Zgodnie z analizą PCoA, pierwsza i druga oś razem wyjaśniają 16,3% całkowitej wariancji. Różnice między próbkami można zatem w dużej mierze wyjaśnić zasoleniem (Rysunek 4). Ponieważ wstępne dane dotyczące stężenia soli były w niektórych przypadkach niedostępne, przyporządkowaliśmy te próbki jakościowo jako próbki zasolone, mieszane (pośrednie) i niezasolone, jak opisano powyżej. Próbki zasolone i niezasolone skutecznie rozdzielają się wzdłuż pierwszej osi. Próbki mieszane ogólnie grupują się pomiędzy próbkami solnymi i niesolnymi. Permutacyjna MANOVA oparta na macierzy odległości Jaccarda również sugeruje, że zasolenie jest głównym czynnikiem abiotycznym kontrolującym rozmieszczenie globalnych zbiorowisk metanogenicznych (R2 = 0,099, P < 0,001) (Tabela 1).
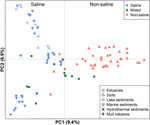
FIGURE 4. Rzędowość analizy współrzędnych głównych (PCoA) na podstawie macierzy odległości Jaccarda bibliotek sekwencji metanogennych genów mcrA porównujących 94 próbki. PCoA jest kolorowana według zasolenia: Symbole czerwone oznaczają środowiska niesolne, niebieskie – środowiska zasolone, a zielone – środowiska pośrednie. Różne symbole przedstawiają różne środowiska. Na osiach zaznaczono procent zmienności wyjaśnionej przez wykreślone współrzędne główne.

TABELA 1. Permutacyjna analiza MANOVA na macierzy odległości Jaccarda wszystkich próbek z sześciu siedlisk w celu sprawdzenia związku wariancji zbiorowości z różnymi zmiennymi środowiskowymi.
Dodatkowo sprawdziliśmy potencjalną zależność między źródłem izolacji czystych kultur metanogennych a optymalnym stężeniem NaCl dla wzrostu. Optymalne stężenie NaCl czystych kultur metanogennych wykazywało tendencję spadkową od ekosystemów morskich do estuariów, osadów jeziornych i gleby (Supplementary Figure S2). Kilka odstających izolatów pochodzi z osadów jezior sodowych lub gleb hipersalinarnych.
Kontrole środowiskowe i taksony wskaźnikowe metanogenów w glebach niesolankowych i osadach jeziornych
W skali globalnej, zbiorowiska metanogenów z gleb niesolankowych i osadów jeziornych ściśle się grupują (Rysunek 4), tak więc przeprowadziliśmy dalszą analizę kontroli środowiskowych zbiorowisk metanogenów z tych dwóch siedlisk, które łącznie obejmują 33 stanowiska badawcze. Analiza skupień dla tych dwóch typów siedlisk ujawniła cztery skupienia w oparciu o macierz odległości Jaccarda (Supplementary Figure S3). Permutacyjna MANOVA sugeruje, że zarówno pH (R2 = 0,099, P < 0,001) jak i temperatura (R2 = 0,069, P < 0,001) wpływają na metanogenną β-różnorodność w niesolankowych glebach i osadach jeziornych (Tabela 2). W związku z tym, przypisaliśmy cztery klastry do pH i MAAT początkowego miejsca pobierania próbek i uzyskaliśmy w dużej mierze spójne podgrupy do grupowania społeczności (Rysunki 5C,D). Połączenie cech środowiskowych i tych czterech skupisk zbiorowisk pozwala nam zdefiniować te cztery podgrupy jako neutralną i zimną grupę 1, kwaśną i zimną grupę 2, kwaśną i umiarkowaną grupę 3 oraz neutralną i ciepłą grupę 4 (Rysunek 5). Dalsze porządkowanie PCoA oparte na macierzy niepodobieństwa Jaccarda sugeruje, że wzdłuż osi PC1, większość próbek z grupy 2 i 3 pochodzi z kwaśnych gleb i osadów jeziornych, podczas gdy grupa 1 i grupa 4 pochodziły głównie ze środowisk neutralnych (Rysunek 5A). Ponadto próbki ze stanowisk umiarkowanych (grupa 3) oddzielały się od próbek ze stanowisk ciepłych i zimnych wzdłuż osi PC2, podczas gdy próbki ze środowisk ciepłych (grupa 4) oddzielały się od pozostałych próbek wzdłuż osi PC3 (Rysunek 5B). Tym samym pierwsze trzy osie rzędowości PCoA wyjaśniają 38,8% całkowitej zmienności.

TABELA 2. Permutacyjna MANOVA oparta na macierzy odległości Jaccarda próbek gleby niesolnej i osadów jeziornych w celu zbadania związku wariancji zbiorowiska z różnymi zmiennymi środowiskowymi.
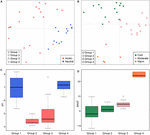
RYSUNEK 5. Wykres analizy współrzędnych głównych na podstawie macierzy odległości Jaccarda dla 33 gleb niesolankowych i osadów jeziornych. Podplot (A) przedstawia składowe PC1 i PC2, a symbole są oznaczone kolorem pH, natomiast podplot (B) przedstawia składowe PC2 i PC3, a symbole są oznaczone kolorem temperatury. Pierwsze trzy składowe wyjaśniają 17,1, 11,7 i 10% wariancji. Na wykresach pudełkowych przedstawiono pH (C) i MAAT (średnia roczna temperatura powietrza) (D) dla czterech zidentyfikowanych podgrup. Kolor pudełka na rysunku C odpowiada kategorii pH w (A). Kolory w boxplotach wskazują na istotność statystyczną w oparciu o test Wilcoxona (P < 0.05), gdzie próbki o tym samym kolorze nie różnią się istotnie od siebie. Podobnie, kolor w (D) jest zgodny z podziałem na grupy temperaturowe w (B). Podgrupy odnoszą się do hierarchicznej analizy skupień podobieństw społeczności.
Zbadaliśmy występowanie linii metanogennych w każdej podgrupie na podstawie częstotliwości występowania (Rysunek 6). Methanoregula dominuje we wszystkich typach siedlisk niesolankowych. Oprócz Methanoregula, podgrupa neutralna i zimna (grupa 1) wykazuje wysoką liczebność Methanosaeta, Methanobacterium i Methanosarcina. Kwaśna i zimna grupa 2 reprezentowana jest przez Methanobacterium, Methanocella i Methanosarcina, podczas gdy Methanosaeta jest tu nieobecna. Methanocella i Methanosaeta są powszechne w kwaśnej i umiarkowanej grupie 3. W neutralnej i ciepłej grupie 4, Methanolinea i Methanosaeta są ważnymi członkami. Jest to jedyna grupa, w której zidentyfikowano Methanoculleus.

RYSUNEK 6. Częstości występowania linii metanogenicznych w obrębie czterech zdefiniowanych grup zbiorowisk metanogenicznych w glebach i osadach jeziornych. Oś pionowa ułożona jest w porządku alfabetycznym. Długość słupka odpowiada średniej częstotliwości występowania dla każdego z linii w obrębie odpowiedniej grupy. Słupki błędów reprezentują odchylenie standardowe dla danego taksonu w różnych próbkach w danej grupie. Gwiazdki pokazują taksony specjalistyczne z P-value < 0.05. Taksonomia jest pokazana dla poziomu rodzaju. Jeśli przypisanie do poziomu rodzaju nie było możliwe, użyto następnego wyższego poziomu taksonomicznego, który można przypisać. Opis dla czterech grup znajduje się na Rysunku 5.
Dla wszystkich czterech grup, taksony o wysokiej częstości występowania to Methanoregula, Methanobacterium, Methanosarcina, Methanosaeta, Methanomassiliicoccus i Methanocella. Taksony specjalistyczne, które są znacznie liczniej reprezentowane na większości stanowisk w obrębie danej grupy, zostały wykryte na podstawie analizy gatunków wskaźnikowych, jak opisano wcześniej. W sumie, sześć z 31 taksonów wykazało znaczącą wartość wskaźnika (P < 0.05) (oznaczone gwiazdką na Rysunku 6). Grupa 1 (neutralna i zimna) wykazała największą liczbę specjalistów z liniami Methanosaeta, Methanolobus i Methanomethylovorans. Methanobacterium był taksonem wyspecjalizowanym w zimnej i kwaśnej grupie 2, podczas gdy Methanolinea została zidentyfikowana jako specjalista w grupie 4 (neutralnej i ciepłej), ale prawie nie była obserwowana w innych grupach. Ponadto Methanoregula jest w dużej mierze reprezentowana w kwaśnej i umiarkowanej grupie 3.
Dispersal Limitation
Analiza regresji liniowej (R2= 0,05, P < 0,001) wykazała słabą korelację między odległością geograficzną a strukturą zbiorowiska metanogenicznego w globalnym zbiorze danych. Jednocześnie, test Mantela wykazał, że zmienne środowiskowe mają wyższą korelację ze strukturą zbiorowiska niż odległości geograficzne (patrz tab. 3). Tendencja ta jest również potwierdzona przez częściowy test Mantela kontrolujący efekty autokorelacji. Wykres odległości geograficznej względem podobieństwa Jaccarda nie wykazuje wyraźnego trendu liniowego, ale wzorce, które wynikają głównie z globalnego rozmieszczenia punktów poboru próbek (patrz Rysunek uzupełniający S4).

TABELA 3. Analizy testu Mantela i częściowego testu Mantela dla określenia wpływu zmiennych środowiskowych i odległości geograficznej na rozmieszczenie drobnoustrojów dla globalnego zbioru danych i podpróby 16 próbek europejskich.
W celu dalszej analizy wpływu dyspersji ograniczyliśmy naszą analizę do Europy, z której pobrano najgęstsze i najbardziej równomierne próbki. Testy Mantela i częściowe testy Mantela na tym podzbiorze odtworzyły tendencję, że dane o zbiorowiskach są bardziej skorelowane ze zmiennymi środowiskowymi niż z odległościami geograficznymi (patrz Tabela 3). Częściowy test Mantela kontrolujący zmienne środowiskowe nie wykrył żadnej statystycznie istotnej korelacji między zbiorowiskiem mikroorganizmów a odległością geograficzną. Przestrzenna analiza PCA przeprowadzona na tych 16 stanowiskach europejskich wskazuje na strukturę przestrzenną zbiorowiska metanogenicznego (23,7% całkowitej wariancji zostało wyjaśnione przez tę strukturę), co odpowiada dodatniej autokorelacji przestrzennej stanowisk, na co wskazuje wskaźnik I Morana (I Morana = 0,4018). Tylko pierwsza wartość własna była stabilna i odpowiadała rozdzieleniu próbek pomiędzy Europę Środkową i kraje bałtyckie (Rysunek uzupełniający S5). Mały zestaw danych komplikuje jednak solidne przypisanie tej obserwowanej struktury przestrzennej do zmiennych geograficznych, środowiskowych lub do obu z nich. Dlatego przeprowadziliśmy analizę skupień na zbiorowiskach metanogennych, jak opisano powyżej, i ujawniliśmy trzy grupy (Rysunek 7A), które rzutowaliśmy na mapę geograficzną (Rysunek 7B). Klasteryzacja nie odtworzyła podziału przestrzennego PCA wzdłuż Morza Bałtyckiego. W związku z tym niektóre miejsca, które są geograficznie bardzo blisko siebie, wykazują odmienne struktury zbiorowisk metanogenicznych i łączą się w różne klastry. Z drugiej strony, niektóre geograficznie bardzo odległe stanowiska wykazują bardzo podobny skład zbiorowiska i łączą się w klastry (Rysunek 7B). Regionalne rozproszenie grup w Europie nie wydaje się odpowiadać strukturze geograficznej ani być przez nią ograniczone.
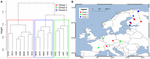
RYSUNEK 7. Hierarchiczna analiza skupień macierzy odległości Jaccarda wśród 16 europejskich próbek gleby i osadów jeziornych. Do analizy wykorzystano metodę grupowania Warda. Miejsca zostały podzielone na trzy grupy pokolorowane różnymi prostokątami w dendrogramie klasteryzacji (A) i rzutowane na mapę Europy (B).
Dyskusja
Identyfikacja i zastosowanie koncepcji biogeografii na zbiorowiskach mikroorganizmów jest głównym przedmiotem zainteresowania ekologii mikroorganizmów. Uważa się, że biogeografia mikroorganizmów jest regulowana przez ewolucyjne i ekologiczne współdziałanie czterech głównych procesów: filtrowania siedlisk, dyspersji, dryfu i mutacji (Hanson i in., 2012). Nawet jeśli wpływ dryfu i mutacji jest poza możliwościami interpretacyjnymi tego opracowania, pokazujemy, że istnieją globalne wzorce zbiorowisk metanogennych w środowiskach naturalnych. Badania te pokazują globalny wzorzec biogeograficzny zbiorowisk metanogennych, który jest bardziej związany z filtrowaniem siedlisk niż z rozproszeniem geograficznym. Zbiorowiska metanogenne z osadów jezior sodowych, na przykład, łączą się blisko z geograficznie odległymi próbkami morskimi (Rysunek 4), a bardzo podobne zbiorowiska metanogenne występują w europejskich osadach glebowych i jeziornych, mimo że znajdują się w dużej odległości od siebie (Rysunek 7). Ogólnie rzecz biorąc, nasze próby ujawnienia potencjalnych ograniczeń dyspersji wykazały słaby wpływ położenia geograficznego na strukturę zbiorowisk metanogennych, co kontrastuje z wyraźnym wpływem warunków środowiskowych. Na podstawie dostępnych danych nie jest możliwe jednoznaczne wykluczenie wpływu przestrzennego na zbiorowiska mikrobiologiczne. Punkty poboru próbek koncentrują się na określonych siedliskach i/lub obszarach, podczas gdy ogólna liczba próbek jest niska. Jeśli istnieje efekt, zakładamy, że efekty przestrzenne występują tylko w skali regionalnej lub lokalnej. Lokalne ograniczenia dyspersji odnotowano dla archaea hipertermofilnych spowodowane barierami geograficznymi (Whitaker et al., 2003), bakterii utleniających amoniak na lokalnych słonych bagnach, ale nie w skali regionalnej i kontynentalnej (Martiny et al., 2011), mikroorganizmów w osadach głębinowych wraz z warunkami środowiskowymi (Schauer et al., 2011).2009), a dla bakterii z dużego zbioru heterogenicznych środowisk śnieżnych głównie ze względu na dostępność węgla allochtonicznego (Lutz i in., 2016). Niewielki wpływ ograniczenia dyspersji na metanogeny w środowiskach naturalnych oznacza, że metanogeny mogą losowo rozprzestrzeniać się w przestrzeni, są z powodzeniem wybierane przez lokalne środowisko, jeśli ich wymagania fizjologiczne są spełnione i mogą tworzyć stabilne zbiorowiska (Martiny i in., 2006; von Mering i in., 2007).
Koncepcja filtrowania siedlisk implikuje, że gatunki o podobnych wymaganiach ekologicznych powinny współwystępować częściej niż jest to oczekiwane przypadkowo (Weiher i Keddy, 2001; Cornwell i in., 2006; Ulrich i in., 2010). Nasze wyniki wskazują, że duże różnice w składzie zbiorowisk metanogenicznych występują pomiędzy ekosystemami morskimi a mokradłami i jeziorami, podczas gdy estuaria plasują się pomiędzy nimi. Wzorce biogeograficzne oparte na zestawie badań genów w próbkach środowiskowych odnotowano również dla bakterii ogólnych (Nemergut i in., 2011), mikrobiomu wycieków metanowych (Ruff i in., 2015), archai utleniających amoniak (Cao i in., 2013), morskich bakterii pelagicznych i bentosowych (Zinger i in., 2011) oraz mikroorganizmów przetwarzających azot (Church i in., 2008). Filtrowanie siedlisk odnotowano w szczególności dla niekulturowych archaea (Auguet i in., 2010), całych zbiorowisk bakteryjnych w zróżnicowanych środowiskach (von Mering i in., 2007; Chaffron i in., 2010) lub w południowoamerykańskich torfowiskach w skali regionalnej (Oloo i in., 2016), jak również specyficzne grupy bakterii, takie jak bakterie utleniające metan (Knief, 2015) i wiążące azot (Nelson i in., 2016).
Nasze wyniki wskazują, że w skali globalnej zasolenie istotnie reguluje skład zbiorowisk metanogennych i determinuje duże różnice między morskimi i lądowymi zespołami metanogennymi. Również metanogeny z osadów jezior sodowych grupują się z metanogenami z osadów morskich (Rysunek 4), podkreślając globalny wpływ zasolenia. Wynik ten jest zgodny z innymi badaniami opartymi na genie 16S rRNA, ujawniającymi, że zasolenie jest głównym czynnikiem kształtującym globalne wzorce ogólnych zbiorowisk bakteryjnych i archeologicznych (Lozupone i Knight, 2007; Auguet i in., 2010; Caporaso i in., 2011; Cao i in., 2013). Niski wpływ separacji geograficznej, ale silny wpływ zasolenia na ogólne zbiorowiska mikroorganizmów został również zaobserwowany we wcześniejszych badaniach (Logares i in., 2013; Yang i in., 2016). Zgodnie z tym, zasolenie w dużej mierze decyduje o tym, które linie mogą przetrwać. W różnych siedliskach aktywność produkcji metanu była ujemnie skorelowana z zasoleniem (Bartlett i in., 1987; Potter i in., 2009; Poffenbarger i in., 2011). Sugeruje się zatem, że hamowanie produkcji metanu przez zasolenie zbiega się w czasie z redukcją wielkości populacji metanogenów (Pattnaik i in., 2000). Wpływ zasolenia na hydrogenotroficzną, acetotroficzną i metylotroficzną metanogenezę zależy od poziomu zasolenia i jest różny dla różnych ścieżek metanogenezy (Liu i in., 2016). Obecnie nie ma jasnego mechanizmu wyjaśniającego wpływ zasolenia na strukturę społeczności, ale kilka hipotez może służyć jako możliwe wyjaśnienie. Fizjologicznie, zasolenie wpływa na zewnętrzną i wewnętrzną osmolarność komórek. Komórki metanogenne nie zasolone wykształciły fizjologiczne przystosowania do przeciwdziałania wewnętrznemu ciśnieniu turgorowemu, podczas gdy komórki przystosowane do zasolenia utraciły tę cechę (Zinder, 1993). Ponadto, rosnące zasolenie może skłaniać metanogeny do syntezy lub pobierania zwiększonej ilości kompatybilnych rozpuszczalników, co wiąże się ze znacznymi kosztami energetycznymi, a tym samym metabolicznymi (McGenity, 2010). Cecha tolerancji na sól przejawia się nawet w optymalnym stężeniu NaCl dla wzrostu czystych kultur metanogennych, ponieważ stwierdziliśmy, że izolaty z osadów morskich i osadów hydrotermalnych mają znacznie wyższe optymalne stężenie NaCl niż izolaty z gleb (Supplementary Figure S2).
W niesolnych ekosystemach lądowych, a konkretnie w glebach i osadach jeziornych, skład zbiorowiska metanogenów jest kontrolowany przez kombinację temperatury i pH. W związku z tym metanogeny tych środowisk można podzielić na cztery grupy (Rysunek 6). W przeciwieństwie do ekosystemów morskich, niesolankowe ekosystemy lądowe wykazują dużą naturalną zmienność zarówno pH, jak i temperatury. Temperatura może wpływać nie tylko na szlak metanogeniczny, ale także na same populacje metanogeniczne (Conrad, 2007; Rooney-Varga i in., 2007). Produkcja metanu może być znacznie zwiększona, jeśli wzrośnie temperatura, co jest konsekwencją wrażliwych na temperaturę etapów fermentacji i acetogenezy (Megonigal i in., 2005; Kotsyurbenko i in., 2007). Dodatkowo, niskie pH może znacząco ograniczyć dostępność octanu poprzez uniemożliwienie jego dysocjacji, a tym samym negatywnie wpłynąć na acetoklastyczną metanogenezę (Fukuzaki i in., 1990; Bridgham i in., 2013). Może to być możliwym powodem, że Methanosaeta była nieobecna w grupie2, podczas gdy Methanosarcina może przełączać się pomiędzy różnymi źródłami i nie uległa znaczącemu wpływowi. Ponadto, pH może regulować efektywność produkcji metanu i szlaki metanogeniczne od torfowisk ombrotroficznych do minerotroficznych, poprzez bezpośrednie hamowanie obu szlaków metanogenezy i pośrednio poprzez wpływ na fermentację (Ye i in., 2012). Zatem zarówno temperatura, jak i pH mogą bezpośrednio lub pośrednio regulować etapy metaboliczne związane z metanogenezą i fermentacją, która dostarcza substratu dla metanogenów.
Methanoregula jest wszechobecna i bardzo liczna we wszystkich czterech grupach siedlisk lądowych (Ryc. 6), ale praktycznie nieobecna w systemie morskim, a zatem może być wskaźnikiem wpływu wód słodkich w sferze morskiej. Jego globalne znaczenie zostało niedawno opisane w innym miejscu (Yang i in., 2017). Pomimo wszechobecnej dystrybucji w glebach i osadach jeziornych, Methanoregula występuje jako linia wskaźnikowa w kwaśnych siedliskach o umiarkowanych temperaturach. Co więcej, (1) Methanolinea wydaje się być szczególnie przystosowana do ciepłych, neutralnych środowisk lądowych, (2) Methanobacterium do zimnych, kwaśnych środowisk, a (3) Methanosaeta do środowisk o neutralnym pH, co jest zgodne z innymi badaniami (Rosenberg i in., 2014) i podkreśla solidność naszego podejścia. Ogólnie rzecz biorąc, warunki geochemiczne otaczające zbiorowiska metanogeniczne będą prowadzić do różnicowania nisz. Ponieważ sortowanie nisz ma tendencję do pozostawiania adaptacyjnych specjalistów (Langenheder i Székely, 2011), postępująca długoterminowa selekcja środowiskowa wygenerowała różnorodne nisze, które zostały wypełnione przez szereg endemicznych specjalistów siedliskowych, które mogą być mniej reprezentowane lub nieobecne w innych odmiennych warunkach środowiskowych. Zbiorowisko jest również kształtowane przez czynniki biotyczne, takie jak interakcje ekologiczne, dynamika, konkurencja i symbioza. Pomimo tych czynników biologicznych, von Mering et al. (2007) stwierdzili, że preferencje siedliskowe są często niezwykle stabilne w czasie, a wyróżniający się skład taksonomiczny zbiorowisk środowiskowych może być ważnym wskaźnikiem ich ekologii i funkcji.
Zgodnie z preferencjami siedliskowymi archaii metanogennych, wydaje się, że blisko spokrewnione szczepy metanogenne były często izolowane z porównywalnych środowisk. Na przykład Methanoregulaceae wydaje się być dość zróżnicowana w naturalnych środowiskach (Yang i in., 2017), a neutralne szczepy mogą opierać się hodowli do tej pory. Obecnie opisane szczepy Methanoregula są zarówno uzyskane z lekko acidofilnych środowisk, podczas gdy dwaj przedstawiciele Methanolinea pochodzą ze stosunkowo ciepłych siedlisk, takich jak odpowiednio osady fermentacyjne i gleba z pól ryżowych (Rosenberg i in., 2014). Chociaż szczepy Methanobacterium zostały wyizolowane z różnych środowisk, około połowa istniejących izolatów tego rodzaju wykazuje optima pH nieco poniżej 7. Oznacza to, że linie wskaźnikowe, które zostały zidentyfikowane na podstawie sekwencji środowiskowych, mogą odzwierciedlać zróżnicowanie fizjologii i źródeł istniejących odmian metanogennych. Na przykład, zasolenie siedliska, jako ogólna właściwość siedliska, może stopniowo narażać organizmy na silną selekcję środowiskową i filtrować montaż nowego zestawu gatunków, które są najlepiej dostosowane do zasolenia otoczenia (Logares i in., 2013).
Ochrona różnorodności biologicznej i zarządzanie nią jest podstawowym wyzwaniem naszego obecnego społeczeństwa. Tutaj pokazujemy, że metanogenne archaea środowisk naturalnych są najbardziej zróżnicowane w osadach estuariów. Estuaria są strefami przejściowymi pomiędzy ekosystemami morskimi i lądowymi. Umożliwia to występowanie dwóch głównych procesów, które mogą przyczyniać się do bogactwa gatunkowego. Po pierwsze, drobnoustroje morskie i lądowe mieszają się w ujściach rzek i w efekcie uzyskują wysoką różnorodność (McLusky i Elliott, 2004). Na przykład zaobserwowano dużą różnorodność bakterii estuariowych, archai, grzybów, a nawet specyficznych bakterii pełniących unikalne funkcje (Cunliffe i in., 2008; Mosier i Francis, 2008; Crump i in., 2012). Innym aspektem jest wysoki poziom składników odżywczych spowodowany lądowymi i pływowymi dopływami, którymi żywią się organizmy estuarium (McLusky i Elliott, 2004; Statham, 2012). W tym kontekście środowiska estuariowe mają istotne znaczenie w odzyskiwaniu nowości gatunkowych dla metanogenów. Jak dotąd, wpływ różnorodności gatunkowej na procesy zachodzące w ekosystemie był przedmiotem wielu badań. Związek pomiędzy różnorodnością biologiczną a funkcją ekosystemu jest wciąż przedmiotem debaty i pozostaje nieuchwytny w przypadku zbiorowisk mikrobiologicznych (Loreau i in., 2001; Tilman i in., 2014). Mimo, że gleby i osady jeziorne są głównymi źródłami metanu, a także siedliskami o dużej różnorodności metanogennej, proponujemy, aby bogactwo gatunkowe nie było odpowiednim wskaźnikiem potencjału produkcji metanu i emisji metanu przez ekosystemy; wydaje się raczej odzwierciedlać heterogeniczność i historię środowiska. Uszeregowanie środowisk według ich bogactwa gatunkowego niekoniecznie oznacza potencjał emisji metanu, który jest najwyższy z gleb i jezior, a porównywalnie mniejszy z estuariów (Rysunek 3 i Supplementary Figure S1).
Wreszcie, brak informacji środowiskowych w publicznych bazach danych mógł utrudnić pełną interpretację czynników środowiskowych obserwowanych tutaj. Mimo że w literaturze i publicznych bazach danych można znaleźć coraz więcej danych sekwencjonowania metanogenów, związane z nimi zmienne abiotyczne są często niespójne i skąpe. Ograniczona ilość spójnych informacji dla zmiennych środowiskowych ogranicza zastosowanie wielowariantowych analiz statystycznych. Przypomnienie, że czynniki abiotyczne w tym badaniu mogą wyjaśnić jedynie ograniczoną część wariancji zbiorowiska sugeruje, że brakuje innych zmiennych objaśniających. Szczególnie ważne mogą być stężenia i dostępność substratów metanogennych, takich jak octan, wodór i metyloaminy. Niemniej jednak, parametry abiotyczne mogą nigdy nie wystarczyć do pełnego wyjaśnienia struktury zespołów metanogenicznych, ponieważ siedliska mają różną historię i mogą być badane tylko lokalnie. Ponadto, wśród dostępnych danych mcrA słabo reprezentowane są rozległe obszary rosyjskiej i kanadyjskiej Subarktyki i Arktyki. Lepsze pokrycie geograficzne i równomierne rozmieszczenie zestawu danych genów mcrA poprawiłoby ocenę zbiorowisk metanogenicznych w skali globalnej.
Wkład autorów
SL i XW zaprojektowali badania. XW i SY zebrali i przeanalizowali dane. XW, SY i FH przeprowadzili analizę statystyczną. XW, MW i SY dokonali korekty filogenetycznej. XW, SY, FH, MW, DW i SL zinterpretowali wyniki i napisali pracę. All authors contributed to the discussions and reviewed the manuscript.
Conflict of Interest Statement
The authors declare that the research was conducted in the absence of any commercial or financial relationships that could be construed as a potential conflict of interest.
Acknowledgments
The financial support to XW (Grant No. 201408620031 to XW) provided by the China Scholarship Council (CSC) is gratefully acknowledged. Badanie to było wspierane przez Helmholtz Gemeinschaft (HGF) poprzez finansowanie Helmholtz Young Investigators Group of SL (VH-NG-919).
Materiały uzupełniające
Materiały uzupełniające do tego artykułu można znaleźć online pod adresem: https://www.frontiersin.org/article/10.3389/fmicb.2017.01339/full#supplementary-material
Footnotes
- ^ http://metanogen.biotech.uni.wroc.pl/
- ^ http://www.ncbi.nlm.nih.gov/
- ^ http://metanogen.biotech.uni.wroc.pl/
- ^ https://www.ngdc.noaa.gov/mgg/shorelines/gshhs.html
- ^ http://qgis.osgeo.org
Auguet, J. C., Barberan, A., and Casamayor, E. O. (2010). Global ecological patterns in uncultured Archaea. ISME J. 4, 182-190. doi: 10.1038/ismej.2009.109
PubMed Abstract | CrossRef Full Text | Google Scholar
Bange, H. W., Bartell, U. H., Rapsomanikis, S., and Andreae, M. O. (1994). Methane in the Baltic and North Seas and a reassessment of the marine emissions of methane. Glob. Biogeochem. Cycles 8, 465-480. doi: 10.1029/94GB02181
CrossRef Full Text | Google Scholar
Barreto, D. P., Conrad, R., Klose, M., Claus, P., and Enrich-Prast, A. (2014). Distance-decay and taxa-area relationships for bacteria, archaea and methanogenic archaea in a tropical lake sediment. PLoS ONE 9:e110128. doi: 10.1371/journal.pone.0110128
PubMed Abstract | CrossRef Full Text | Google Scholar
Bartlett, K. B., Bartlett, D. S., Harriss, R. C., and Sebacher, D. I. (1987). Methane emissions along a salt marsh salinity gradient. Biogeochemistry 4, 183-202. doi: 10.1007/BF02187365
CrossRef Full Text | Google Scholar
Borrel, G., O’Toole, P. W., Harris, H. M. B., Peyret, P., Brugere, J. F., and Gribaldo, S. (2013). Phylogenomic data support a seventh order of methylotrophic methanogens and provide insights into the evolution of methanogenesis. Genome Biol. Evol. 5, 1769-1780. doi: 10.1093/Gbe/Evt128
PubMed Abstract | CrossRef Full Text | Google Scholar
Bousquet, P., Ciais, P., Miller, J., Dlugokencky, E., Hauglustaine, D., Prigent, C., et al. (2006). Contribution of anthropogenic and natural sources to atmospheric methane variability. Nature 443, 439-443. doi: 10.1038/nature05132
PubMed Abstract | CrossRef Full Text | Google Scholar
Bridgham, S. D., Cadillo-Quiroz, H., Keller, J. K., and Zhuang, Q. (2013). Methane emissions from wetlands: biogeochemical, microbial, and modeling perspectives from local to global scales. Glob. Chang. Biol. 19, 1325-1346. doi: 10.1111/gcb.12131
PubMed Abstract | CrossRef Full Text | Google Scholar
Cao, H., Auguet, J.-C., and Gu, J.-D. (2013). Global ecological pattern of ammonia-oxidizing archaea. PLoS ONE 8:e52853. doi: 10.1371/journal.pone.0052853
PubMed Abstract | CrossRef Full Text | Google Scholar
Caporaso, J. G., Lauber, C. L., Walters, W. A., Berg-Lyons, D., Lozupone, C. A., Turnbaugh, P. J., et al. (2011). Global patterns of 16S rRNA diversity at a depth of millions of sequences per sample. Proc. Natl. Acad. Sci. U.S.A. 108, 4516-4522. doi: 10.1073/pnas.1000080107
PubMed Abstract | CrossRef Full Text | Google Scholar
Chaffron, S., Rehrauer, H., Pernthaler, J., and von Mering, C. (2010). A global network of coexisting microbes from environmental and whole-genome sequence data. Genome Res. 20, 947-959. doi: 10.1101/gr.104521.109
PubMed Abstract | CrossRef Full Text | Google Scholar
Church, M. J., Bjorkman, K. M., Karl, D. M., Saito, M. A., and Zehr, J. P. (2008). Regional distributions of nitrogen-fixing bacteria in the Pacific Ocean. Limnol. Oceanogr. 53, 63-77. doi: 10.4319/lo.2008.53.1.0063
CrossRef Full Text | Google Scholar
Conrad, R. (2007). „Microbial ecology of methanogens and methanotrophs,” in Advances in Agronomy, ed. L. S. Donald (Cambridge, MA: Academic Press), 1-63.
Google Scholar
Cornwell, W. K., Schwilk, D. W., and Ackerly, D. D. (2006). A trait-based test for habitat filtering: convex hull volume. Ecology 87, 1465-1471. doi: 10.1890/0012-9658(2006)872.0.CO;2
PubMed Abstract | CrossRef Full Text | Google Scholar
Crump, B. C., Amaral-Zettler, L. A., and Kling, G. W. (2012). Microbial diversity in arctic freshwaters is structured by inoculation of microbes from soils. ISME J. 6, 1629-1639. doi: 10.1038/ismej.2012.9
PubMed Abstract | CrossRef Full Text | Google Scholar
Cui, M., Ma, A., Qi, H., Zhuang, X., Zhuang, G., and Zhao, G. (2015). Warmer temperature accelerates methane emissions from the Zoige wetland on the Tibetan Plateau without changing methanogenic community composition. Sci. Rep. 5:11616. doi: 10.1038/srep11616
PubMed Abstract | CrossRef Full Text | Google Scholar
Cunliffe, M., Schafer, H., Harrison, E., Cleave, S., Upstill-Goddard, R., and Murrell, J. C. (2008). Phylogenetic and functional gene analysis of the bacterial and archaeal communities associated with the surface microlayer of an estuary. ISME J. 2, 776-789. doi: 10.1038/ismej.2008.28
PubMed Abstract | CrossRef Full Text | Google Scholar
Dray, S., and Dufour, A. B. (2007). The ade4 package: implementing the duality diagram for ecologists. J. Stat. Softw. 22, 1-20. doi: 10.18637/jss.v022.i04
CrossRef Full Text | Google Scholar
Dufrene, M., and Legendre, P. (1997). Zespoły gatunków i gatunki wskaźnikowe: potrzeba elastycznego, asymetrycznego podejścia. Ecol. Monogr. 67, 345-366. doi: 10.2307/2963459
CrossRef Full Text | Google Scholar
Edgar, R. C., Haas, B. J., Clemente, J. C., Quince, C., and Knight, R. (2011). UCHIME poprawia czułość i szybkość wykrywania chimer. Bioinformatics 27, 2194-2200. doi: 10.1093/bioinformatics/btr381
PubMed Abstract | CrossRef Full Text | Google Scholar
Evans, P. N., Parks, D. H., Chadwick, G. L., Robbins, S. J., Orphan, V. J., Golding, S. D., et al. (2015). Methane metabolism in the archaeal phylum Bathyarchaeota revealed by genome-centric metagenomics. Science 350, 434-438. doi: 10.1126/science.aac7745
PubMed Abstract | CrossRef Full Text | Google Scholar
Fish, J. A., Chai, B., Wang, Q., Sun, Y., Brown, C. T., Tiedje, J. M., et al. (2013). FunGene: the functional gene pipeline and repository. Front. Microbiol. 4:291. doi: 10.3389/fmicb.2013.00291
PubMed Abstract | CrossRef Full Text | Google Scholar
Frank-Fahle, B. A., Yergeau, E., Greer, C. W., Lantuit, H., and Wagner, D. (2014). Microbial functional potential and community composition in permafrost-affected soils of the NW Canadian Arctic. PLoS ONE 9:e84761. doi: 10.1371/journal.pone.0084761.g001
PubMed Abstract | CrossRef Full Text | Google Scholar
Fukuzaki, S., Nishio, N., and Nagai, S. (1990). Kinetics of the methanogenic fermentation of acetate. Appl. Environ. Microbiol. 56, 3158-3163.
Google Scholar
Hanson, C. A., Fuhrman, J. A., Horner-Devine, M. C., and Martiny, J. B. (2012). Beyond biogeographic patterns: processes shaping the microbial landscape. Nat. Rev. Microbiol. 10, 497-506. doi: 10.1038/Nrmicro2795
PubMed Abstract | CrossRef Full Text | Google Scholar
Holm, G. O., Perez, B. C., McWhorter, D. E., Krauss, K. W., Johnson, D. J., Raynie, R. C., et al. (2016). Ecosystem level methane fluxes from tidal freshwater and brackish marshes of the Mississippi River Delta: implications for coastal wetland carbon projects. Wetlands 36, 401-413. doi: 10.1007/s13157-016-0746-7
CrossRef Full Text | Google Scholar
Hunger, S., Schmidt, O., Hilgarth, M., Horn, M. A., Kolb, S., Conrad, R., et al. (2011). Competing formate- and carbon dioxide-utilizing prokaryotes in anoxic methane-emitting fen soil. Appl. Environ. Microbiol. 77, 3773-3785. doi: 10.1128/AEM.00282-11
PubMed Abstract | CrossRef Full Text | Google Scholar
IPCC (2007). „Climate Change 2007: the Physical Science Basis,” in Proceedings of the Contribution of Working Group I to the Fourth Assessment Report of the Intergovernmental Panel on Climate Change, eds D. Qin, S. Solomon, M. Manning, Z. Chen, M. Marquis, K. B. Averyt, et al. (Cambridge: Cambridge University Press).
Google Scholar
Jabłoński, S., Rodowicz, P., and Łukaszewicz, M. (2015). Methanogenic archaea database containing physiological and biochemical characteristics. Int. J. Syst. Evol. Microbiol. 65, 1360-1368. doi: 10.1099/ijs.0.000065
PubMed Abstract | CrossRef Full Text | Google Scholar
Kirschke, S., Bousquet, P., Ciais, P., Saunois, M., Canadell, J. G., Dlugokencky, E. J., et al. (2013). Three decades of global methane sources and sinks. Nat. Geosci. 6, 813-823. doi: 10.1038/Ngeo1955
CrossRef Full Text | Google Scholar
Knief, C. (2015). Diversity and habitat preferences of cultivated and uncultivated aerobic methanotrophic bacteria evaluated based on pmoA as molecular marker. Front. Microbiol. 6:1346. doi: 10.3389/fmicb.2015.01346
PubMed Abstract | CrossRef Full Text | Google Scholar
Kotsyurbenko, O., Friedrich, M., Simankova, M., Nozhevnikova, A., Golyshin, P., Timmis, K., et al. (2007). Shift from acetoclastic to H2-dependent methanogenesis in a West Siberian peat bag at low pH values and isolation of an acidophilic Methanobacterium strain. Appl. Environ. Microbiol. 73, 2344-2348. doi: 10.1128/AEM.02413-06
PubMed Abstract | CrossRef Full Text | Google Scholar
Langenheder, S., and Székely, A. J. (2011). Species sorting and neutral processes are both important during the initial assembly of bacterial communities. ISME J. 5, 1086-1094. doi: 10.1038/ismej.2010.207
PubMed Abstract | CrossRef Full Text | Google Scholar
Liebner, S., Ganzert, L., Kiss, A., Yang, S., Wagner, D., and Svenning, M. M. (2015). Shifts in methanogenic community composition and methane fluxes along the degradation of discontinuous permafrost. Front. Microbiol. 6:356. doi: 10.3389/fmicb.2015.00356
PubMed Abstract | CrossRef Full Text | Google Scholar
Liu, Y. Q., Priscu, J. C., Xiong, J. B., Conrad, R., Vick-Majors, T., Chu, H. Y., et al. (2016). Salinity drives archaeal distribution patterns in high altitude lake sediments on the Tibetan Plateau. FEMS Microbiol. Ecol. 92:fiw033. doi: 10.1093/femsec/fiw033
PubMed Abstract | CrossRef Full Text | Google Scholar
Logares, R., Lindstrom, E. S., Langenheder, S., Logue, J. B., Paterson, H., Laybourn-Parry, J., et al. (2013). Biogeografia zbiorowisk bakteryjnych narażonych na postępujące, długotrwałe zmiany środowiskowe. ISME J. 7, 937-948. doi: 10.1038/ismej.2012.168
PubMed Abstract | CrossRef Full Text | Google Scholar
Loreau, M., Naeem, S., Inchausti, P., Bengtsson, J., Grime, J. P., Hector, A., et al. (2001). Różnorodność biologiczna i funkcjonowanie ekosystemu: obecna wiedza i przyszłe wyzwania. Science 294, 804-808. doi: 10.1126/science.1064088
PubMed Abstract | CrossRef Full Text | Google Scholar
Lozupone, C. A., and Knight, R. (2007). Global patterns in bacterial diversity. Proc. Natl. Acad. Sci. U.S.A. 104, 11436-11440. doi: 10.1073/pnas.0611525104
PubMed Abstract | CrossRef Full Text | Google Scholar
Luton, P. E., Wayne, J. M., Sharp, R. J., and Riley, P. W. (2002). The mcrA gene as an alternative to 16S rRNA in the phylogenetic analysis of methanogen populations in landfill. Microbiology 148, 3521-3530. doi: 10.1099/00221287-148-11-3521
PubMed Abstract | CrossRef Full Text | Google Scholar
Lutz, S., Anesio, A. M., Raiswell, R., Edwards, A., Newton, R. J., Gill, F., et al. (2016). The biogeography of red snow microbiomes and their role in melting arctic glaciers. Nat. Commun. 7:11968. doi: 10.1038/ncomms11968
PubMed Abstract | CrossRef Full Text | Google Scholar
Martiny, J. B. H., Bohannan, B. J. M., Brown, J. H., Colwell, R. K., Fuhrman, J. A., Green, J. L., et al. (2006). Microbial biogeography: putting microorganisms on the map. Nat. Rev. Microbiol. 4, 102-112. doi: 10.1038/Nrmicro1341
PubMed Abstract | CrossRef Full Text | Google Scholar
Martiny, J. B. H., Eisen, J. A., Penn, K., Allison, S. D., and Horner-Devine, M. C. (2011). Drivers of bacterial beta-diversity depend on spatial scale. Proc. Natl. Acad. Sci. U.S.A. 108, 7850-7854. doi: 10.1073/pnas.1016308108
PubMed Abstract | CrossRef Full Text | Google Scholar
McArdle, B. H., and Anderson, M. J. (2001). Fitting multivariate models to community data: a comment on distance-based redundancy analysis. Ecology 82, 290-297. doi: 10.1890/0012-9658(2001)0822.0.CO;2
CrossRef Full Text | Google Scholar
McCalley, C. K., Woodcroft, B. J., Hodgkins, S. B., Wehr, R. A., Kim, E.-H., Mondav, R., et al. (2014). Methane dynamics regulated by microbial community response to permafrost thaw. Nature 514, 478-481. doi: 10.1038/nature13798
PubMed Abstract | CrossRef Full Text | Google Scholar
McGenity, T. J. (2010). „Methanogens and Methanogenesis in Hypersaline Environments,” in Handbook of Hydrocarbon and Lipid Microbiology, ed. K. N. Timmis (Berlin: Springer), 665-680. doi: 10.1007/978-3-540-77587-4_53
CrossRef Full Text | Google Scholar
McLusky, D. S., and Elliott, M. (2004). The Estuarine Ecosystem: Ecology, Threats and Management. New York, NY: Oxford University Press. doi: 10.1093/acprof:oso/9780198525080.001.0001
CrossRef Full Text | Google Scholar
Megonigal, J. P., Mines, M. E., and Visscher, P. T. (2005). „Anaerobic metabolism: linkages to trace gases and aerobic processes,” in Biogeochemistry, ed. W. H. Schlesinger (Oxford: Elsevier), 350-362.
Google Scholar
Milferstedt, K., Youngblut, N. D., and Whitaker, R. J. (2010). Spatial structure and persistence of methanogen populations in humic bog lakes. ISME J. 4, 764-776. doi: 10.1038/Ismej.2010.7
PubMed Abstract | CrossRef Full Text | Google Scholar
Mosier, A. C., and Francis, C. A. (2008). Relative abundance and diversity of ammonia-oxidizing archaea and bacteria in the San Francisco Bay estuary. Environ. Microbiol. 10, 3002-3016. doi: 10.1111/j.1462-2920.2008.01764.x
PubMed Abstract | CrossRef Full Text | Google Scholar
Nazaries, L., Murrell, J. C., Millard, P., Baggs, L., and Singh, B. K. (2013). Metan, mikroby i modele: podstawowe zrozumienie cyklu metanowego w glebie dla przyszłych prognoz. Environ. Microbiol. 15, 2395-2417. doi: 10.1111/1462-2920.12149
PubMed Abstract | CrossRef Full Text | Google Scholar
Nelson, M. B., Martiny, A. C., and Martiny, J. B. H. (2016). Global biogeography of microbial nitrogen-cycling traits in soil. Proc. Natl. Acad. Sci. U.S.A. 113, 8033-8040. doi: 10.1073/pnas.1601070113
PubMed Abstract | CrossRef Full Text | Google Scholar
Nemergut, D. R., Costello, E. K., Hamady, M., Lozupone, C., Jiang, L., Schmidt, S. K., et al. (2011). Global patterns in the biogeography of bacterial taxa. Environ. Microbiol. 13, 135-144. doi: 10.1111/j.1462-2920.2010.02315.x
PubMed Abstract | CrossRef Full Text | Google Scholar
Nobu, M. K., Narihiro, T., Kuroda, K., Mei, R., and Liu, W. T. (2016). Chasing the elusive Euryarchaeota class WSA2: genomes reveal a uniquely fastidious methyl-reducing methanogen. ISME J. 10, 2478-2487. doi: 10.1038/ismej.2016.33
PubMed Abstract | CrossRef Full Text | Google Scholar
Oksanen, J., Blanchet, F. G., Kindt, R., Legendre, P., Minchin, P. R., O’Hara, R. B., et al. (2015). vegan: Community Ecology Package. R Package Version 2.2.0. Dostępne na: https://CRAN.R-project.org/package=vegan
Google Scholar
Oloo, F., Valverde, A., Quiroga, M. V., Vikram, S., Cowan, D., and Mataloni, G. (2016). Heterogeniczność siedliska i łączność kształtują społeczności mikrobialne na torfowiskach Ameryki Południowej. Sci. Rep. 6:25712. doi: 10.1038/srep25712
PubMed Abstract | CrossRef Full Text | Google Scholar
Pattnaik, P., Mishra, S. R., Bharati, K., Mohanty, S. R., Sethunathan, N., and Adhya, T. K. (2000). Influence of salinity on methanogenesis and associated microflora in tropical rice soils. Microbiol. Res. 155, 215-220. doi: 10.1016/S0944-5013(00)80035-X
PubMed Abstract | CrossRef Full Text | Google Scholar
Poffenbarger, H. J., Needelman, B. A., and Megonigal, J. P. (2011). Salinity influence on methane emissions from tidal marshes. Wetlands 31, 831-842. doi: 10.1007/s13157-011-0197-0
PubMed Abstract | CrossRef Full Text | Google Scholar
Potter, E. G., Bebout, B. M., and Kelley, C. A. (2009). Isotopic composition of methane and inferred methanogenic substrates along a salinity gradient in a hypersaline microbial mat system. Astrobiology 9, 383-390. doi: 10.1089/ast.2008.0260
PubMed Abstract | CrossRef Full Text | Google Scholar
R Core Team (2014). R: A Language and Environment for Statistical Computing (wersja 3.0.3). Wiedeń: R Foundation for Statistical Computing.
Google Scholar
Roberts, D. (2016). labdsv: Ordination and Multivariate Analysis for Ecology. R Package Version 1.8-0. dostępne na: https://CRAN.R-project.org/package=labdsv
Google Scholar
Rooney-Varga, J. N., Giewat, M. W., Duddleston, K. N., Chanton, J. P., and Hines, M. E. (2007). Links between archaeal community structure, vegetation type and methanogenic pathway in Alaskan peatlands. FEMS Microbiol. Ecol. 60, 240-251. doi: 10.1111/j.1574-6941.2007.00278.x
PubMed Abstract | CrossRef Full Text | Google Scholar
Rosenberg, E., DeLong, E. F., Lory, S., Stackebrandt, E., and Thompson, F. (2014). The Prokaryotes: Other Major Lineages of Bacteria and the Archaea. Berlin: Springer.
Google Scholar
Ruff, S. E., Biddle, J. F., Teske, A. P., Knittel, K., Boetius, A., and Ramette, A. (2015). Global dispersion and local diversification of the methane seep microbiome. Proc. Natl. Acad. Sci. U.S.A. 112, 4015-4020. doi: 10.1073/pnas.1421865112
PubMed Abstract | CrossRef Full Text | Google Scholar
Schauer, R., Bienhold, C., Ramette, A., and Harder, J. (2009). Bacterial diversity and biogeography in deep-sea surface sediments of the South Atlantic Ocean. ISME J. 4, 159-170. doi: 10.1038/ismej.2009.106
PubMed Abstract | CrossRef Full Text | Google Scholar
Schloss, P. D., Westcott, S. L., Ryabin, T., Hall, J. R., Hartmann, M., Hollister, E. B., et al. (2009). Introducing mothur: open-source, platform-independent, community-supported software for describing and comparing microbial communities. Appl. Environ. Microbiol. 75, 7537-7541. doi: 10.1128/Aem.01541-09
PubMed Abstract | CrossRef Full Text | Google Scholar
Statham, P. J. (2012). Składniki odżywcze w estuariach – przegląd i potencjalny wpływ zmian klimatycznych. Sci. Total Environ. 434, 213-227. doi: 10.1016/j.scitotenv.2011.09.088
PubMed Abstract | CrossRef Full Text | Google Scholar
Thauer, R. K., Kaster, A.-K., Seedorf, H., Buckel, W., and Hedderich, R. (2008). Methanogenic archaea: ecologically relevant differences in energy conservation. Nat. Rev. Microbiol. 6, 579-591. doi: 10.1038/Nrmicro1931
PubMed Abstract | CrossRef Full Text | Google Scholar
Tilman, D., Isbell, F., and Cowles, J. M. (2014). Różnorodność biologiczna i funkcjonowanie ekosystemów. Annu. Rev. Ecol. Evol. Syst. 45, 471-493. doi: 10.1146/annurev-ecolsys-120213-091917
CrossRef Full Text | Google Scholar
Ulrich, W., Hajdamowicz, I., Zalewski, M., Stańska, M., Ciurzycki, W., and Tykarski, P. (2010). Asortyment gatunkowy czy filtrowanie siedliska: studium przypadku zbiorowisk pająków na wyspach jeziornych. Ecol. Res. 25, 375-381. doi: 10.1007/s11284-009-0661-y
CrossRef Full Text | Google Scholar
Vanwonterghem, I., Evans, P. N., Parks, D. H., Jensen, P. D., Woodcroft, B. J., Hugenholtz, P., et al. (2016). Metanogeneza metylotroficzna odkryta w archeologicznym phylum Verstraetearchaeota. Nat. Microbiol. 1:16170. doi: 10.1038/nmicrobiol.2016.170
PubMed Abstract | CrossRef Full Text | Google Scholar
von Mering, C., Hugenholtz, P., Raes, J., Tringe, S. G., Doerks, T., Jensen, L. J., et al. (2007). Quantitative phylogenetic assessment of microbial communities in diverse environments. Science 315, 1126-1130. doi: 10.1126/science.1133420
PubMed Abstract | CrossRef Full Text | Google Scholar
Wagner, D., and Liebner, S. (2009). „Globalne ocieplenie i dynamika węgla w glebach wiecznej zmarzliny: produkcja metanu i utlenianie”, w Permafrost Soils, red. R. Margesin (Berlin: Springer), 219-236.
Google Scholar
Weiher, E., and Keddy, P. (2001). Ecological Assembly Rules: Perspectives, Advances, Retreats. Cambridge: Cambridge University Press.
Google Scholar
Whitaker, R. J., Grogan, D. W., and Taylor, J. W. (2003). Geographic barriers isolate endemic populations of hyperthermophilic archaea. Science 301, 976-978. doi: 10.1126/science.1086909
PubMed Abstract | CrossRef Full Text | Google Scholar
Wickham, H. (2009). ggplot2: Elegant Graphics for Data Analysis. New York, NY: Springer-Verlag. doi: 10.1007/978-0-387-98141-3
CrossRef Full Text | Google Scholar
Yang, J., Ma, L. A., Jiang, H., Wu, G., and Dong, H. (2016). Salinity shapes microbial diversity and community structure in surface sediments of the Qinghai-Tibetan Lakes. Sci. Rep. 6:25078. doi: 10.1038/srep25078
PubMed Abstract | CrossRef Full Text | Google Scholar
Yang, S., Liebner, S., Alawi, M., Ebenhöh, O., and Wagner, D. (2014). Taxonomic database and cut-off value for processing mcrA gene 454 pyrosequencing data by MOTHUR. J. Microbiol. Methods 103, 3-5. doi: 10.1016/j.mimet.2014.05.006
PubMed Abstract | CrossRef Full Text | Google Scholar
Yang, S., Liebner, S., Winkel, M., Alawi, M., Horn, F., Dörfer, C., et al. (2017). In-depth analysis of core methanogenic communities from high elevation permafrost-affected wetlands. Soil Biol. Biochem. 111, 66-77. doi: 10.1016/j.soilbio.2017.03.007
CrossRef Full Text | Google Scholar
Ye, R. Z., Jin, Q. S., Bohannan, B., Keller, J. K., McAllister, S. A., and Bridgham, S. D. (2012). pH controls over anaerobic carbon mineralization, the efficiency of methane production, and methanogenic pathways in peatlands across an ombrotrophic-minerotrophic gradient. Soil Biol. Biochem. 54, 36-47. doi: 10.1016/j.soilbio.2012.05.015
CrossRef Full Text | Google Scholar
Zinder, S. H. (1993). „Fizjologiczna ekologia metanogenów”, w: Metanogeneza: Ecology, Physiology, Biochemistry & Genetics, ed. J. G. Ferry (Boston, MA: Springer), 128-206.
Google Scholar
Zinger, L., Amaral-Zettler, L. A., Fuhrman, J. A., Horner-Devine, M. C., Huse, S. M., Welch, D. B. M., et al. (2011). Global patterns of bacterial beta-diversity in seafloor and seawater ecosystems. PLoS ONE 6:e24570. doi: 10.1371/journal.pone.0024570
PubMed Abstract | CrossRef Full Text | Google Scholar
.